

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
蛋白质乳酸化修饰(Lactylation)是一种新型的蛋白质翻译后修饰,发生在赖氨酸残基上,广泛存在于细胞核、细胞质及线粒体中的各种蛋白。其中乳酸部分来源于糖酵解、谷氨酰胺分解等代谢途径,并可形成乳酰‑CoA或乳酰‑AMP中间体,最终在赖氨酸乙酰转移酶p300、CBP的介导下为各种蛋白添加乳酸化修饰,带有乳酸化修饰的蛋白也可以被去乙酰酶如HDAC1‑3、SIRT2‑3等介导去除乳酸化[1]。
乳酸化修饰蛋白在表观遗传调控中发挥关键作用[2],能够直接改变染色质构象和基因启动子的活性[3],从而显著影响各种基因的转录调控。蛋白乳酸化修饰的异常将会导致基因表达失衡[4],与肿瘤发生[2]、免疫抑制[5]、代谢紊乱[6]、心血管疾病[7]及神经系统疾病[8]密切相关,因此是一种新兴且极具潜力的疾病干预靶点[9]。
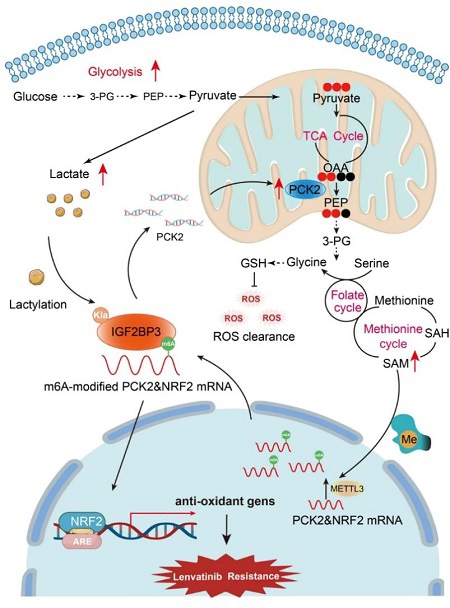
图1.乳酸化修饰增强IGF2BP3对m6A mRNA的结合并上调其表达,最终增强细胞代谢、促进肝癌转移[10]
Aksomics重磅发布乳酸化蛋白质组学分析平台,采用高效的抗体富集策略以及高准确性质谱技术,能够同步解析蛋白质乳酸化修饰的水平与具体位点。平台利用泛乳酸化抗体(Pan‑Kla)对乳酸化肽段进行特异性捕获,再通过LC‑MS/MS实现高精度定量与位点鉴定。既可以检测细胞内所有发生乳酸化的蛋白,也可以针对您感兴趣的特定乳酸化组蛋白进行检测。在高通量条件下提供全面的修饰图谱,为深入研究乳酸化在肿瘤免疫调节、代谢稳态及炎症反应等重要生物学过程中的作用机制提供强有力的支持。
1.检测范围广阔:平台采用泛乳酸化抗体(Pan‑Kla),对具有乳酸化修饰的所有蛋白都可进行特异性识别和结合,实现对乳酸化修饰的全面检测定量。
2.专业数据分析:标准的搜库流程,专业的统计和生信分析,出版级的图表展示,完整全面的分析报告。
3.与Aksomics提供的其它服务平台联合分析:包括label-free非标定量蛋白质组、ChIP-seq、Cut&Tag-Seq、CoIP-MS蛋白互作检测实验、eCLIP-Seq蛋白-RNA互作检测实验、ChIP-seq、mRNA-Seq等。
乳酸化蛋白质组学检测首先通过蛋白酶解获得肽段,再通过使用泛乳酸化抗体(Pan‑Kla)选择性富集带有乳酸化修饰的肽段。经LC‑MS/MS高通量检测可精确定位带有乳酸化修饰的赖氨酸残基位置,并定量和差异分析蛋白的乳酸化修饰水平。
1.蛋白乳酸化修饰水平定量和修饰位点鉴定
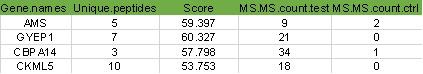
2.蛋白乳酸化修饰水平差异分析
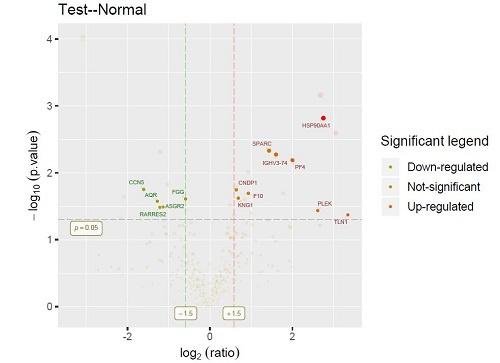
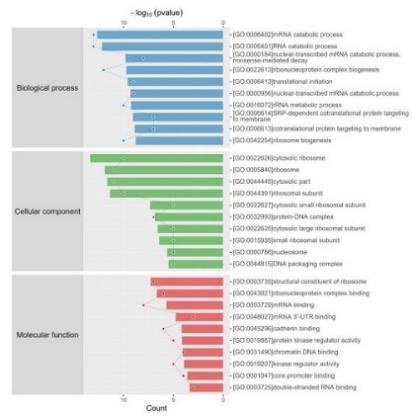
3.功能富集分析和蛋白互作网络分析
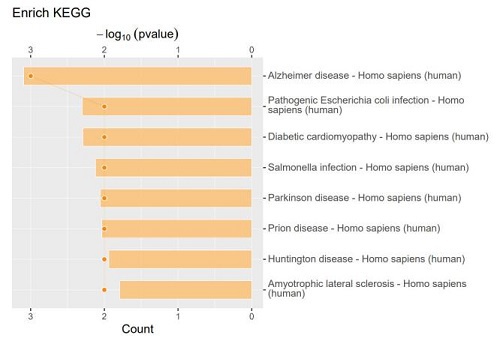
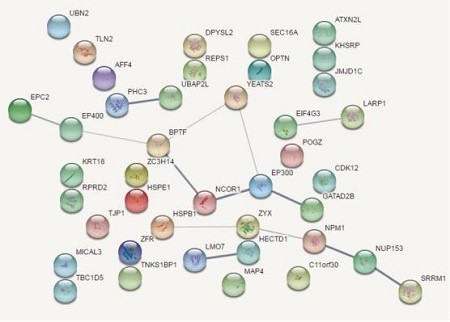
在亚砷酸盐相关的特发性肺纤维化疾病中,组蛋白H3在C端第18位赖氨酸上的乳酸化修饰(H3K18la)有广泛的显著升高,研究发现这些异常的H3K18la乳酸化修饰上调由成纤维细胞中异常活跃的乳酸代谢驱动,通过增强染色质开放性促进下游基因的转录活性,从而加速肺上皮细胞纤维化进程[11]。
本研究通过质谱检测明确了特发性肺纤维化疾病模型中H3K18la组蛋白乳酸化水平的异常上调,随后通过ChIP-seq实验鉴定H3K18la修饰主要定位于YTHDF1基因的启动子区域、并显著提高了YTHDF1基因的转录水平,从而表达出更多YTHDF1蛋白,使其通过m6A reader作用进一步结合带有m6A修饰的mRNA Nrep、增强了Nrep的mRNA稳定性,最终促进纤维化通路的激活。抑制乳酸生成或敲低YTHDF1均可抑制这一作用、减缓细胞纤维化的进程。总之,H3K18la乳酸化修饰的异常上调在亚砷酸盐相关的肺纤维化病变过程中是关键节点和有前景的治疗靶点,具有重要的研究价值和临床意义。
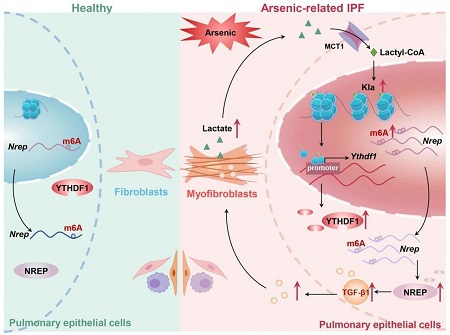
图2. 启动子处的乳酸化修饰上调促进YTHDF1转录,从而稳定m6A-Nrep的表达,促进肺上皮细胞纤维化[11]。
1. Ren, H., Y. Tang, and D. Zhang, The emerging role of protein L-lactylation in metabolic regulation and cell signalling. Nat Metab, 2025. 7(4): p. 647-664.
2. Li, W., et al., Tumor-derived lactate promotes resistance to bevacizumab treatment by facilitating autophagy enhancer protein RUBCNL expression through histone H3 lysine 18 lactylation (H3K18la) in colorectal cancer. Autophagy, 2024. 20(1): p. 114-130.
3. Yu, J., et al., Histone lactylation drives oncogenesis by facilitating m(6)A reader protein YTHDF2 expression in ocular melanoma. Genome Biol, 2021. 22(1): p. 85.
4. Chen, Y., et al., Metabolic regulation of homologous recombination repair by MRE11 lactylation. Cell, 2024. 187(2): p. 294-311.e21.
5. Chen, L., et al., Lactate-Lactylation Hands between Metabolic Reprogramming and Immunosuppression. Int J Mol Sci, 2022. 23(19).
6. Mao, Y., et al., Hypoxia induces mitochondrial protein lactylation to limit oxidative phosphorylation. Cell Res, 2024. 34(1): p. 13-30.
7. Wang, L., et al., Serpina3k lactylation protects from cardiac ischemia reperfusion injury. Nat Commun, 2025. 16(1): p. 1012.
8. Hagihara, H., et al., Protein lactylation induced by neural excitation. Cell Rep, 2021. 37(2): p. 109820.
9. Wang, J., et al., Ubiquitous protein lactylation in health and diseases. Cell Mol Biol Lett, 2024. 29(1): p. 23.
10. Lu, Y., et al., Lactylation-Driven IGF2BP3-Mediated Serine Metabolism Reprogramming and RNA m6A-Modification Promotes Lenvatinib Resistance in HCC. Adv Sci (Weinh), 2024. 11(46): p. e2401399.
11.Wang, P., et al., H3K18 lactylation promotes the progression of arsenite-related idiopathic pulmonary fibrosis via YTHDF1/m6A/NREP. J Hazard Mater, 2024. 461: p. 132582.

