

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
Arraystar Small RNA 芯片利用直接末端标记和和巧妙的探针设计,在同一芯片上同时定性及定量检测多种small RNA,包括miRNA、pre-miRNA、tRFs&tiRNAs、tRNAs和snoRNAs,为Small RNA的调控研究和生物标志物研究提供了重要的表达信息。
芯片特点
• 同时分析了主要的small RNA类别:miRNA、pre-miRNA、tRNA、tRF&tiRNA 和snoRNA。
• 提高Small RNA表达谱的门槛,达到高灵敏度、特异性和准确性。
• 实验流程简单直接,克服了small RNA-seq中由于RNA修饰、RNA二级结构、反转录阻碍、PCR扩增和数据分析不准确造成的偏差。
• 所需的RNA样本量低至100ng,为许多研究创造了机会。
• 可以检测低质量RNA样本,如降解的RNA、血清/血浆等体液RNA、FFPE RNA。
Arraystar Small RNA芯片列表
| 服务名称 | 描述 | 规格 |
|---|---|---|
| Arraystar Human Small RNA 芯片 | miRNAs, pre-miRNAs, tRFs&tiRNAs, tRNAs和snoRNAs | 8 x 15K |
| Arraystar Mouse Small RNA 芯片 | miRNAs, pre-miRNAs, tRFs&tiRNAs, tRNAs 和snoRNAs | 8 x 15K |
| Arraystar Rat Small RNA 芯片 | miRNAs, pre-miRNAs, tRFs&tiRNAs, tRNAs 和snoRNAs | 8 x 15K |
• 同时筛选主要的small RNA类型
Arraystar Small RNA Array 包含miRNA、pre-miRNA、tRFs&tiRNAs、tRNAs和snoRNAs。这些small RNA因其重要的基因调控功能和生物标志物潜力而对生物学和临床科学具有特别的意义。通过small RNA-seq筛选这些small RNAs需要单独的化学方法来构建测序文库和分析,以覆盖具有不同生化特性的small RNA。例如,miRNA-seq和tRNA-seq不能一起建库来分析miRNA和tRNA。现在Arraystar Small RNA Array 可以在同一实验同一芯片上检测这些small RNAs。
• 提高小RNA表达检测的标准,达到高灵敏度、特异性和准确性
Arraystar Small RNA Array 使用高亲和力的探针杂交,即使对低丰度的small RNAs也具有非常高的灵敏度。巧妙的探针设计采用了5’-发夹结构和标准化的序列靶标区域,以特异性的区分只有1~2核苷酸差异的small RNAs。
• 简单直接的步骤确保了比small RNA-seq甚至qPCR有更好的无偏差定量
RNA修饰、RNA二级结构和复杂的RNA-seq建库过程使得Small RNA-seq长期存在挑战。在Arraystar Small RNA芯片中,Small RNAs在其3’-OH端直接与Cy3染料连接,完全避免RNA预处理去除内部RNA修饰,由于RNA修饰和RNA二级结构导致的逆转录失败,以及有偏倚的PCR扩增步骤的影响。所有这些都有助于保持Small RNA水平的保真度,并实现比RNA-seq甚至qPCR更好的无偏的高定量精度。
• RNA样本量要求较低
Arraystar Small RNA Microarray 只需100ng的总RNA,比Small RNA-seq所需样本量要低。芯片由于直接进行RNA末端标记,没有RNA预处理导致的RNA损失,显著减少RNA总量需求,特别是对高度修饰的Small RNA(如tRNA和tRF&tiRNA )。Arraystar Small RNA Microarray对样本量需求低,为样本量少的研究提供了机会。
• 质量较低的RNA样本也能检测
Arraystar small RNA芯片的RNA直接末端标记对底物RNA序列中的核苷酸损伤相对不敏感,因为它不依赖于逆转录cDNA复制。这与Small RNA-seq形成鲜明对比,Small seq容易受到Small RNA序列的任何核苷酸损伤的影响。此外,虽然芯片探针不受不相关序列存在的影响,但降解RNA样本中丰富的rRNA片段可以污染相似大小的Small RNA,抑制Small RNA-seq中的Small RNA检出。
由于这些原因,Small RNA芯片对于长期保存或化学处理的样品、降解的样品特别有利。例如,血清/血浆等体液RNA/FFPE RNA。
小RNA定量检测的挑战和解决方案
Small RNA-seq 已被用于分析 microRNA 和其他小 RNA。 然而,利用small RNA-seq 测序数据来量化的小 RNA 的相对丰度与 qPCR/Northern Blot 结果不一致。 例如,通过small RNA-seq 测得的 miR-143 的水平比 miR-145 高 40 倍,但通过 qPCR 或 Northern Blot 测得它们的表达水平相当 [1-3]。 small RNA-seq 的不准确性主要是由于 small RNA-seq 文库制备和数据分析过程的偏差对结果造成的误导[4, 5],在某些情况下,与真实丰度的偏差可高达 10,000 倍[6-11]。
SmallRNA定量检测的挑战
RNA修饰的干扰
在测序文库构建和小RNA定量过程中,小RNA内部的修饰会严重干扰cDNA合成。 小RNA修饰(例如 m1A、m3C 和 m1G)会阻碍反转录并中止cDNA复制,导致序列偏向于启动端,最终导致构建无偏倚全长序列文库失败。 例如,由于tRF&tiRNA TΨC 环区域中存在m1A修饰的阻碍,small RNA-seq通常识别 18nt 3'tRF&tiRNA ,但遗漏了由Northern Blot检测到的更主要的22nt tRF&tiRNA 异构体。
文库扩增的偏差
为了产生足够的 cDNA 量进入测序仪,small RNA-seq需要对文库进行PCR扩增 [12]。 然而,RNA G/C 含量、序列组成、二级结构、RNA 长度、启动以及反应条件会导致 PCR 产物的偏差和扭曲。 也就是说,当转录组中的不同模板在PCR反应中一起扩增时,模板的偏好或阻滞扩增总是会导致 RNA 无法呈现真实的丰度 [13]。 因此,需要多轮 PCR 扩增的small RNA-seq定量从来都不是绝对定量,需要使用正交方法进行验证。
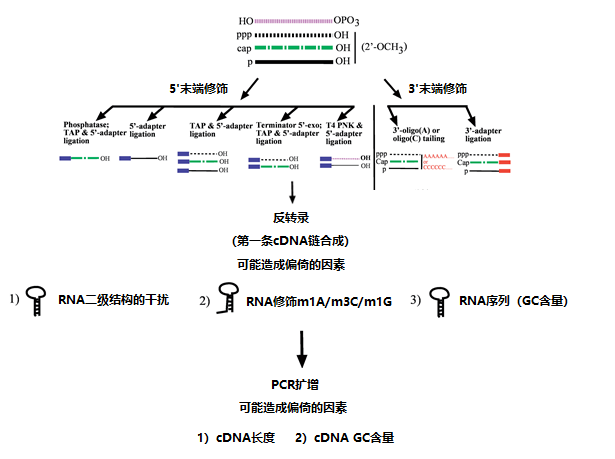
图 1. cDNA构建所涉及步骤的图示,包括潜在的偏差来源。 (A) 具有不同5’端和3’端修饰的非蛋白质编码RNA的起始池,由不同的线型示意性表示。 (B) 左图描述了在RNA 5’端连接之前的不同酶预处理,以富集不同的RNA亚型。在各自的5’端预处理后可用于接头连接的RNA类别在每个预处理下示意性表示。右图描述了可用于3’端(-oligo(A)或-oligo(C)尾)和接头连接的RNA类别的亚型。 (C)与RNA 5’(左)和3’(右)末端修饰以及cDNA构建的后续步骤相关的可能偏差。
采用TPM衡量small RNA表达水平的潜在问题
TPM(Reads Per Million reads)值通常是small RNA-seq用于表示小 RNA 转录本的相对丰度的指标,简单地用测到的每百万小RNA片段做归一化。 然而,在这 100 万次读取中,一个小RNA的读取计数的变化将改变所有其他RNA的TPM值,即使它们实际的绝对表达水平没有改变。
所需样品数量
对于tRNA和tRF&tiRNA 测序,在文库构建之前,目标RNA的分离和预处理需要 5-100ug[14] 的总RNA,这排除了样本量有限的研究项目。
Small RNA筛选的解决方案
为了克服这些挑战,Arraystar设计了Arraystar Small RNA Array 作为一种实用且有效的解决方案,以高灵敏度和准确度分析全谱small RNA,但需要的总RNA量要少得多。 Arraystar small RNA Array 结合直接末端标记和巧妙的探针设计微阵列技术,在一张芯片上同时检测和量化包括miRNA、pre-miRNA、tRF&tiRNA 、tRNA和snoRNA在内的小RNA(图 1),为研究小RNA的调控功能和生物标志物潜力提供重要的表达信息。
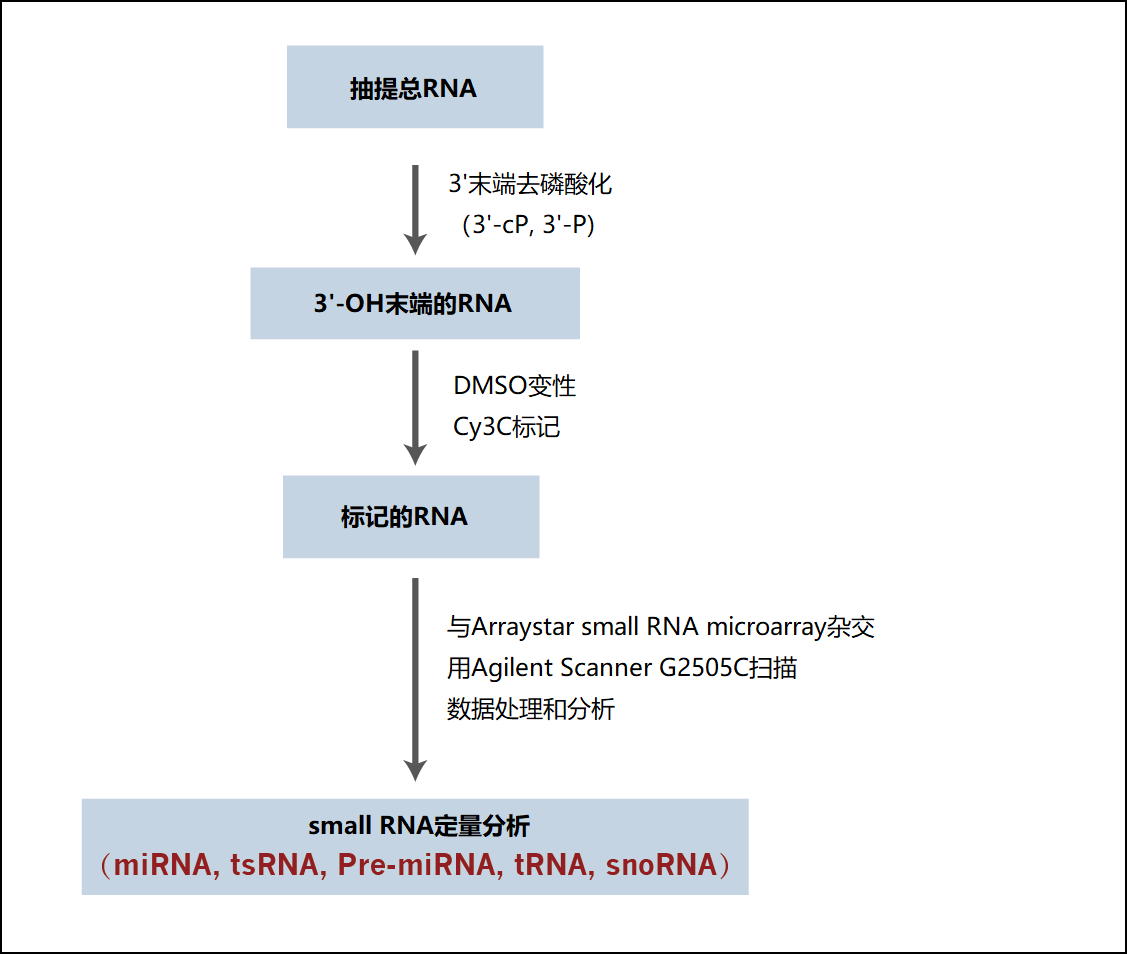
图 1. Arraystar Small RNA芯片的实验流程。总RNA用T4多核苷酸激酶 (T4 PNK) 去磷酸化,去除小RNA 3'端的磷酸(P)或环磷酸(cP)基团,形成3-OH末端。 然后3-OH末端的小RNA用DMSO打开二级结构并酶促连接Cy3标记。标记的RNA杂交到 Arraystar small RNA 芯片上,用于分析小RNA的表达。
参考文献
[1] Kent OA. et al (2010) “Repression of the miR-143/145 cluster by oncogenic Ras initiates a tumor-promoting feed-forward pathway” Genes Dev 24(24):2754-9 [PMID:21159816]
[2] Chivukula RR. et al (2014) “An essential mesenchymal function for miR-143/145 in intestinal epithelial regeneration” Cell 157(5):1104-16 [PMID:24855947]
[3] Akao Y. et al (2007) “Downregulation of microRNAs-143 and -145 in B-cell malignancies” Cancer Sci 98(12):1914-20 [PMID:17892514]
[4] Raabe, C. A., et al. (2014) "Biases in small RNA deep sequencing data" Nucleic Acids Res 42(3):1414-26 [PMID: 24198247]
[5] Witwer, K. W. and Halushka, M. K. (2016) "Toward the promise of microRNAs - Enhancing reproducibility and rigor in microRNA research" RNA Biol 13(11):1103-1116 [PMID: 27645402]
[6] McCormick, K. P., et al. (2011) "Experimental design, preprocessing, normalization and differential expression analysis of small RNA sequencing experiments" Silence 2(1):2 [PMID: 21356093]
[7] Linsen, S. E., et al. (2009) "Limitations and possibilities of small RNA digital gene expression profiling" Nat Methods 6(7):474-6 [PMID: 19564845]
[8] Sorefan, K., et al. (2012) "Reducing ligation bias of small RNAs in libraries for next generation sequencing" Silence 3(1):4 [PMID: 22647250]
[9] Hafner, M., et al. (2011) "RNA-ligase-dependent biases in miRNA representation in deep-sequenced small RNA cDNA libraries" RNA 17(9):1697-712 [PMID: 21775473]
[10] Tian, G., et al. (2010) "Sequencing bias: comparison of different protocols of microRNA library construction" BMC Biotechnol 10:64 [PMID: 20815927]
[11] Fuchs RT. et al (2015) "Bias in ligation-based small RNA sequencing library construction is determined by adaptor and RNA structure" PLoS One 10(5):e0126049 [PMID:25942392]
[12] Kapranov P. et al (2012) "Profiling of short RNAs using Helicos single-molecule sequencing" Methods Mol Biol 822:219-32 [PMID:22144202]
[13] Dabney J. and Meyer M. (2012) "Length and GC-biases during sequencing library amplification: a comparison of various polymerase-buffer systems with ancient and modern DNA sequencing libraries" Biotechniques 52(2):87-94 [PMID:22313406]
[14] Junchao S., et al. (2021) "PANDORA-seq expands the repertoire of regulatory small RNAs by overcoming RNA modifications" Nat Cell Biol 23(6):676 [PMID: 33927371]
Arraystar Small RNA 芯片实验流程
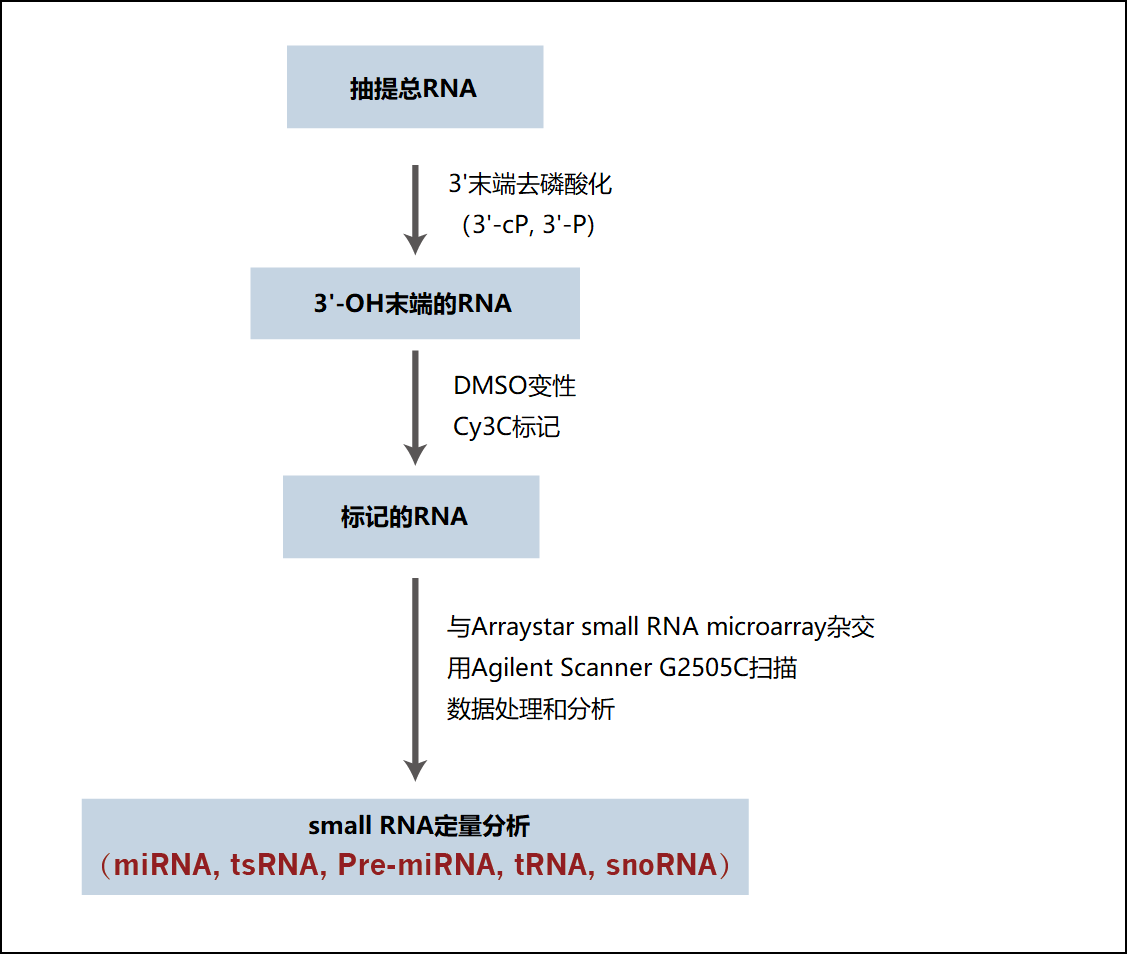
用T4多核苷酸激酶(T4 PNK)处理总RNA,去除3‘端残留的磷酸盐(P)和环磷酸盐(cP)基团,以暴露3-OH。3-OH末端的Small RNA被DMSO变性,并通过连接用Cy3酶标记。将标记的RNA与Arraystar small RNA表达芯片杂交,以分析small RNA表达谱情况。
Arraystar Human small RNA 芯片 V1.0
| 探针总数 | 14,707 |
|---|---|
| 探针设计策略 | 整个探针由5’cap区, small RNA特异性区和3’linker区组成。 |
| 探针结合位点 |
5-p-miRNA 和 5'tRF&tiRNA : small RNA的3’区域 3-p-miRNA 和 3'tRF&tiRNA : small RNA的5’区域 Pre-miRNA: pre-miRNA的loop设计 tRNA: 成熟tRNA的反密码子环序列 snoRNA: snoRNA全长中的特异性序列 |
| 探针特异性 | small RNA特异性 |
| miRNA数目 | 2,627 (1,318个5-p-miRNAs, 1,309个3-p-miRNAs) |
| pre-miRNAs数目 | 1,745 |
| tRFs&tiRNAs数目 | 4254 |
| mature-tRNAs数目 | 346 |
| snoRNAs数目 | 955 |
| Small RNA来源数据库 |
miRNA: miRBase (v22) pre-miRNA: miRBase (v22) tRF&tiRNA: tRFdb, MINTbase, GtRNADb (更新至 18.1 2019.08) tRNA: GtRNADb(更新至 18.1 2019.08), ENSEMBL(v99) snoRNA: ENSEMBL(v99) 文献: 公开发表的文献至 2019 (references 1-40)
|
| 芯片规格 | 8 x 15K |
Arraystar Mouse small RNA 芯片 V1.0
| 探针总数 | 14,192 |
|---|---|
| 探针设计策略 | 整个探针由5’cap区, small RNA特异性区和3’linker区组成。 |
| 探针结合位点 |
5-p-miRNA 和 5'tRF&tiRNA: small RNA的3’区域 3-p-miRNA 和 3'tRF&tiRNA: small RNA的5’区域 Pre-miRNA: pre-miRNA的loop环设计 tRNA: 成熟tRNA的反密码子环序列 snoRNA: snoRNA全长中的特异性序列 |
| 探针特异性 | small RNA特异性 |
| miRNA数目 | 1949 (966个5-p-miRNAs, 983个3-p-miRNAs) |
| pre-miRNAs数目 | 1,122 |
| tRFs&tiRNAs数目 | 1,767 |
| mature-tRNAs数目 | 270 |
| snoRNAs数目 |
1,324 |
| Small RNA来源数据库 |
miRNA: miRBase (v22) pre-miRNA: miRBase (v22) tRF&tiRNA : tRFdb, GtRNADb (更新至18.1 2019.08) tRNA: GtRNAdb(更新至to 18.1 2019.08), ENSEMBL(v99) snoRNA: ENSEMBL(v99) 文献: 公开发表的文献至 2019 [1-40] |
| 芯片规格 | 8 x 15K |
Arraystar Rat small RNA 芯片 V1.0
| 探针总数 | 14,238 |
|---|---|
| 探针设计策略 | 整个探针由5’cap区, small RNA特异性区和3’linker区组成。 |
| 探针结合位点 |
5-p-miRNA 和 5'tRF&tiRNA : small RNA的3’区域 3-p-miRNA 和 3'tRF&tiRNA : small RNA的5’区域 miRNAs和tRF&tiRNA : small RNA 的3'序列 Pre-miRNA: pre-miRNA的loop环设计 tRNA: 成熟tRNA的反密码子环序列snoRNA: snoRNA全长中的特异性序列 |
| 探针特异性 | small RNA特异性 |
| miRNA数目 | 749 (355个5-p-miRNAs, 394个3-p-miRNAs) |
| pre-miRNAs数目 | 448 |
| tRFs&tiRNAs数目 | 1,135 |
| mature-tRNAs数目 | 197 |
| snoRNAs数目 |
1,486 |
| Small RNA来源数据库 |
miRNA: miRBase (v22) pre-miRNA: miRBase (v22) tRF&tiRNA : tRFdb, GtRNADb (更新至18.1 2019.08) tRNA: GtRNAdb(更新至to 18.1 2019.08), ENSEMBL(v99) snoRNA: ENSEMBL(v99) |
| 芯片规格 |
8 x 15K |
References
1. Guzzi N et al: Pseudouridylation of tRNA-Derived Fragments Steers Translational Control in Stem Cells. Cell 2018, 173(5):1204-1216 e1226.[PMID: 29628141]
2. Keam SP et al: The human Piwi protein Hiwi2 associates with tRNA-derived piRNAs in somatic cells. Nucleic Acids Res 2014, 42(14):8984-8995.[PMID: 25038252]
3. Keam SP, Sobala A, Ten Have S, Hutvagner G: tRNA-Derived RNA Fragments Associate with Human Multisynthetase Complex (MSC) and Modulate Ribosomal Protein Translation. J Proteome Res 2017, 16(2):413-420.[PMID: 27936807]
4. Zhang X et al: IL-4 Inhibits the Biogenesis of an Epigenetically Suppressive PIWI-Interacting RNA To Upregulate CD1a Molecules on Monocytes/Dendritic Cells. J Immunol 2016, 196(4):1591-1603.[PMID: 26755820]
5. Honda S et al: The biogenesis pathway of tRNA-derived piRNAs in Bombyx germ cells. Nucleic Acids Res 2017, 45(15):9108-9120.[PMID: 28645172]
6. Cole C et al: Filtering of deep sequencing data reveals the existence of abundant Dicer-dependent small RNAs derived from tRNAs. RNA 2009, 15(12):2147-2160.[PMID: 19850906]
7. Sobala A, Hutvagner G: Small RNAs derived from the 5' end of tRNA can inhibit protein translation in human cells. RNA Biol 2013, 10(4):553-563.[PMID: 23563448]
8. Lee YS, Shibata Y, Malhotra A, Dutta A: A novel class of small RNAs: tRNA-derived RNA fragments (tRFs). Genes Dev 2009, 23(22):2639-2649.[PMID: 19933153]
9. Huang B et al: tRF/miR-1280 Suppresses Stem Cell-like Cells and Metastasis in Colorectal Cancer. Cancer Res 2017, 77(12):3194-3206.[PMID: 28446464]
10. Kuscu C et al: tRNA fragments (tRFs) guide Ago to regulate gene expression post-transcriptionally in a Dicer-independent manner. RNA 2018, 24(8):1093-1105.[PMID: 29844106]
11. Kim HK et al: A transfer-RNA-derived small RNA regulates ribosome biogenesis. Nature 2017, 552(7683):57-62.[PMID: 29186115]
12. Kim HK et al: A tRNA-Derived Small RNA Regulates Ribosomal Protein S28 Protein Levels after Translation Initiation in Humans and Mice. Cell Rep 2019, 29(12):3816-3824 e3814.[PMID: 31851915]
13. Yeung ML et al: Pyrosequencing of small non-coding RNAs in HIV-1 infected cells: evidence for the processing of a viral-cellular double-stranded RNA hybrid. Nucleic Acids Res 2009, 37(19):6575-6586.[PMID: 19729508]
14. Schorn AJ, Gutbrod MJ, LeBlanc C, Martienssen R: LTR-Retrotransposon Control by tRNA-Derived Small RNAs. Cell 2017, 170(1):61-71 e11.[PMID: 28666125]
15. Maute RL et al: tRNA-derived microRNA modulates proliferation and the DNA damage response and is down-regulated in B cell lymphoma. Proc Natl Acad Sci U S A 2013, 110(4):1404-1409.[PMID: 23297232]
16. Ruggero K et al: Small noncoding RNAs in cells transformed by human T-cell leukemia virus type 1: a role for a tRNA fragment as a primer for reverse transcriptase. J Virol 2014, 88(7):3612-3622.[PMID: 24403582]
17. Falconi M et al: A novel 3'-tRNA(Glu)-derived fragment acts as a tumor-suppressor in breast cancer by targeting nucleolin. FASEB J 2019:fj201900382RR.[PMID: 31560576]
18. Zhou K et al: A tRNA fragment, tRF5-Glu, regulates BCAR3 expression and proliferation in ovarian cancer cells. Oncotarget 2017, 8(56):95377-95391.[PMID: 29221134]
19. Goodarzi H et al: Endogenous tRNA-Derived Fragments Suppress Breast Cancer Progression via YBX1 Displacement. Cell 2015, 161(4):790-802.[PMID: 25957686]
20. Natt D et al: Human sperm displays rapid responses to diet. PLoS Biol 2019, 17(12):e3000559.[PMID: 31877125]
21. Veneziano D et al: Dysregulation of different classes of tRNA fragments in chronic lymphocytic leukemia. Proc Natl Acad Sci U S A 2019, 116(48):24252-24258.[PMID: 31723042]
22. Haussecker D et al: Human tRNA-derived small RNAs in the global regulation of RNA silencing. RNA 2010, 16(4):673-695.[PMID: 20181738]
23. Balatti V et al: tsRNA signatures in cancer. Proc Natl Acad Sci U S A 2017, 114(30):8071-8076.[PMID: 28696308]
24. Cho H et al: Regulation of La/SSB-dependent viral gene expression by pre-tRNA 3' trailer-derived tRNA fragments. Nucleic Acids Res 2019, 47(18):9888-9901.[PMID: 31504775]
25. Babiarz JE et al: Mouse ES cells express endogenous shRNAs, siRNAs, and other Microprocessor-independent, Dicer-dependent small RNAs. Genes Dev 2008, 22(20):2773-2785.[PMID: 18923076]
26. Hasler D et al: The Lupus Autoantigen La Prevents Mis-channeling of tRNA Fragments into the Human MicroRNA Pathway. Mol Cell 2016, 63(1):110-124.[PMID: 27345152]
27. Pekarsky Y et al: Dysregulation of a family of short noncoding RNAs, tsRNAs, in human cancer. Proc Natl Acad Sci U S A 2016, 113(18):5071-5076.[PMID: 27071132]
28. Liao JY et al: Deep sequencing of human nuclear and cytoplasmic small RNAs reveals an unexpectedly complex subcellular distribution of miRNAs and tRNA 3' trailers. PLoS One 2010, 5(5):e10563.[PMID: 20498841]
29. La Ferlita A et al: Identification of tRNA-derived ncRNAs in TCGA and NCI-60 panel cell lines and development of the public database tRFexplorer. Database (Oxford) 2019, 2019.[PMID: 31735953]
30. Honda S et al: Sex hormone-dependent tRNA halves enhance cell proliferation in breast and prostate cancers. Proc Natl Acad Sci U S A 2015, 112(29):E3816-3825.[PMID: 26124144]
31. Donovan J, Rath S, Kolet-Mandrikov D, Korennykh A: Rapid RNase L-driven arrest of protein synthesis in the dsRNA response without degradation of translation machinery. RNA 2017, 23(11):1660-1671.[PMID: 28808124]
32. Hanada T et al: CLP1 links tRNA metabolism to progressive motor-neuron loss. Nature 2013, 495(7442):474-480.[PMID: 23474986]
33. Saikia M et al: Angiogenin-cleaved tRNA halves interact with cytochrome c, protecting cells from apoptosis during osmotic stress. Mol Cell Biol 2014, 34(13):2450-2463.[PMID: 24752898]
34. Wang Q et al: Identification and functional characterization of tRNA-derived RNA fragments (tRFs) in respiratory syncytial virus infection. Mol Ther 2013, 21(2):368-379.[PMID: 23183536]
35. Deng J et al: Respiratory Syncytial Virus Utilizes a tRNA Fragment to Suppress Antiviral Responses Through a Novel Targeting Mechanism. Mol Ther 2015, 23(10):1622-1629.[PMID: 26156244]
36. Zhou J et al: Identification of two novel functional tRNA-derived fragments induced in response to respiratory syncytial virus infection. J Gen Virol 2017, 98(7):1600-1610.[PMID: 28708049]
37. Yang X et al: 5-methylcytosine promotes mRNA export - NSUN2 as the methyltransferase and ALYREF as an m(5)C reader. Cell Res 2017, 27(5):606-625.[PMID: 28418038]
38. Ivanov P et al: Angiogenin-induced tRNA fragments inhibit translation initiation. Mol Cell 2011, 43(4):613-623.[PMID: 21855800]
39. Ivanov P et al: G-quadruplex structures contribute to the neuroprotective effects of angiogenin-induced tRNA fragments. Proc Natl Acad Sci U S A 2014, 111(51):18201-18206.[PMID: 25404306]
40. Schaffer AE et al: CLP1 founder mutation links tRNA splicing and maturation to cerebellar development and neurodegeneration. Cell 2014, 157(3):651-663.[PMID: 24766810]
数据分析结果包括检测指标的荧光信号值、统计学分析、注释信息和可供发表的高质量图表。
差异表达的small RNAs

Mature-ID: 成熟的miRNA在miRBase ID .
FC: 两组比较的差异倍数.
P-value: t-test的p值.
q-value (FDR): p-value的校正值(FDR).
Mature-Sequence: 成熟miRNA的序列.
Seed: 成熟miRNA的种子序列. (成熟miRNA的2-8位碱基).
High_Confidence: miRNA有强有力证据支持作为一个经典的miRNA基因.
Type: MirGeneDB基于表达水平注释miRNA类型.
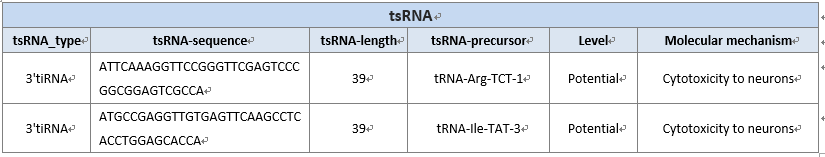
tRF&tiRNA _type: tRF&tiRNA type (tRF-5, tRF-3, tRF-1, 5-Leader, 5-tiRNA, 3-tiRNA和i-tRF).
tRF&tiRNA -sequence:tRF&tiRNA 的序列.
tRF&tiRNA -length:tRF&tiRNA 的长度.
tRF&tiRNA -precursor: pre-tRF&tiRNA 的基因名.
Level: tRF&tiRNA 的可信水平
Functional - 具有特定的生物学功能或疾病关联的记录;
Reliable - 在tRFdb中记录或有文献报道,但未作进一步研究;
Potential - 由Arraystar根据RNA片段长度和tRNA中的切割位置进行预测。
Mechanism: tRF&tiRNA 的分子机制.
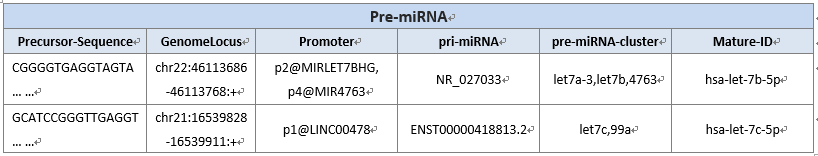
Precursor-Sequence: pre-miRNA序列.
GenomeLocus: miRNA gene在基因组上的定位.
Promoter: miRNA 基因在Fantom5的启动子名字.
pri-miRNA: 在Fantom5数据库中miRNA初级转录本(仅适用于人类和小鼠).
pre-miRNA-cluster: 一组在同一染色体和DNA链上的mirna间距离<10kb的pre-miRNA。
Mature-ID: 该pre-miRNA产生的mature miRNA(不是star miRNA)的ID.
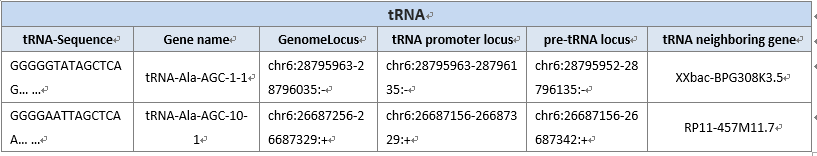
Sequence: tRNA isodecoder的序列.
Gene name: isodecoder tRNA的基因名.
GenomeLocus: tRNA isodecoder在基因组上的定位.
tRNA promoter Locus: tRNA isodecoder 启动子在基因组上的定位.
tRNA promoter – 包括一个tRNA的基因加上 100bp的序列 (PMC6108506).
pre-tRNA locus:precursor tRNA isodecoder在基因组上的定位. .
pre-tRNA - precursor tRNA 其中包括一个tRNA基因加上100个碱基对上游序列和一个3’leader
tRNA neighboring gene: tRNA isodecoder最邻近基因名.
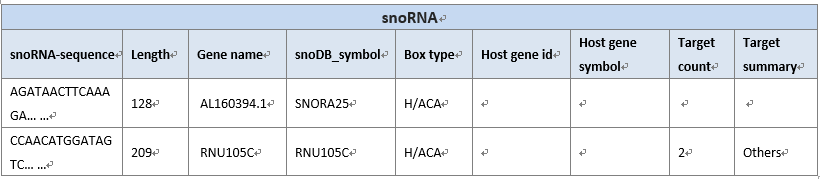
snoRNA-sequence: snoRNA的序列.
Length: snoRNA长度.
Gene name: snoRNA的基因名.
snoDB_symbol: snoRNA 在snoDB数据库的基因名 (http://scottgroup.med.usherbrooke.ca/snoDB/).
Box type: 基于高度保守基序的snoRNA盒类型(H/ACA和C/D).
Host gene id:内含子序列与snoRNA重叠的基因ID。
Host gene symbol: snoRNA宿主基因的基因名
Target count: snoRNA靶标数目
Target summary: snoRNA 靶标类型(lncrna, mirna, ncrna, protein coding, pseudogene, rrna, snorna, snrna, trna 和其他).
差异表达Small RNAs的层次聚类热图
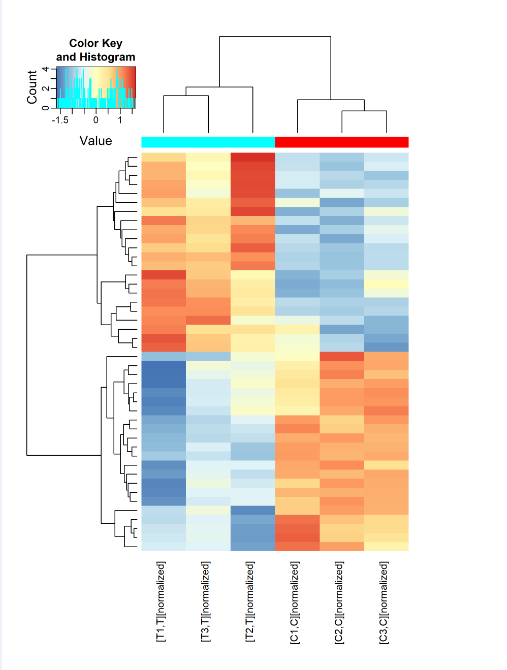
图1。差异表达miRNA的层次聚类热图。miRNA的表达水平用左上角颜色键中引用的红蓝色标度表示。顶部的树状图显示了样本之间表达谱的相对接近度。样本分组情况由热图上方的颜色条表示。

