

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
相关服务
超级增强子lncRNA芯片 LncPath™疾病/信号通路特异性芯片 T-UCR芯片 lncRNA-qPCR mRNA&lncRNA表观转录组芯片相关产品
nrStar™ Functional LncRNA PCR 芯片(H/M)相关资源
Arraystar lncRNA芯片检测的三个不可替代的要素 Arraystar Human LncRNA V5.0版本——完备、强化的全长lncRNA收录 lncRNAs的详细注释和功能分析 【SCI 20分客户成果】P53响应的lncRNA GUARDIN参与维护基因组的稳定 芯片更适用于低丰度RNA分析 客户成果丨lncRNA JPX通过ceRNA机制调控肺癌的发生发展和转移 客户成果丨LncRNA-APC1通过减少癌细胞外泌体的产生抑制结直肠癌的发生发展长链非编码RNA (LncRNA)是一类长度超过200nt的RNA,它们本身并不编码蛋白,而是以RNA的形式在多种层面上(表观遗传调控、转录调控以及转录后调控等)调控基因的表达水平。近年来的研究表明:LncRNA广泛参与各种生物学过程,LncRNA的异常表达与包括癌症在内的多种疾病密切相关。通过LncRNA芯片,研究人员能够快速高通量的获得与特定生物学过程或者疾病相关的LncRNA的表达变化,从而为后续的LncRNA功能研究或生物标志物筛选提供极大的便利。
Arraystar LncRNA芯片可同时检测LncRNA和mRNA,迄今为止,国内客户采用Arraystar LncRNA芯片已发表的SCI论文已超600篇,其中多篇发表在Cancer Cell,Mol Cell,Hepatology,Blood,EMBO等国际顶尖杂志上。
现Arraystar lncRNA芯片已全面升级至人V.50,小鼠V4.0,大鼠V3.0,新增了lncRNA全长信息,并更新或增加了其它注释。
芯片特点
• 检测lncRNA表达量灵敏度高、技术成熟,优于RNA-seq Learn more>>
• 收录有完备的全长lncRNA*,覆盖所有权威数据库与重要文章中的lncRNA全长信息 Learn more>>
• 注释lncRNA更系统、更专业,包含基因组信息、表观基因组信息*、序列完整性*、亚细胞定位**、miRNA识别位点… Learn more>>
• 设计针对lncRNA不同转录本异构体的转录本特异性探针,对不同转录本检测更准确、特异性更高 Learn more>>
• 同时检测lncRNA与mRNA,便于调控型lncRNA与编码蛋白的mRNA之间共表达及关联研究
*适用于Human V5.0 ** 适用于Human V5.0 和 Mouse V4.0
Aksomics(原康成生物)是Arraystar中国区唯一代理商,独家为您提供Arraystar公司长链非编码RNA芯片全程一站式技术服务。您只需要提供保存完好的组织或细胞标本,Aksomics的芯片技术服务人员就可为您完成全部实验操作,并提供完整的实验报告。同时,根据您的研究需要,Aksomics还提供各种深入数据挖掘服务。
Arraystar公司LncRNA芯片产品列表
| 服务 | 芯片 | 规格 | 描述 |
|---|---|---|---|
| Human LncRNA Microarray Service | Human LncRNA Array V5.0 | 8x60K | 39,317 LncRNAs and 21,174 mRNAs |
| Mouse LncRNA Microarray Service | Mouse LncRNA Array V4.0 | 8×60K | 37,949 LncRNAs and 22,692 mRNAs |
| Rat LncRNA Microarray Service | Rat LncRNA Array V3.0 | 4×44K | 10,333 LncRNAs and 28,287 mRNAs |
比RNA-Seq更适合于lncRNA表达谱检测
lncRNA一般表达水平低,且在低水平即可发挥功能,使用RNA-Seq检测时常常被高丰度的RNA所遮盖。以下是RNA-seq进行lncRNA表达研究的局限性:
• 由于lncRNAs丰度低和缺少全长注释信息,导致lncRNAs的定量不准。
• RNA-Seq对剪接体覆盖度差,通常缺少跨越剪接位点的reads,难以准确检测lncRNA转录本异构体
• 分析缺少公共lncRNA数据库,无法快速的系统性注释和分析lncRNA
>>Learn more
完备、强化的全长lncRNA内容
与具有详尽注释的蛋白编码基因不同,lncRNAs常常缺乏注释,信息分散且收集不全。Arraystar拥有高质量的转录组和lncRNA数据库,对各种来源的lncRNA进行了全面收集,包括所有权威数据库、高水平文章以及通过独家自有收集流程所得到的lncRNA。
Arraystar 人 lncRNA芯片 V5.0: FANTOM5 CAT (v1), GENECODE (v29), RefSeq (更新至 2018.11), BIGTranscriptome (v1), knownGene (更新至2018.11), LncRNAdb, LncRNAWiki, RNAdb, NRED, CLS FL, NONCODE (v5), MiTranscriptome (v2), 从超过 47 Tb RNA-seq数据中收集lncRNA. 共包含 39,317 lncRNAs,主要分为金标准lncRNA和可靠的lncRNA两大类. Learn more>>
Arraystar小鼠 lncRNA芯片 V4.0: GENECODE(VM19), RefSeq(更新至2018.11), KnownGene(更新至 2018.11), GenBank.
Arraystar 大鼠 lncRNA芯片 V3.0: Ensembl (92.rn6), RefSeq(更新至2019.1.1).
lncRNAs的详细注释和功能分析
一站式芯片结果包含系统、详细和专业的lncRNA注释,如基因组信息、表观基因组信息、子类分析、转录本序列完整性、亚细胞定位、miRNA识别位点、物种保守性、组织/细胞特异性及短肽编码潜力、相关的生物学过程以及疾病相关性,帮助您深入了解lncRNAs的生物功能和分子机制。 Learn more> >
同时检测lncRNA和蛋白编码mRNA的表达,便于调控型lncRNA与编码蛋白mRNA之间共表达及关联研究
Arraystar 人LncRNA 芯片V5.0
| 探针总数 | 60,491 |
| 探针长度 | 60 nt |
| 探针结合位点 | 转录本的外显子或剪接位点处设计特异性探针 |
| 探针特异性 | 转录本特异性 |
| 蛋白编码mRNAs | 21,174 |
| LncRNAs | 39,317 (8,393金标准LncRNAs 和 30,924可靠的 LncRNAs) |
| mRNA来源 | Refseq, UCSC, GENCODE, FANTOM5 CAT |
| LncRNA来源 |
Arraystar LncRNA collection pipelines: lncRNAs from all major databases and literatures up to 2018. “Canonical” or “longest” priority assigned to the transcript for each lncRNA gene. External Databases (current in 2018): FANTOM5 CAT (v1), GENCODE (v29), RefSeq (Updated to 2018.11), BIGTranscriptome (v1), knownGene (Updated to 2018.11), LncRNAdb, LncRNAWiki, RNAdb, NRED, CLS FL, NONCODE (v5), MiTranscriptome (v2) Literatures: Scientific publications up to 2018 |
| 芯片规格 | 8 × 60 K |
Arraystar 小鼠LncRNA 芯片V4.0
| 探针总数 | 60,641 |
| 探针长度 | 60 nt |
| 探针结合位点 | 转录本的外显子或剪接位点处设计特异性探针 |
| 探针特异性 | 转录本特异性 |
| 蛋白编码mRNAs | 22,692 |
| LncRNAs | 37,949 |
| mRNA来源 | Refseq, Known Gene, GENCODE |
| LncRNA来源 |
Arraystar LncRNA collection pipelines: lncRNAs from all major databases and literatures up to 2018. “Canonical” or “longest” priority assigned to the transcript for each lncRNA gene. External Databases (current in 2018): GENCODE(VM19), RefSeq, KnownGene, GenBank Literatures: Scientific publications up to 2018 |
| 芯片规格 | 8 × 60 K |
Arraystar大鼠LncRNA芯片V3.0
| 探针总数 | 38,620 |
| 探针长度 | 60 nt |
| 探针结合位点 | 转录本的外显子或剪接位点处设计特异性探针 |
| 探针特异性 | 转录本特异性 |
| 蛋白编码mRNAs | 28,287 |
| LncRNAs | 10,333 |
| mRNA来源 | Refseq, Ensembl |
| LncRNA来源 |
Arraystar LncRNA collection pipelines: lncRNAs from all major databases and literatures up to 2018. “Canonical” or “longest” priority assigned to the transcript for each lncRNA gene. External Databases (current in 2018): Refseq, Ensembl Literatures: Scientific publications up to 2018 |
| 芯片规格 | 4 × 44 K |
1.样品总RNA抽提
若实验对象为组织样品,取适量(50-100mg)新鲜组织样品或正确保存的组织样品,加1ml的RNA抽提试剂Trizol(Invitrogen),匀浆后抽提RNA。
若实验对象为细胞样品,每份样品取1×106~1×107细胞,完全吸去培养液后加1ml的RNA抽提试剂Trizol(Invitrogen),裂解后抽提RNA。
2. RNA质量检测
使用Nanodrop测定RNA在分光光度计260nm、280nm和230nm的吸收值,以计算浓度并评估纯度。
使用甲醛变性琼脂糖凝胶电泳检测RNA纯度及完整性
3. cDNA样品合成和标记
4.标记效率质量检测
使用Nanodrop检测荧光标记效率,以保证后续芯片实验结果的可靠性。
5.芯片杂交
在标准条件下将标记好的探针和高密度芯片进行杂交。
6.图像采集和数据分析
使用GenePix 4000B芯片扫描仪扫描芯片的荧光强度,并将实验结果转换成数字型数据保存,使用配套软件对原始数据进行分析运算。
7.提供实验报告
芯片扫描图
实验方法中英文报告
RNA质检报告
芯片数据结果报告,包括差异表达LncRNA列表,差异表达基因列表
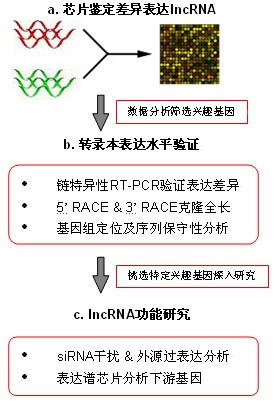
基本数据分析
1. 差异LncRNA的筛选
对于每个实验组有三次及三次以上生物学重复的实验设计,我们应用T检验筛选任意两个实验组之间差异表达的LncRNA,方差分析(ANOVA)则用于从三个以上实验组筛选差异表达的LncRNA,并以邻近(<100kb)mRNAs进行注释。Aksomics除了提供常规的通过p值筛选的差异LncRNA,还提供通过 FDR筛选的差异LncRNA。与p值筛选相比,FDR筛选对多重筛选过程中的假阳性率进行控制,是一种更加严谨的筛选方法,更适合于生物学重复较多的临床样本。
适用范围:一般用于两组或多组实验状态下的比较,建议每组实验至少有3次生物学重复。

2. 差异表达的LncRNA的聚类图
为了全面而直观地展示样品之间的关系及基因表达差异情况,将表达基因做层次聚类分析。用挑选的基因的表达情况来计算样品直接的相关性。一般来说,同一类样品能通过聚类出现在同一个簇(Cluster)中,聚在同一个簇的基因可能具有类似的生物学功能。
适用范围:两组或多组样品的表达谱数据

3. 差异LncRNA的邻近基因分析
很多LncRNA是通过调控邻近发挥生物学功能,因此通过邻近基因的分析可以为后续LncRNA的功能研究提供线索。Aksomics将差异LncRNAs数据与其临近(< 100 kb)mRNAs的差异表达数据整合,以期提供LncRNAs的功能推断。
适用范围:两组或多组数据比较获得的差异LncRNA
1) GO分析
Gene Ontology(简称GO)是基因功能国际标准分类体系。GO可分为分子功能(Molecular Function),生物过程(Biological Process)和细胞组成(Cellular Component)三个部分。Aksomics对位于差异LncRNA附近(< 100 kb),并且差异表达的蛋白编码基因进行了GO分析。这一方面便于客户从整体上了解差异LncRNA所涉及的GO条目,另一方面也为客户挑选LncRNA提供了线索:客户可以挑选与感兴趣的GO条目相关的差异LncRNA进行后续研究。
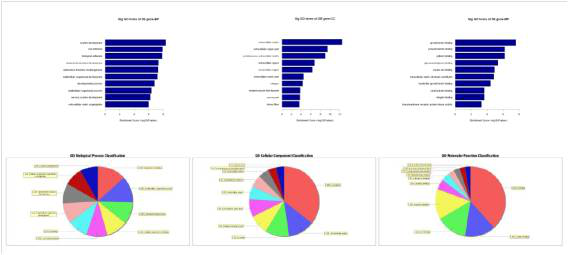
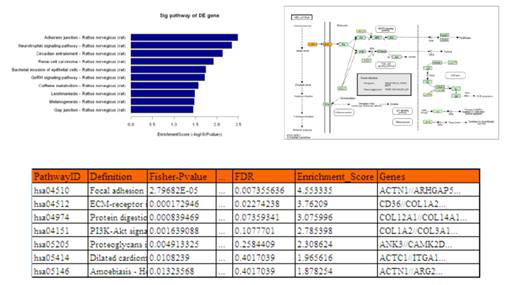
2) Pathway分析
Aksomics对位于差异LncRNA附近(< 100 kb),并且差异表达的蛋白编码基因进行了Pathway分析。这一方面便于客户从整体上了解差异LncRNA所参与的信号通路,另一方面也为客户挑选LncRNA提供了线索:客户可以挑选与感兴趣的信号通路有关的差异LncRNA进行后续研究。
4. LncRNAs的子类分析
适用范围:两组或多组数据比较获得的差异LncRNA
(1)差异antisence LncRNAs与相应mRNAs联合分析
在LncRNA中,研究得比较深入的是反义(Antisense) LncRNA,有超过30%的已注释的人类转录本有相应的反义LncRNA。这些反义LncRNA通过多种机制在转录水平或转录后水平调控相应的正义(sense)mRNA,从而发挥生物学功能。如下图中反义LncRNA诱导染色质和DNA的表观遗传改变,从而影响相应的正义mRNA的表达。Aksomics将差异antisense LncRNAs与相应的sense mRNAs信息进行整合,以推断LncRNAs的功能.
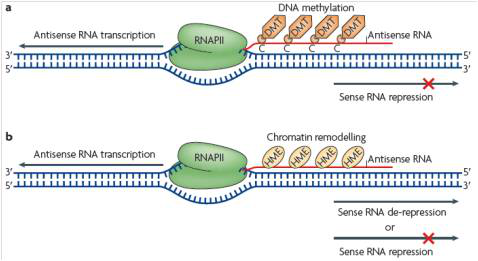
*图释:反义LncRNA诱导染色质和DNA的表观遗传改变,从而影响相应的正义mRNA的表达。

(2)差异Enhancer LncRNAs与相应mRNAs联合分析
LncRNA可以发挥类似增强子的功能,增强邻近蛋白质编码基因的表达,从而在发育和分化等过程中发挥关键作用。Aksomics对Ørom UA等人在人类细胞系中发现的3千多条具有增强子功能的LncRNA 进行了详细的注释,并将差异的Enhancer LncRNAs与相应的mRNAs(<300kb)进行了整合,以帮助客户推断这些LncRNAs的功能。
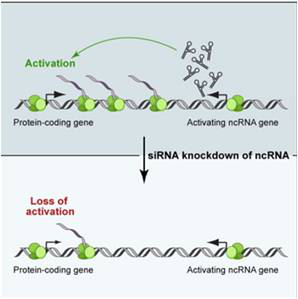
*图释:LncRNA作为增强子,增强邻近蛋白质编码基因表达水平;下图:经siRNA敲除ncRNA后,邻近蛋白质编码基因表达水平下降

(3)差异LincRNAs与相应mRNAs联合分析
lincRNAs是目前研究的热点之一。Aksomics根据Rinn等构建的lincRNAs数据库,将芯片中符合标准的差异LncRNA挑选出来与相应mRNA(< 300 kb)进行联合分析,以推断lincRNA功能。

高级数据分析
1. CNC(coding-noncoding gene co-expression)分析
CNC分析是一种通过LncRNA和mRNA共表达数据,将LncRNA与mRNA联系起来的分析方法。通过CNC分析可以发现与某个LncRNA具有相同表达模式的mRNA,通过这些mRNA的功能,可以将LncRNA与特定信号通路或疾病状况联系起来,从而便于预测LncRNA的功能,并揭示其作用机制。
适用范围:芯片数量≥6张
AksomicsCNC分析结果展示,下图数据来源于Aksomics客户已发表的文献(Hepatology,影响因子:12.003):
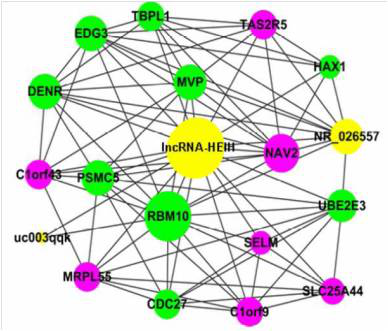
*图释:LncRNA-HEIH在HCC中的共表达网络。黄色的节点代表LncRNA,绿颜色的节点代表与肿瘤生长和药物耐受相关的蛋白编码基因,紫色节点代表功能未知的蛋白编码基因。
2. 生物标志物分析
Aksomics还为客户提供包括课题设计到数据分析在内的一站式biomarker分析,以常见的筛选预后biomarker为例,我们首先通过Cox回归模型筛选预后相关的LncRNA,然后通过Logistic regression,Nearest Template Prediction (NTP)等多种统计学方法构建预测模型,然后从中挑选出效果最理想的预测模型用于后续分析。无论是LncRNA的筛选,还是预测模型的建立,Aksomcis都通过严谨和科学的分析为客户发表文章提供帮助。
1.P53响应的lncRNA GUARDIN参与维护基因组稳定的功能机制研究(GUARDIN is a p53-responsive long non-coding RNA that is essential for genomic stability. Nature Cell Biology, 2018)IF:20.06
作者应用Arraystar Human LncRNA Array对带有WT p53表达系统以及没有p53表达的H1299人肺腺癌细胞的lncRNA表达谱进行了研究,研究筛选出了一个受p53调控的lncRNA GUARDIN。qPCR验证与芯片结果一致,生信分析发现GUARDIN有三个异构体,其中最长的亚型在p53诱导时含量最丰富。接下来作者通过过表达/敲除p53、qPCR、WB、免疫沉淀等实验验证了GUARDIN是p53响应的lncRNA。在HTC116细胞中,敲除GUARDIN,细胞数目、细胞活力、克隆形成及异移植都明显下降,表明GUARDIN是细胞存活和增殖所必需的。机制研究表明,GUARDIN可以结合miR-23a且能调控miR-23a的靶基因TRF2的表达。进一步研究发现GUARDIN可以作为分子支架介导BRCA1与BARD1的相互作用来调控BRCA1的表达。然后作者通过DNA损伤彗星实验发现,GUARDIN敲除后,DNA损伤加剧等,表明了GUARDIN对基因组稳定性的作用,而且TRF2和BRCA1共同过表达可以消除GUARDIN下调引起的DNA损伤。上述实验表明GUARDIN可以通过调控TRF2和BRCA1的表达促进基因组稳定性。
2.lncRNA GClnc1作为胃癌预后生物标志物及其调控胃癌发生发展的功能机制研究(LncRNA GClnc1 Promotes Gastric Carcinogenesis and May Act as a Modular Scaffold of WDR5 and KAT2A Complexes to Specify the Histone Modification Pattern. Cancer Discovery, 2016)IF: 19.783
该研究首先借助Arraystar lncRNA芯片检测了10个胃癌患者的癌组织和癌旁组织中lncRNA的表达情况。之后作者利用严格的筛选条件(P值<0.01, Foldchange>2和原始信号值>1500)从差异表达上调的lncRNA中选取了8个指标进行后续的大样本qPCR验证(n=165),以及临床相关性分析,发现lncRNA GClnc1的异常表达和胃癌的不良预后高度相关,预示它可以作为胃癌预后诊断的潜在biomarker。接着作者对于lncRNA GClnc1是如何调控胃癌的发生和发展进行了深入的功能和机制探究,首先通过GOF/LOF实验证明lncRNA GClnc1可以促进胃癌细胞的增殖、侵袭和转移,然后通过RNA-seq、CNC、ChIP以及ChIRP等细胞生物学实验和生物信息学分析来挖掘lncRNA GClnc1的调控靶点以及相应的调控机制。结果表明lncRNA GClnc1可以作为支架分子(scaffold)锚定在邻近基因SOD2的启动子区域,然后招募组蛋白修饰酶WDR5和KAT2A来顺式(cis)调控基因SOD2的表达,从而促进了胃癌的发生和发展。
→ H. pylori infection alters repair of DNA double-strand breaks via SNHG17 The Journal of Clinical Investigation . 2020
→ Long noncoding RNA HCP5 participates in premature ovarian insufficiency by transcriptionally regulating MSH5 and DNA damage repair via YB1 . Nucleic Acids Research . 2020
→ lncRNA JPX/miR-33a-5p/Twist1 axis regulates tumorigenesis and metastasis of lung cancer by activating Wnt/β-catenin signaling . Molecular Cancer . 2020
→ Roles of DANCR/microRNA-518a-3p/MDMA ceRNA network in the growth and malignant behaviors of colon cancer cells . BMC Cancer . 2020
→ Long non‐coding RNA mediates stroke‐induced neurogenesis . STEM CELLS . 2020
→ Functional Implications of Cathelicidin Antimicrobial Protein in Breast Cancer and Tumor-Associated Macrophage Microenvironment . Biomolecules . 2020
→ LncRNA Oprm1 overexpression attenuates myocardial ischemia/reperfusion injury by increasing endogenous hydrogen sulfide via Oprm1/miR-30b-5p/CSE axis . Life Sciences . 2020
→ MicroRNA‐127‐5p impairs function of granulosa cells via HMGB2 gene in premature ovarian insufficiency . Journal of Cellular Physiology . 2020
→ Identification and analysis of key lncRNAs in malignant-transformed BEAS-2B cells induced with coal tar pitch by microarray analysis . Environmental Toxicology and Pharmacology . 2020
→ A comprehensive evaluation of differentially expressed mRNAs and lncRNAs in cystitis glandularis with gene ontology, KEGG pathway, and ceRNA network analysis . Translational Andrology and Urology . 2020
→ Long noncoding RNA CCAT1 inhibits miR‐613 to promote nonalcoholic fatty liver disease via increasing LXRα transcription . Journal of Cellular Physiology . 2020
→ Inflammation and DNA methylation coregulate the CtBP-PCAF-c-MYC transcriptional complex to activate the expression of a long non-coding RNA CASC2 in acute pancreatitis . Int J Biol Sci . 2020
→ The Long Noncoding RNA ZFAS1 Potentiates the Development of Hepatocellular Carcinoma via the microRNA-624/MDK/ERK/JNK/P38 Signaling Pathway . OncoTargets and Therapy . 2020
→ A long noncoding RNA cluster-based genomic locus maintains proper development and visual function . Nucleic Acids Research . 2019
→ LncRNA PCAT1 activates AKT and NF-κB signaling in castration-resistant prostate cancer by regulating the PHLPP/FKBP51/IKKα complex . Nucleic acids research . 2019
→ m 6 A-induced lncRNA RP11 triggers the dissemination of colorectal cancer cells via upregulation of Zeb1 . Molecular cancer . 2019
→ Long non-coding RNA GBCDRlnc1 induces chemoresistance of gallbladder cancer cells by activating autophagy . Molecular cancer . 2019
→ NKILA lncRNA promotes tumor immune evasion by sensitizing T cells to activation-induced cell death . Nature immunology . 2018
→ Long Non-coding RNA GMAN, Upregulated in Gastric Cancer Tissues, is Associated with Metastasis in Patients and Promotes Translation of Ephrin A1 by Competitively Binding GMAN-AS . Gastroenterology . 2018
→ GUARDIN is a p53-responsive long non-coding RNA that is essential for genomic stability . Nature Cell Biology . 2018
→ Long noncoding RNA lnc-TSI inhibits renal fibrogenesis by negatively regulating the TGF-β/Smad3 pathway . Science translational medicine . 2018
→ A 3-LncRNA Risk Scoring System for Prognosis of Adult Acute Myeloid Leukemia . Blood . 2018
→ GUARDIN is a p53-responsive long non-coding RNA that is essential for genomic stability. Nature Cell Biology. 2018

