

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
tRNA是基因与蛋白之间重要的信息传递者,通过与特定氨基酸结合而发挥功能。越来越多的文献表明,tRNA及其来源的RNA片段(tRFs)在细胞增殖、分化、代谢、应激反应以及多种疾病中发挥着调控作用[1]。tRNA携带种类丰富、数目众多的转录后修饰。这些修饰为tRNA功能发挥所必需,参与tRNA的正确折叠,稳定维持和基因解码功能。比如,位于颈环部位的修饰影响tRNA的折叠与稳定性,反密码子环上的修饰确保翻译准确性,34号位置的修饰决着密码子的摆动性,反密码子环附近的修饰调控密码子与反密码子识别(图1)。
tRNA修饰受各类tRNA修饰相关蛋白的动态调控[2],这些蛋白突变或者功能紊乱,与多种疾病的发生有关。例如,NSUN2负责催化胞嘧啶5’甲基化,其突变会导致机体头小畸形症[3];t6A甲硫基转移酶CDKAL1功能紊乱可诱发Ⅱ型糖尿病[4]。tRNA修饰对于疾病治疗的重要性,以及这些修饰是如何被调控,仍待进一步的研究[5,6]。
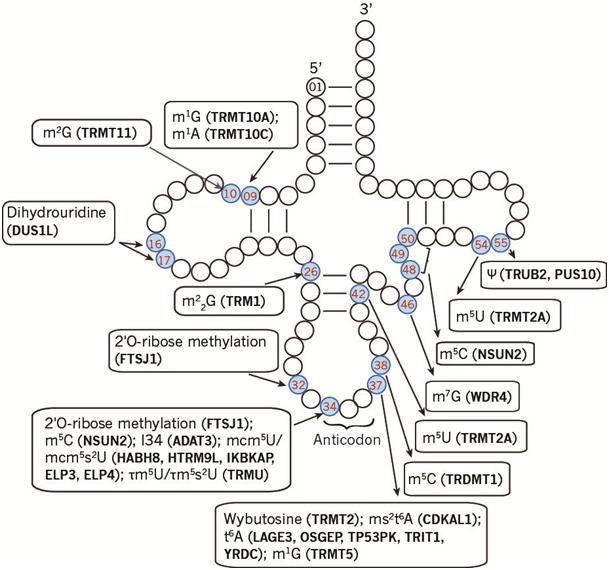
图1. 人类tRNA修饰类型与修饰相关蛋白。
美国Arraystar公司发布了聚焦tRNA修饰蛋白检测的PCR芯片 ——NuRNA™ tRNA Modification Enzymes PCR Array。参考已有文献与权威数据库(UniProt和Modomics),此款芯片囊括了85个tRNA修饰酶和蛋白因子,覆盖了所有已知的tRNA修饰酶。PCR芯片上的每对引物在多个组织和细胞系中通过了严格验证。
Aksomics(原康成生物)是Arraystar公司中国地区唯一代理商,独家为您提供Arraystar公司PCR芯片一站式技术服务,您只需要的提供保存完好的的组织或者细胞标本,Aksomics的芯片技术服务人员可为您完成全部操作,并提供完整的实验报告。
产品信息:
|
服务名称
|
芯片
|
规格
|
描述
|
|
NuRNA™ Human tRNA Modification Enzymes PCR Array Service
|
NuRNA™ Human tRNA Modification Enzymes PCR Array
|
384-well(4*96)/ plate
|
同时检测85个tRNA修饰酶及相关蛋白因子
|
芯片特点:
• 覆盖度广—囊括了UniProt与Modomics中所有已知的tRNA修饰酶/蛋白因子
• 严谨性强—所有引物都通过了多样本严格验证
• 快速简易—即用型384孔板规格,只需将cDNA与qPCR Master Mix混合试剂加入PCR板中,4小时内得到实验结果。cDNA无需预先扩增。
tRNA修饰及相关蛋白 (85):
|
ADAT1(m1I37), ADAT2(m1I34), ADAT3(-), ALKBH1(mcm5U), ALKBH8(mchm5U34/mcm5s2U34/mcm5U34/mcm5Um34), C9orf64(-), CDK5RAP1(ms2A37/ms2 i6A37), CDKAL1(ms2A37/ms2 i6A37), CDKL1(ms2t6A), CTU1(mcm5S2U ), CTU2(mcm5S2U), DUS1L(D16/17), DUS2(D20), DUS3L(D47), DUS4L(D), ELP3(-), ELP4(mcm5s2U), FBLL1(-), FTSJ1(Cm32/Gm34), GTPBP3(tm5U34), HSD17B10(m1A9/m1G9), IKBKAP(-), KIAA0391(m1A9/m1G9), KIAA1456(Um), LAGE3(t6A37), LCMT2(o2yW), METTL1(m7G46), METTL2A(Cm), METTL2B(3mC), MOCS3(mcm5S2U ), MTO1(tm5U34), NAT10(Ac4C), NFS1(s2U34/tm5s2U34), NSUN2(m5C49), NSUN6(m5C72), OSGEP(t6A37), OSGEPL1(t6A37), PUS1(pseudouridine27/pseudouridine28), PUS10(pseudouridine 55), PUS3(pseudouridine39), PUS7L(pseudouridine), PUSL1(pseudouridine), QTRT1 (Q34), QTRT2 (Q34), RPUSD1(pseudouridine), RPUSD2(pseudouridine31/pseudouridine32), RPUSD3(pseudouridine), RPUSD4(pseudouridine31/pseudouridine32), SSB(-), TARBP1(-), THUMPD1(Acetylcytidine), THUMPD2(Gm), THUMPD3(Gm), TP53RK(t6A37), TPRKB(t6A37), TRDMT1(m5C38), TRIT1(i6A37), TRMO(m6t6A), TRMT1(m2G26/m22G26), TRMT10A(m1G9), TRMT10B(m1G9), TRMT10C(m1A9/m1G9), TRMT11(m2G10), TRMT112(mchm5U34/mcm5s2U34/mcm5U34/mcm5Um34), TRMT12(o2yW), TRMT13(m4C/m4A ), TRMT1L(m2G26/m22G26), TRMT2A(Um), TRMT2B(m5U54), TRMT44(Um44), TRMT5(m1G37/o2yW), TRMT6(m1A58), TRMT61A(m1A58), TRMT61B(m1A58), TRMU(s2U34/tm5s2U34), TRUB1(pseudouridine), TRUB2(pseudouridine55), TYW1(o2yW), TYW1B(o2yW), TYW3(o2yW), TYW5 (o2yW), UBA5(cyclic t6A), URM1(mcm5S2U), WDR4(m7G46), YRDC(t6A37)
|
注:tRNA修饰缩写参考Modomics数据库,基因名参考UniProt。
PCR芯片实验流程
1. RNA抽提与质量检测
进行RNA常规抽提,使用NanoDrop ND-1000检测RNA浓度和纯度,使用琼脂糖凝胶电泳检测RNA纯度和完整性。详细的样品QC结果见Arraystar服务报告。
2. cDNA合成
每样本取1.5 μg RNA,使用rtStar™ First-Strand Synthesis Kit (Cat# AS-FS-001, Arraystar) 试剂盒合成cDNA第一链。详细步骤参照Arraystar产品操作手册。
3. Real-time PCR扩增
将cDNA与 Arraystar SYBR Green qPCR Master Mix (Cat# AS-MR-005-5, Arraystar)混合,加入至384孔板中,在ABI 7900 PCR仪上进行Real-time PCR扩增。
4. 熔解曲线分析与原始数据导出
PCR扩增完成后,进行熔解曲线分析,使用PCR仪自带软件导出原始数据。出具Raw Data文件夹,包含原始Ct值、PCR扩增曲线图和熔解曲线图。
PCR芯片数据分析流程
1. PCR芯片数据质控
质控参数:Ct(Blank)>35; Ct(GDC)>35; Ct(RNA Spike-in)<25; Ct (PPC) <25. 符合上述条件的样本进入下一步分析。
2. 数据校正与△Ct值计算
板间校正:使用Ct(PPC)对不同PCR板进行板间校正。
内参校正与△Ct值计算:挑选优质内参,使用内参均值计算△Ct值。
3. 差异倍数计算 (2^(-△△Ct))
使用 △△Ct 方法计算不同样品组之间的表达差异倍数。
4. P值计算
对样本组进行t检验,计算P值。
5. 其他常规数据分析
散点图分析;火山图分析;TOP20表达上调和下调transcript柱形图分析
6. 提供服务报告与数据分析结果
a. Arraystar服务报告(包括RNA样本QC和详细实验数据分析步骤)
b. Excel芯片结果汇总表(包括Transcript列表,数据分析结果和各类图表)
c. Raw Data文件夹 (包含原始数据、扩增曲线图和熔解曲线图)
NuRNA™ Human tRNA Modification Enzymes PCR芯片服务部分结果展示
1. 差异表达转录本列表(默认筛选参数:差异倍数>2;P<0.05,客户可指定筛选参数值):
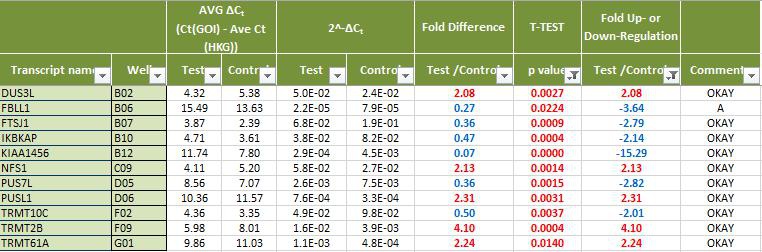
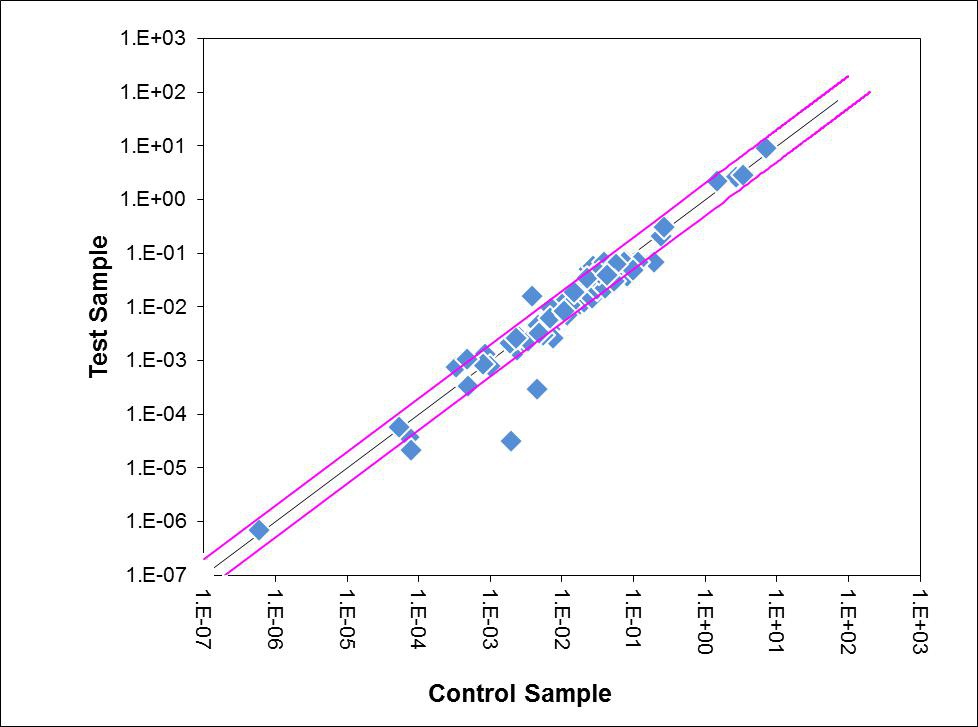
2. 散点图 (黑色斜线代表差异倍数为1,红色斜线代表差异倍数为2):
3. 火山图(黑色垂线代表差异倍数为1;粉色垂线代表上调或下调倍数为2;蓝色水平线代表P值为0.05):

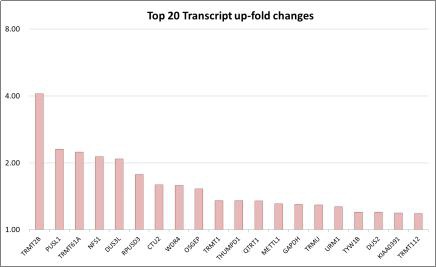
4. TOP20表达上调transcript柱状图:
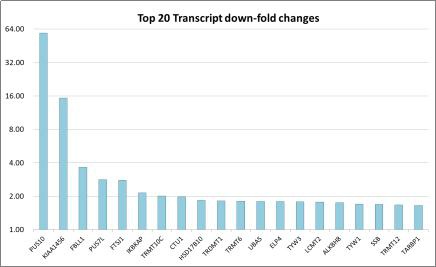
5. TOP20表达下调transcript柱状图:
参考文献:
[1] Kirchner S, Ignatova Z. Emerging roles of tRNA in adaptive translation, signalling dynamics and disease. Nature reviews Genetics 2015;16:98-112.
[2] El Yacoubi B, Bailly M, de Crecy-Lagard V. Biosynthesis and function of posttranscriptional modifications of transfer RNAs. Annual review of genetics 2012;46:69-95.
[3] Blanco S, Dietmann S, Flores JV, Hussain S, Kutter C, Humphreys P, et al. Aberrant methylation of tRNAs links cellular stress to neuro-developmental disorders. The EMBO journal 2014;33:2020-39.
[4] Zhou B, Wei FY, Kanai N, Fujimura A, Kaitsuka T, Tomizawa K. Identification of a splicing variant that regulates type 2 diabetes risk factor CDKAL1 level by a coding-independent mechanism in human. Human molecular genetics 2014;23:4639-50.
[5] Zhang X, Cozen AE, Liu Y, Chen Q, Lowe TM. Small RNA Modifications: Integral to Function and Disease. Trends in molecular medicine 2016.
[6] Suzuki T, Nagao A, Suzuki T. Human mitochondrial tRNAs: biogenesis, function, structural aspects, and diseases. Annual review of genetics 2011;45:299-329.
Small RNA是指长度小于200bp,不具蛋白编码能力的一类RNA分子。small RNAs不仅在细胞中具有较高的表达量,也广泛存在于各类体液中。根据长度、结构形态、功能形式以及合成途径的不同,科研人员将small RNAs分成了七种类型:microRNA (miRNA)、PIWI-interacting RNA (piRNA)、small interfering RNA (siRNA)、small nucleolar RNA (snoRNA)、small nuclear RNA (snRNA)、transfer RNA (tRNA)、tRNA-derived fragment (tRF&tiRNA)(图1)。这些small RNA分子在转录水平或转录后水平,参与调控基因表达[1-3]。

图 1. Small RNAs的分类与功能
small RNA的合成途径极其复杂,具体过程有待进一步的挖掘。在许多疾病中,科研人员都检测到了不同small
RNA的合成紊乱。例如,DROSHA与DICER1是miRNA合成过程中非常关键的两个蛋白,其表达水平会随着肿瘤的发生发展而变化,并可作为神经母细胞瘤预后诊断的潜在标记物[4]。近些年来,small RNA吸引了越来越多的关注,small RNA合成途径的研究也受到了更多的重视。
对于特定通路或聚焦领域中的基因而言,PCR芯片是其表达研究可靠便捷的手段。为了帮助研究人员方便快捷地分析small
RNA合成相关蛋白的表达水平,Arraystar开发了small
RNA合成蛋白检测PCR芯片。该芯片能够同时分析185个mRNA,它们都是在各类small RNAs合成过程中发挥关键作用的蛋白分子的表达水平,是small
RNAs研究的一件利器。
Aksomics是Arraystar公司中国地区唯一代理商,独家为您提供Arraystar公司PCR芯片一站式技术服务,您只需要的提供保存完好的的组织或者细胞标本,Aksomics的芯片技术服务人员可为您完成全部操作,并提供完整的实验报告。
产品信息:
|
芯片
|
规格
|
描述
|
|
NuRNA™ Human Small RNAs Biogenesis-Related Protein PCR Array
Service
|
384-well(2*192)/ plate
|
miRNAs(57个)+piRNAs(17个)+siRNAs(24个)+snoRNAs(24个)+snRNAs(41个)+tRNAs/tRFs(47个)
|
芯片特点:
• 覆盖度广—收集了GO通路中所有与small RNA合成相关的蛋白分子
• 严谨性强—所有引物都通过了多样本严格验证
• 快速简易—即用型384孔板规格,只需将cDNA与qPCR Master
Mix混合试剂加入PCR板中,4小时内得到实验结果。cDNA无需预先扩增。
表 1. 人类small RNAs合成相关蛋白(185个蛋白)
PCR芯片实验流程
1.RNA抽提与质量检测
进行RNA常规抽提,使用NanoDrop
ND-1000检测RNA浓度和纯度,使用琼脂糖凝胶电泳检测RNA纯度和完整性。详细的样品QC结果见Arraystar服务报告。
2.cDNA合成
每样本取1.5
μg RNA,使用rtStar™ First-Strand Synthesis Kit (Cat# AS-FS-001, Arraystar)
试剂盒合成cDNA第一链。详细步骤参照Arraystar产品操作手册。
3.Real-time
PCR扩增
将cDNA与 Arraystar SYBR Green qPCR Master Mix (Cat# AS-MR-005-5,
Arraystar)混合,加入至384孔板中,在ABI 7900 PCR仪上进行Real-time
PCR扩增。
4.熔解曲线分析与原始数据导出
PCR扩增完成后,进行熔解曲线分析,使用PCR仪自带软件导出原始数据。出具Raw
Data文件夹,包含原始Ct值、PCR扩增曲线图和熔解曲线图。
PCR芯片数据分析流程
1.PCR芯片数据质控
质控参数:Ct(Blank)>35;
Ct(GDC)>35; Ct(RNA Spike-in)<25; Ct (PPC) <25.
符合上述条件的样本进入下一步分析。
2.数据校正与△Ct值计算
板间校正:使用Ct(PPC)对不同PCR板进行板间校正。
内参校正与△Ct值计算:挑选优质内参,使用内参均值计算△Ct值。
3.差异倍数计算
(2^(-△△Ct))
使用 △△Ct
方法计算不同样品组之间的表达差异倍数。
4.P值计算
对样本组进行t检验,计算P值。
5.其他常规数据分析
散点图分析;火山图分析;TOP20表达上调和下调transcript柱形图分析
6.提供服务报告与数据分析结果
a.Arraystar服务报告(包括RNA样本QC和详细实验数据分析步骤)
b.Excel芯片结果汇总表(包括transcript列表,数据分析结果和各类图表)
c.Raw
Data文件夹 (包含原始数据、扩增曲线图和熔解曲线图)
NuRNA™ Human small RNA Biogenesis PCR芯片服务部分结果展示
1.差异表达转录本列表(默认筛选参数:差异倍数>2;P<0.05,客户可指定筛选参数值)

2. 散点图 (黑色斜线代表差异倍数为1,红色斜线代表差异倍数为2)

3. 火山图(黑色垂线代表差异倍数为1;粉色垂线代表上调或下调倍数为2;蓝色水平线代表P值为0.05)

4. TOP20表达上调transcript柱状图

5. TOP20表达下调transcript柱状图

参考文献
[1] Kim VN. Small RNAs:
classification, biogenesis, and function. Mol cells 19, 1-15. (2005),
[2]
Esteller M. Non-coding RNAs in human disease. Nat Rev Genet 12, 861-74. (2011),
PMID:22094949.
[3] Matera AG, et al. Non-coding RNAs: lessons from the small
nuclear and small nucleolar RNAs. Nat Rev Mol Cell Biol 8, 209-20. (2007),
PMID:17318225.
[4] Lin RJ, et al. microRNA signature and expression of Dicer
and Drosha can predict prognosis and delineate risk groups in neuroblastoma.
Cancer Res 70, 7841-50. (2010), PMID:20805302.
近年来,RNA修饰所介导的基因表达调控不断取得突破性进展,直接导致了表观转录组学(Epitranscriptomics)的诞生。研究发现,m6A、m5C、m1A、hm5C、甲尿嘧啶(ψ)与肌苷(I)等多种表观转录修饰存在于mRNA上,影响mRNA的代谢与功能[1],是一种重要的基因表达调控方式(图1)。这些表观转录组修饰由特定的酶添加(Writer)或去除(Eraser),可被特定的蛋白识别(Reader)。表观转录修饰水平的异常,以及相关酶或蛋白的异常,与许多疾病的发生紧密关联。
m6A是真核生物mRNA上含量最丰富的表观转录修饰,参与调控mRNA的二级结构、剪切、成熟、细胞定位与翻译等过程。m6A由甲基转移酶复合物(包含METTL3,METTL14,WTAP,KIAA1429,RBM15,RBM15B)催化产生,可被去甲基化酶ALKBH5或FTO去除。目前已发现了多种特异性识别m6A位点的蛋白或复合物,包括YTH家族蛋白(YTHDF1-3,
YTHDC1)、转录起始复合物eIF3、核糖核蛋白(HNRNPA2B1,HNRNPC)以及RNA结合蛋白SRSF2(表1)。m6A的动态调控在减数分裂与细胞多潜能分化等过程中发挥着重要作用[3]。与正常组织相比,肿瘤干细胞中的m6A修饰水平显著升高。高水平的m6A增强了多个关键肿瘤基因的蛋白表达,从而促进肿瘤干细胞的增殖与肿瘤的发生发展,且常与不良预后有关。此外,METTL3[4]、FTO[2]及ALKBH5[5]等表观转录相关蛋白因子在肿瘤的发生发中也发挥着关键性作用。在病毒RNA上也检测到了m6A修饰,其与病毒感染及复制有关。
显而易见,表观转录修饰与表观转录修饰相关蛋白在基因表达调控中发挥着至关重要的作用。然而,目前对其研究才刚刚起步。为了帮助研究人员更方便快捷的进行表观转录组学研究,Arraystar开发了检测表观转录修饰相关蛋白因子的PCR芯片。此款芯片可同时检测89个已知的或潜在的表观转录修饰相关蛋白,是表观转录研究的强有力工具。

|
|
m6A
|
m5C
|
I
|
ψ
|
m1A
|
|
Writers
|
KIAA1429
METTL3
METTL4
METTL14
RBM15
RBM15B
WTAP
|
NSUN2
TRDMT1
|
ADAR
ADARB1
ADARB2
|
DKC1
PUS1
PUS7
TRUB1
|
None identified
|
|
Erasers
|
ALKBH5
FTO
|
TET1
TET2
TET3
|
None identified
|
None identified
|
ALKBH1
ALKBH3
|
|
Readers
|
YTH Family
eIF3 complex
HNRNPA2B1
HNRNPC
SRSF2
|
None identified
|
None identified
|
None identified
|
None identified
|
Aksomics(原康成生物)是Arraystar公司中国地区唯一代理商,独家为您提供Arraystar公司PCR芯片一站式技术服务,您只需要的提供保存完好的的组织或者细胞标本,Aksomics的芯片技术服务人员可为您完成全部操作,并提供完整的实验报告。
产品信息:
|
芯片
|
规格
|
描述
|
|
NuRNA™ Human Epitranscriptomics PCR Array Service
|
384-well(4*96)/ plate
|
同时检测89个已知的或潜在的表观转录修饰相关蛋白
|
芯片特点:
• 覆盖度广—覆盖了所有目前已知的表观转录修饰相关蛋白
•
转录本水平检测—检测编码Uniprot经典蛋白的转录本
•
严谨性强—所有引物都通过了多样本严格验证
• 快速简易—即用型384孔板规格,只需将cDNA与qPCR
Master Mix混合试剂加入PCR板中,4小时内得到实验结果。cDNA无需预先扩增。
|
表观转录修饰相关蛋白检测列表
|
|
m6A
Writers: KIAA1429, METTL14, METTL3, METTL4, RBM15, RBM15B, WTAP
Readers: DGCR8, EIF3A, EIF3B, ELAVL1, HNRNPA2, HNRNPB1, HNRNPC1, HNRNPC2,
SRSF2, YTHDC1, YTHDC2, YTHDF1, YTHDF2, YTHDF3
Erasers: ALKBH5, FTO
m1A
Writers: HSD17B10, KIAA0391, TRMT10C, TRMT6, TRMT61A, TRMT61B
Erasers: ALKBH1, ALKBH3
m5C
Writers: DNMT1 , DNMT3A, DNMT3B, DNMT3L, NOP2, NSUN2, NSUN3, NSUN4, NSUN5,
TRDMT1
Readers: MECP2, UHRF1
Erasers: TET1, TET2, TET3
hm5C
Writers: TET1, TET2, TET3
Readers: CHTOP, EGR1, ERH, HMCES, MEP50, MGME1, NEIL1, PRMT1, PRMT5, SMUG1,
TDG, THYN1, WDR76, WT1
Inosine
Writers: ADAD1, ADAD2, ADAR, ADARB1, ADARB2, ADAT1, ADAT2, ADAT3
Pseudouridine
Writers: DKC1, GAR1, NAF1, NHP2, NOP10, PUS1, PUS10, PUS3, PUS7, PUS7L,
PUSL1, RPUSD1, RPUSD2, RPUSD3, RPUSD4, TRUB1, TRUB2
Other modifications (m7G)
CMTR1, CMTR2, RNGTT, RNMT
|
PCR芯片实验流程
1.RNA抽提与质量检测
进行RNA常规抽提,使用NanoDrop
ND-1000检测RNA浓度和纯度,使用琼脂糖凝胶电泳检测RNA纯度和完整性。详细的样品QC结果见Arraystar服务报告。
2.cDNA合成
每样本取1.5
μg RNA,使用rtStar™ First-Strand Synthesis Kit (Cat# AS-FS-001, Arraystar)
试剂盒合成cDNA第一链。详细步骤参照Arraystar产品操作手册。
3.Real-time
PCR扩增
将cDNA与 Arraystar SYBR Green qPCR Master Mix (Cat# AS-MR-005-5,
Arraystar)混合,加入至384孔板中,在ABI 7900 PCR仪上进行Real-time
PCR扩增。
4.熔解曲线分析与原始数据导出
PCR扩增完成后,进行熔解曲线分析,使用PCR仪自带软件导出原始数据。出具Raw
Data文件夹,包含原始Ct值、PCR扩增曲线图和熔解曲线图。
PCR芯片数据分析流程
1.PCR芯片数据质控
质控参数:Ct(Blank)>35;
Ct(GDC)>35; Ct(RNA Spike-in)<25; Ct (PPC) <25.
符合上述条件的样本进入下一步分析。
2.数据校正与△Ct值计算
板间校正:使用Ct(PPC)对不同PCR板进行板间校正。
内参校正与△Ct值计算:挑选优质内参,使用内参均值计算△Ct值。
3.差异倍数计算
(2^(-△△Ct))
使用 △△Ct
方法计算不同样品组之间的表达差异倍数。
4.P值计算
对样本组进行t检验,计算P值。
5.其他常规数据分析
散点图分析;火山图分析;TOP20表达上调和下调transcript柱形图分析
6.提供服务报告与数据分析结果
a.Arraystar服务报告(包括RNA样本QC和详细实验数据分析步骤)
b.Excel芯片结果汇总表(包括transcript列表,数据分析结果和各类图表)
c.Raw
Data文件夹 (包含原始数据、扩增曲线图和熔解曲线图)
NuRNA™ Human Epitranscriptomics PCR芯片服务部分结果展示
1.差异表达转录本列表(默认筛选参数:差异倍数>2;P<0.05,客户可指定筛选参数值)

2.散点图 (黑色斜线代表差异倍数为1,红色斜线代表差异倍数为2)

3.火山图(黑色垂线代表差异倍数为1;粉色垂线代表上调或下调倍数为2;蓝色水平线代表P值为0.05)

4.TOP20表达上调transcript柱状图

5.TOP20表达下调transcript柱状图

参考文献
1. Gilbert, W.V., et al. (2016).
Messenger RNA modifications: Form, distribution, and function. Science 352,
1408-1412, PMID:27313037.
2. Iles, M.M., et al. (2013). A variant in FTO
shows association with melanoma risk not due to BMI. Nature genetics 45,
428-432, 432e421, PMID:23455637.
3. Klungland, A., et al. (2016). Reversible
RNA modifications in meiosis and pluripotency. Nature methods 14, 18-22,
PMID:28032624.
4. Lin, S., et al. (2016). The m(6)A Methyltransferase METTL3
Promotes Translation in Human Cancer Cells. Molecular cell 62, 335-345,
PMID:27117702.
5. Zhang, C., et al. (2016). Hypoxia induces the breast cancer
stem cell phenotype by HIF-dependent and ALKBH5-mediated m(6)A-demethylation of
NANOG mRNA. Proceedings of the National Academy of Sciences of the United States
of America 113, E2047-2056, PMID:27001847.
新陈代谢是机体生命活动的基础,影响细胞的各个方面。生长、增殖、分化或凋亡等不同的细胞过程,存在着显著不同的代谢网络[1,4]。细胞内的新陈代谢除了受信号通路等多种机制调控外,也可作为信号分子,或独立调控生物学过程[8]。研究表明,新陈代谢参与调控表观遗传[6,7]、自噬[3,5]、凋亡、坏死[9]等多种细胞生命活动。代谢异常是引起肥胖、糖尿病及肿瘤等多种疾病的重要因素(图1)。肿瘤被认为是一种代谢疾病,代谢改变是癌症发生的显著标志之一。癌细胞进行代谢重编程,以满足肿瘤转化、起始、发展、侵袭与迁移等不同阶段以及不同营养环境下的代谢需求[1,8](图2)。系统地分析疾病的代谢改变,对寻找有效的代谢标记物与疾病治疗靶点非常必需。
Arraystar公司新推出的NuRNA™ Human Central Metabolism PCR
Array,可同时检测373个编码重要代谢酶与代谢物转运蛋白的转录本,是进行代谢研究必不可少的工具。该芯片检测的代谢通路与代谢物转运系统包括:葡萄糖转运蛋白,糖酵解、TCA循环、脂类合成、乳酸合成及转运、糖异生途径、糖原代谢、氨基己糖代谢、磷酸戊糖旁路、一碳单位代谢、谷氨酰胺转运及代谢、氧化还原平衡与GSH合成、脂肪酸氧化、乙酸代谢、核苷酸代谢等。研究不同生物学过程的代谢特征,寻找生理病理状态下的代谢改变,将有助于深入理解细胞生命活动,并为代谢疾病的治疗提供新的思路。

图1.新陈代谢与生理病理过程。

图2.肿瘤代谢示意图
数谱生物(原康成生物)是Arraystar公司中国地区唯一代理商,独家为您提供Arraystar公司PCR芯片一站式技术服务,您只需要的提供保存完好的的组织或者细胞标本,数谱生物的芯片技术服务人员可为您完成全部操作,并提供完整的实验报告。
产品信息:
|
芯片
|
规格
|
描述
|
|
NuRNA™ Human Central Metabolism PCR Array Service
|
384-well/plate
|
检测373个编码重要代谢酶与代谢物转运蛋白的转录本
|
芯片特点:
• 覆盖度高—覆盖了关键代谢途径与重要代谢物转运系统中的酶和蛋白因子
• 注释全面—整合了基因/转录本水平信息与蛋白水平信息,为每个代谢基因提供详细注释
• 转录本水平检测—检测编码Uniprot经典蛋白的转录本
• 严谨性强—所有引物都通过了多样本严格测试
• 快速简易—即用型384孔板规格,只需将cDNA与qPCR Master
Mix混合试剂加入PCR板中,4小时内得到实验结果。cDNA无需预先扩增。
数据库:
|
葡萄糖转运(13):
SLC2A1, SLC2A2, SLC2A3, SLC2A4, SLC2A5, SLC2A6, SLC2A7, SLC2A8,
SLC2A9, SLC2A10, SLC2A11, SLC2A12, SLC2A14
糖酵解(36):
ADPGK, ALDOA, ALDOB, ALDOC, BPGM, ENO1, ENO2, ENO3, GAPDH,
GAPDHS, GCK, GPI, HK1, HK2, HK3, HKDC1, PFKFB1, PFKFB2, PFKFB3-isoform 1,
PFKFB3-isoform 2, PFKFB3-isoform 3, PFKFB3-isoform 4, PFKFB4, PFKL, PFKM, PFKP,
PGAM1, PGAM2, PGAM4, PGK1, PGK2, PKL, PKM1, PKM2, PKR, TPI1
乳酸合成及转运(18):
LDHA, LDHB, LDHC, LDHAL6A, LDHAL6B, UEVLD, SLC16A1, SLC16A2,
SLC16A3, SLC16A4, SLC16A5, SLC16A6, SLC16A7, SLC16A8, SLC16A9, SLC16A10,
SLC16A11, SLC16A12
糖异生(13):
BCAT1, BCAT2, FBP1, FBP2, G6PC, G6PC2, G6PC3, GPT, GPT2, LDHD,
PC, PCK1, PCK2
糖原代谢(6):
GBE1, GYS1, GYS2, PGM1, PGM2, UGP2
氨基己糖代谢(6):
GFPT1, GFPT2, GNPNAT1, PGM3, UAP1, UAP1L1
PPP途径(15):
G6PD, H6PD, PGD, PGLS, PRPS1, PRPS1L1, PRPS2, RBKS, RPE, RPEL1,
RPIA, TALDO1, TKT, TKTL1, TKTL2
脂类合成(31):
ACACA, ACACB, ACAT1, ACAT2, ACLY, ACSBG1, ACSBG2, ACSL1, ACSL3,
ACSL4, ACSL5, ACSL6, ACSM1, ACSM2A, ACSM2B, ACSM3, ACSM4, ACSM5, FADS1, FADS2,
FASN, GPD1, GPD1L, HMGCR, HMGCS1, HMGCS2, MLYCD, SCD, SCD5, SLC25A1,
SLC27A2
一碳单位代谢(28):
AHCY, AHCYL1, AHCYL2, AMT, BHMT, DHFR, DHFRL1, DLD, DNMT1,
DNMT3A, DNMT3B, DNMT3L, GCSH, GLDC, MAT1A, MAT2A, MAT2B, MTHFD1, MTHFD1L,
MTHFD2, MTHFD2L, MTHFR, MTR, PHGDH, PSAT1, PSPH, SHMT1, SHMT2
TCA循环(42):
ACO1, ACO2, D2HGDH, DHTKD1, DLAT, DLD, DLST, FH, IDH1, IDH2,
IDH3A, IDH3B, IDH3G, L2HGDH, MDH1, MDH1B, MDH2, OGDH, OGDHL, PDHA1, PDHA2, PDHB,
PDHX, PDK1, PDK2, PDK3, PDK4, PDP1, PDP2, PDPR, SDHA, SDHAF1, SDHAF2, SDHAF3,
SDHAF4, SDHB, SDHC, SDHD, SUCLA2, SUCLG1, SUCLG2, UEVLD
谷氨酰胺转运及降解(18):
GLS2, GLS-isoform 1, GLS-isoform 2, GLS-isoform 3, GLUD1, GLUD2,
GOT1, GOT2, SLC1A1, SLC1A2, SLC1A3, SLC1A4, SLC1A5, SLC1A6, SLC38A1, SLC38A3,
SLC38A5, SLC38A7
氧化还原平衡(16):
CBS, CTH, G6PD, GCLC, GCLM, GSR, GSS, IDH1, IDH2, ME1, ME2, ME3,
MTHFD1, NNT, PGD, SLC7A11
GSH合成(7):
CBS, CTH, GCLC, GCLM, GSR, GSS, SLC7A11
脂肪酸氧化(51):
AADAC, ABHD12, ABHD6, ACAA1, ACAA2, ACAD10, ACAD11, ACAD8,
ACAD9, ACADL, ACADM, ACADS, ACADSB, ACADVL, ALDH1B1, ALDH2, ALDH3A2, ALDH7A1,
ALDH9A1, CEL, CPT1A, CPT1B, CPT1C, CPT2, ECH1, ECHS1, ECI1, ECI2, EHHADH, ETFA,
ETFB, HADH, HADHA, HADHB, HSD17B10, HSD17B4, LIPC, LIPE, LIPF, LIPG, MGLL,
PAFAH1B1, PAFAH1B2, PAFAH1B3, PNLIP, PNLIPRP1, PNLIPRP2, PNLIPRP3, PNPLA2,
PNPLA3, SCP2
乙酸代谢(4):
ACOT12, ACSS1, ACSS2, ACSS3
核苷酸代谢(83):
ADA, ADCY1, ADCY10, ADCY2, ADCY3, ADCY4, ADCY5, ADCY6, ADCY7,
ADCY9, ADSL, ADSS, ADSSL1, AK1, AK2, AK3, AK4, AK5, AK6, AK7, AK8, AK9, AMPD1,
AMPD2, AMPD3, APRT, ATIC, CAD, CANT1, CDA, CECR1, CMPK1, CMPK2, CTPS1, DCK,
DCTD, DHODH, DTYMK, DUT, GART, GDA, GMPS, GUCA1A, GUCA1B, GUCA1C, GUCA2A,
GUCA2B, GUCD1, GUCY1A2, GUCY1A3, GUCY1B3, GUCY2C, GUCY2D, GUCY2F, GUK1, HPRT1,
IMPDH1, IMPDH2, NME1, NME2, NME3, NME4, NME6, NME7, NT5C2, PAICS, PDE10A, PDE4D,
PFAS, PNP, PPAT, RRM1, RRM2, RRM2B, TK1, TK2, TYMP, TYMS, UCKL1, UMPS, UPP1,
UPP2, XDH
|
PCR芯片实验流程
1.RNA抽提与质量检测
进行RNA常规抽提,使用NanoDrop
ND-1000检测RNA浓度和纯度,使用琼脂糖凝胶电泳检测RNA纯度和完整性。详细的样品QC结果见Arraystar服务报告。
2.cDNA合成
每样本取1.5
μg RNA,使用rtStar™ First-Strand Synthesis Kit (Cat# AS-FS-001, Arraystar)
试剂盒合成cDNA第一链。详细步骤参照Arraystar产品操作手册。
3.Real-time
PCR扩增
将cDNA与 Arraystar SYBR Green qPCR Master Mix (Cat# AS-MR-005-5,
Arraystar)混合,加入至384孔板中,在ABI 7900 PCR仪上进行Real-time
PCR扩增。
4.熔解曲线分析与原始数据导出
PCR扩增完成后,进行熔解曲线分析,使用PCR仪自带软件导出原始数据。出具Raw
Data文件夹,包含原始Ct值、PCR扩增曲线图和熔解曲线图。
PCR芯片数据分析流程
1.PCR芯片数据质控
质控参数:Ct(Blank)>35;
Ct(GDC)>35; Ct(RNA Spike-in)<25; Ct (PPC) <25.
符合上述条件的样本进入下一步分析。
2.数据校正与△Ct值计算
板间校正:使用Ct(PPC)对不同PCR板进行板间校正。
内参校正与△Ct值计算:挑选优质内参,使用内参均值计算△Ct值。
3.差异倍数计算
(2^(-△△Ct))
使用 △△Ct
方法计算不同样品组之间的表达差异倍数。
4.P值计算
对样本组进行t检验,计算P值。
5.其他常规数据分析
散点图分析;火山图分析;TOP20表达上调和下调transcript柱形图分析
6.提供服务报告与数据分析结果
a.Arraystar服务报告(包括RNA样本QC和详细实验数据分析步骤)
b.Excel芯片结果汇总表(包括transcript列表,数据分析结果和各类图表)
c.Raw
Data文件夹 (包含原始数据、扩增曲线图和熔解曲线图)
NuRNA™ Human Central Metabolism PCR芯片服务部分结果展示
1.差异表达转录本列表(默认筛选参数:差异倍数>2;P<0.05,客户可指定筛选参数值)

2.散点图 (黑色斜线代表差异倍数为1,红色斜线代表差异倍数为2)

3.火山图(黑色垂线代表差异倍数为1;粉色垂线代表上调或下调倍数为2;蓝色水平线代表P值为0.05)

4.TOP20表达上调transcript柱状图

5.TOP20表达下调transcript柱状图

参考文献
1. Agathocleous, M., and
Harris, W.A. (2013). Metabolism in physiological cell proliferation and
differentiation. Trends in cell biology 23, 484-492, PMID:23756093.
2. Enns,
G. Metabolic disease (Encyclopædia Britannica, inc.).
3. Galluzzi, L., et al.
(2014). Metabolic control of autophagy. Cell 159, 1263-1276,
PMID:25480292.
4. Green, D.R., et al. (2014). Cell biology. Metabolic control
of cell death. Science 345, 1250256, PMID:25237106.
5. Kaur, J., and Debnath,
J. (2015). Autophagy at the crossroads of catabolism and anabolism. Nature
reviews Molecular cell biology 16, 461-472, PMID:26177004.
6. Kinnaird, A.,
et al. (2016). Metabolic control of epigenetics in cancer. Nature reviews Cancer
16, 694-707, PMID:27634449.
7. Lu, C., and Thompson, C.B. (2012). Metabolic
regulation of epigenetics. Cell metabolism 16, 9-17,
PMID:22768835.
8. Metallo, C.M., and Vander Heiden, M.G. (2013).
Understanding metabolic regulation and its influence on cell physiology.
Molecular cell 49, 388-398, PMID:23395269.
9. Vanden Berghe, T., et al.
(2014). Regulated necrosis: the expanding network of non-apoptotic cell death
pathways. Nature reviews Molecular cell biology 15, 135-147, PMID:24452471.

