

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
相关服务
N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 RIP-seq/RIP 芯片相关产品
相关资源
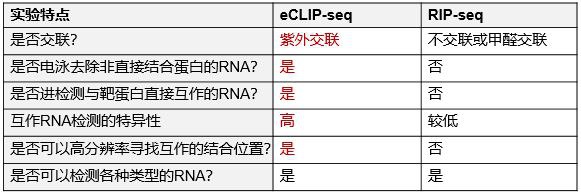
RNA结合蛋白(RBP)作为调控基因表达的关键因子,通过与靶RNA分子相互作用在细胞生理过程中发挥关键作用,控制着RNA在细胞内的加工、运输和翻译[1]。这些调控功能对正常人体生理至关重要,RBP功能缺陷与多种遗传性和获得性疾病相关,如神经退行性疾病、自身免疫缺陷和癌症[2,3]。为探索RBP影响RNA加工的机制,RNA免疫共沉淀(RIP)和CLIP等能全面鉴定RBP相互作用RNA底物的技术被广泛应用。然而RIP-seq不能鉴定RBP在RNA上的结合位置;经典iCLIP-seq技术能鉴定RNA的结合位置,但存在失败率高、有效文库复杂度低、重复性差等问题。
eCLIP-seq(enhanced UV crosslinking and immunoprecipitation增强型紫外交联免疫共沉淀测序)是在经典iCLIP-seq技术基础上优化而成的具有高效的“蛋白-RNA”相互作用研究手段[4]。该技术首先通过紫外交联固定细胞内特定蛋白与其结合的RNA的相互作用,然后利用特异性抗体富集目标RNA结合蛋白(RBP)及其结合的RNA片段,最终通过高通量测序实现在全转录组范围内精确绘制RBP结合图谱。
作为研究转录后调控机制的核心工具,eCLIP-seq能够精准揭示RBP在RNA剪接、稳定性调控、翻译控制等过程中的分子机制,为深入理解基因表达调控网络提供了关键技术支撑。
4、可重复性高:标准化且稳健的实验流程,大幅降低技术难度与实验失败率,支持大规模、可重复的RBP结合图谱绘制。
eCLIP-seq相对于RIP-seq的优势:

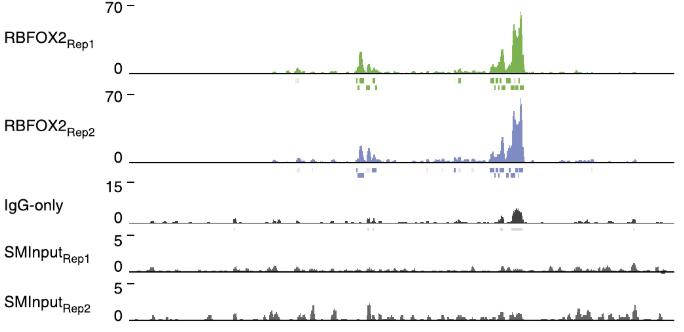
2.RBP蛋白eCLIP富集区可视化峰图
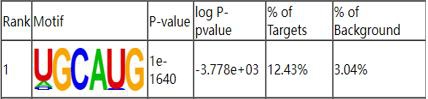
3.RBP蛋白结合序列motif分析
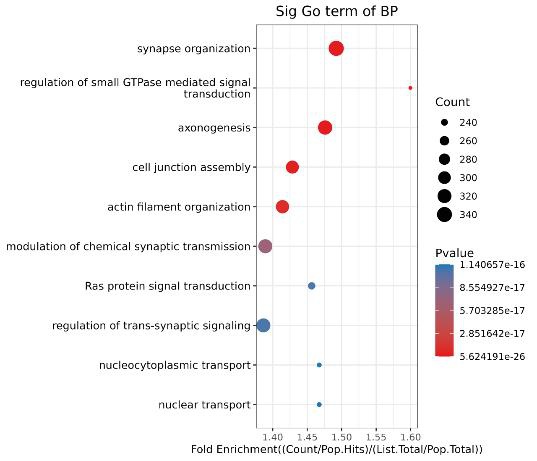
4.RBP蛋白结合靶标RNA GO/pathway分析
PRMT5(蛋白精氨酸甲基转移酶5)介导的FXR1精氨酸甲基化修饰对其RNA结合活性至关重要,可增强其与mRNA的结合能力,并参与癌细胞生长与增殖过程。FXR1优先结合含有G4结构的mRNA靶标, FXR1精氨酸修饰位点突变或RNA G4结构破坏会抑制FXR1与靶RNA结合。PRMT5抑制导致FXR1蛋白不稳定,从而下调其靶标mRNA的表达,抑制肿瘤细胞的生长和增殖。
本研究利用eCLIP-seq技术,精准绘制了FXR1在癌细胞全转录组范围内的RNA结合图谱[5]。结果表明,FXR1与多个富含G4结构的mRNA靶标(包括AHNAK、MAP1B、AHNAK2、HUWE1、DYNC1H1和UBR4)结合,并调控这些mRNA在癌细胞中的表达。因此,FXR1的结构特性及其对G4 RNA的亲和性,为揭示其在口腔癌细胞中的作用机制提供了新见解,并提示FXR1-PRMT5界面可能是一个潜在的治疗靶点。
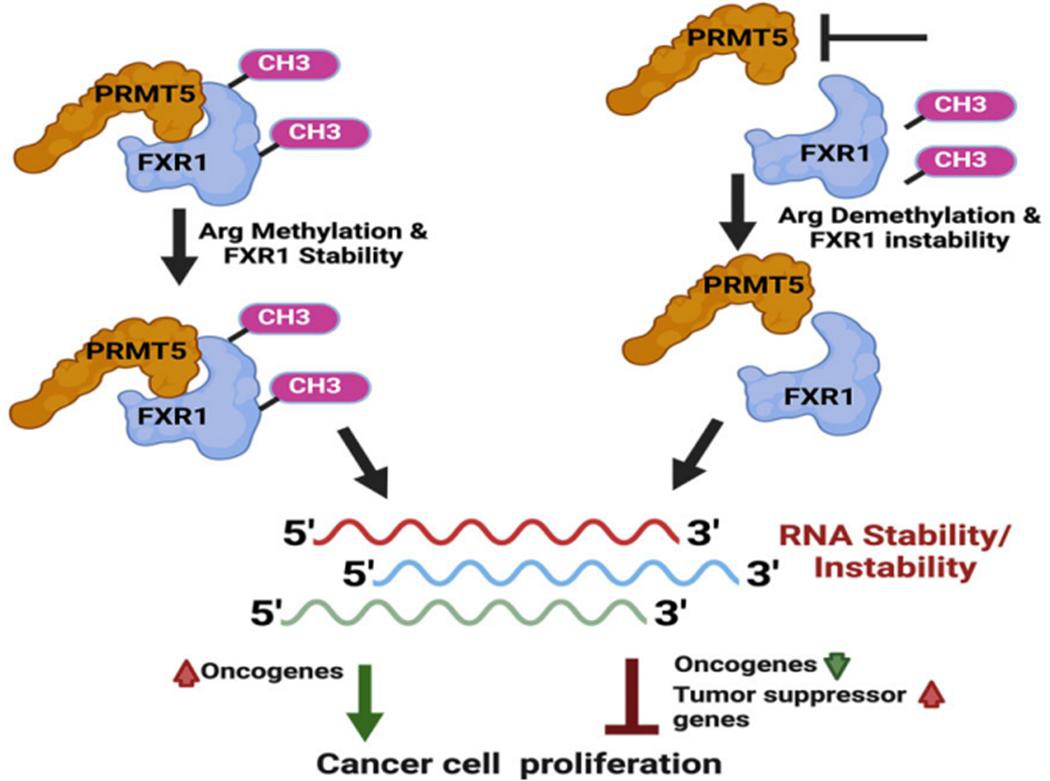
图1. FXR1 eCLIP-seq实验证明口腔癌中PRMT5-精氨酸甲基化修饰FXR1结合靶mRNA影响原癌和抑癌基因RNA稳定性进而调控肿瘤细胞增殖[5]。
参考文献
1.Gerstberger, S., Hafner, M. & Tuschl, T. A census of human RNA-binding proteins. Nat. Rev. Genet. 5, 829–845 (2014)
2.Castello, A., Fischer, B., Hentze, M.W. & Preiss, T. RNA-binding proteins in Mendelian disease. Trends. Genet. 29, 318–327 (2013).
3.Nussbacher, J.K., Batra, R., Lagier-Tourenne, C. & Yeo, G.W. RNA-binding proteins in neurodegeneration: Seq and you shall receive. Trends Neurosci. 38, 226–236 (2015).
4.Van Nostrand EL, Pratt GA, Shishkin AA, et al. Robust transcriptome-wide discovery of RNA-binding protein binding sites with enhanced CLIP (eCLIP). Nat Methods. 2016;13(6):508-514.
5.Vijayakumar A, Majumder M, Yin S, et al. PRMT5-mediated arginine methylation of FXR1 is essential for RNA binding in cancer cells. Nucleic Acids Res. 2024;52(12):7225-7244.

