|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
相关服务
snoRNA测序相关产品
相关资源
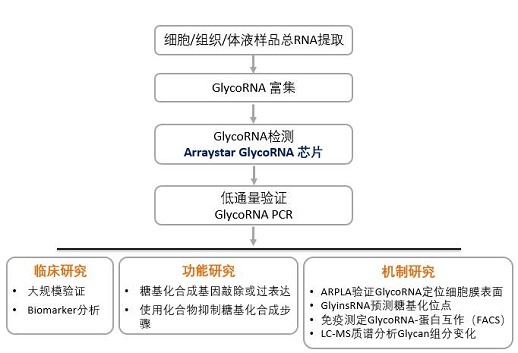
芯片更适用于低丰度RNA分析 为什么要研究GlycoRNA? 什么是糖基化RNA(GlycoRNA)? 如何研究GlycoRNAArraystar GlycoRNA芯片结合了GlycoRNA的生物化学富集和RNA定量分析方法,利用这两种方法在特异性、灵敏度和准确性方面的优势,检测GlycoRNA的表达。
芯片涵盖了广泛的糖基化小RNA类别,包括Y-RNAs/Y-RNA片段、tRNAs、tRF&tiRNAs、pre-miRNAs、miRNAs、snRNAs/snRNA片段、snoRNAs/snoRNA片段和rRNAs/rRNA片段。
利用这种新的方法,研究者可以获得全面的GlycoRNA表达细节,以发现和理解这类在基因调控、细胞功能和人类疾病中发挥功能的新RNA分子。
Arraystar GlycoRNA芯片的一些关键优势包括:
GlycoRNA 的全面检测Arraystar芯片的RNA直接标记方法,避免了RNA修饰和空间折叠对cDNA反转的阻断,使得全面检测tRF&tiRNA和tRNA成为可能,从而实现对glycoRNA的全面检测,包括Y-RNAs/Y-RNA片段、tRNAs、tRFs&tiRNAs、pre-miRNAs、miRNAs、snRNAs/snRNA片段、snoRNAs/snoRNA片段以及rRNAs/rRNA片段。由于这些小RNA具有独特的生化特性,通过测序进行GlycoRNA检测需要分别进行不同类别的小RNA的专门测序,而Arraystar GlycoRNA芯片可以在一次实验中同时检测以上各类小RNA。
适用于所有样品类型
Arraystar GlycoRNA芯片的 glycoRNA 捕获方法可检测各类细胞及临床样品类型的 glycoRNA:活细胞或非活细胞、组织、生物体液。
新型小GlycoRNA的发现
Arraystar GlycoRNA芯片包含了基因组中的所有的小RNA种类,无论其是否已知为糖基化。如果在微阵列上检测到的GlycoRNA之前未被报道,即可将其识别为新型GlycoRNA。因此无需担心预先设计的芯片是否具有与测序相同的发现新型GlycoRNA的能力。
Arraystar GlycoRNA 芯片
|
探针设计 |
带有5’发卡帽和3’间隔区的小RNA特异性序列 |
|||
|
探针结合位点 |
miRNA/5'tRF&tiRNA: 3' 区域
tRNA: 成熟tRNA的反密码子环序列 |
|||
|
探针特异性 |
小RNA特异性 |
|||
|
芯片规格 |
8x15K |
|||
|
小RNA覆盖情况 |
来源 |
|||
|
|
Human |
Mouse |
Rat |
|
|
探针总数 |
7,646 |
7,420 |
5,312 |
|
|
YsRNA (Y-RNA来源小RNA) |
10 |
5 |
7 |
人: 文献来源 小鼠和大鼠: 预测 |
|
snsRNA (snRNA来源小RNA) |
4 |
35 |
35 |
|
|
sdRNA (snoRNA来源小RNA) |
289 |
1,334 |
1,464 |
|
|
rRF (rRNA来源小RNA) |
210 |
479 |
280 |
人: MINTbase(V1) 大鼠和小鼠: 预测 |
|
miRNA |
2,627 |
1,949 |
749 |
miRBase(v22) |
|
tRF&tiRNA (tRNA来源小RNA) |
1,432 |
910 |
653 |
tRFdb, MINTbase, GtRNADb (v18.1, 2019.08) 公开发表的高分文献至2019 |
|
pre-miRNA |
1,745 |
1,122 |
448 |
miRBase(v22) |
|
mature-tRNA |
338 |
267 |
195 |
GtRNAdb (v18.1, 2019.08) ENSEMBL (v99) |
|
snoRNA |
955 |
1,297 |
1,464 |
ENSEMBL (v99) |
|
Y-RNA |
4 |
2 |
3 |
RNAcentral (V24) RefSeq (2024.08) |
|
snRNA |
27 |
15 |
9 |
|
|
rRNA |
5 |
5 |
5 |
|
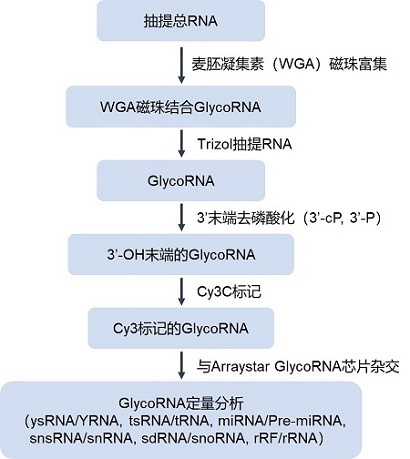
Table1 差异GlycoRNA表达列表
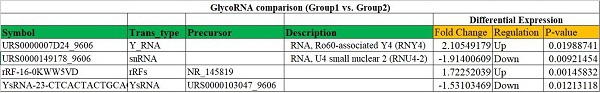
Symbol: GlycoRNA的名称。
Trans_type:GlycoRNA的转录本类型。
Precursor:GlycoRNA的前体RNA。
Description:GlycoRNA的注释信息。
Fold Change:表示实验组与对照组之间GlycoRNA的变化倍数。
Regulation:两组比较的上调(up)或者下调(down)。
P-value:评估两组GlycoRNA表达水平的差异是否具有统计学显著性的p值。