

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
microRNA是一类大小为21-23nt的非编码小RNA分子,通过沉默或降解其靶点mRNA分子调控基因表达。大量研究表明,microRNA在正常发育过程以及疾病发生过程中具有重要的作用,在诊断和治疗领域具有光明的应用前景。然而目前研究microRNA的方法主要是通过实时定量PCR以及基因芯片技术,这些方法主要关注microRNA的表达与定量,并仅局限于研究那些序列信息或二级茎环结构信息已知的microRNA,无法寻找和发现新的microRNA分子。基于高通量测序平台的microRNA测序技术突破了目前研究技术手段上的局限性,使研究人员能够直接对样本中指定大小的所有microRNA分子进行高通量测序,在无需任何序列信息的前提下研究microRNA的表达谱并在此基础上发现和鉴定新的microRNA分子,并提供了更加灵活和深入的研究分析方法,这是传统的研究方法所无法比拟的。
Aksomics(原康成生物)为您提供microRNA测序服务,您只需提供保存完好的RNA,细胞或组织样品,Aksomics的技术人员即可为您完成全部实验操作,包括文库构建、高通量测序和数据分析,并提供直接可用的数据报告及Paper级结果图表。我们的标准报告涵盖其他服务商没有或者仅作为可选项的高级分析项目,助力您深入microRNA研究。
1.一站式解决方案
您只需要提供保存完好的组织或细胞样本,Aksomics的测序技术服务人员就可为您完成全部实验操作,并提供完整的实验报告。
2. 适合样品量少的客户
测序实验经过优化,从总RNA开始实验,不需要对MicroRNA进行富集和反复的纯化,因此对于样品量少的客户更加适合, 1ug就可以进行实验。
3. 节省实验的步骤,减少实验误差
传统方法是先对microRNA进行富集,再两边加接头,而且每一步都要纯化回收,很容易产生实验误差,而我们只需要对总RNA就能完成实验,减少实验误差。
Aksomics拥有强大的生物信息学团队,能够根据不同客户的要求进行标准和深入的数据分析,提供能直接用于文章发表的图表和数据。
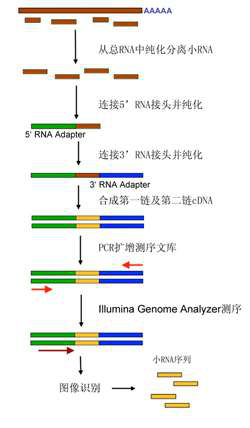
1.样品RNA准备
2.测序文库构建
PAGE胶纯化特定大小的microRNA分子
连接5’RNA接头
连接3’RNA接头
逆转录反应获得cDNA测序文库
高保真聚合酶扩增构建的测序文库
3. DNA成簇(Cluster)扩增
4.高通量测序
5.数据分析
原始数据读取
使用公用microRNA数据库注释已知microRNA
深层次数据分析
6.提供实验报告
原始数据报告(Fasta-Q格式),包含所有测序序列信息,碱基读取质量评估
基本数据分析报告(Excel表格),包含有效序列的序列信息、与参考基因组比对后的注释信息等
高级数据分析(应客户要求定制),如使用已知数据库注释已知microRNA及分析不同样本间表达水平差异,使用已知数据库鉴别rRNA,tRNA,snRNA,snoRNA并预测新的microRNA,预测microRNA的靶点基因。
miRNA测序基本数据分析结果展示
1.测序数据统计
|
Sample Name |
Clean Reads* |
Adaptor Trimmed** |
microRNA Reads*** |
|
Z381-FCort |
7,237,456 |
7,000,106 |
5,906,193 |
|
Z381-PCort |
7,483,217 |
7,199,637 |
5,743,606 |
*图释:*: 通过高通量测序数据质控的高质量read的数目;**:去掉接头序列后长度大于或等于15nt的read数目;***:能够与新版miRBase数据库中的pre-microRNA序列匹配(至多允许一个碱基的错配)的read数目
2.microRNA表达谱数据
microRNA测序常见的用途就是对不同样品中microRNA的表达进行定量分析和比较。但是由于原始read数与测序数据量相关,直接用原始read值衡量特定microRNA的表达是不合适的。因此,我们对microRNA的表达量进行了标准化:将原始的read值转化为TPM(在每兆可与miRBase数据库中的pre-microRNA匹配的reads中,特定microRNA转录本所占的read数目)。TPM值可以用于衡量特定microRNA在样品中的表达丰度,以及对不同样品间差异表达的microRNA的分析。

*图释:microRNA表达谱数据示例
3.microRNA差异表达数据
根据标准化后的microRNA表达值(TPM),我们会计算出每个microRNA在不同样品间(如处理组 vs 对照组)的表达变化(fold change),并通过ttest计算样品间microRNA表达量变化的统计学显著性。那些表达变化倍数在1.5倍以上,p值小于0.05的microRNA被挑选出来,并定义为显著性差异表达的microRNA。针对多重比较,p值被校正为FDR。客户可以根据倍数变化,p值,FDR等参数对差异表达的microRNA进行排序和筛选。
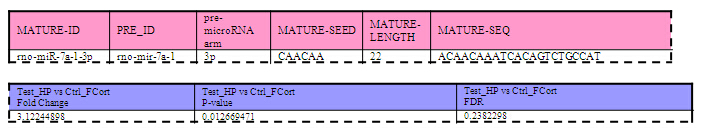
*图释:样品间差异表达的microRNA数据示例
4.差异表达microRNA的聚类图
层次聚类是一种最常见的用于分析表达数据的聚类方法。它可以根据样品中基因的表达水平将样品自动的分组,可以让客户从整体上评估样品间的基因表达差异,以及样品间的关系。左侧的树状图可以反映样品间基因表达模式的关系。
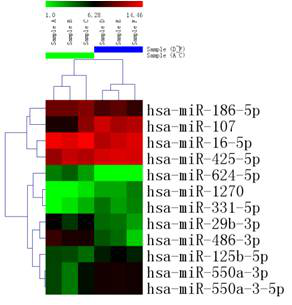
*图释:不同样品组之间差异表达microRNA的聚类示例
5.差异表达microRNA的火山图
火山图可以对不同样品组之间差异表达的microRNA进行图形化的展示。横轴代表处理组和对照组之间microRNA表达倍数的变化(log2转化),纵轴代表相应的p值(-log10转化)。火山图可以直观的展示microRNA在样品组间的倍数变化与相应的统计学显著性之间的关系(图5)。纵向的绿线分别对应1.5倍的上调(右侧)和下调(左侧),绿色平行线对应0.05的p值。下面火山图中的红色点代表在不同样品组中具有统计学显著性的差异microRNA。
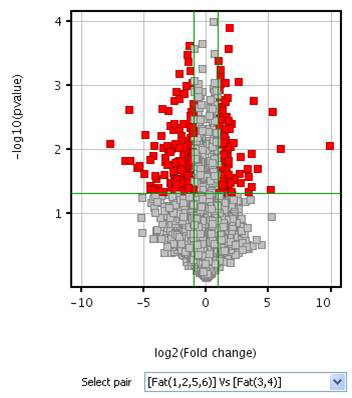
*图释:火山图示例
6.新的microRNA预测
通过microRNA测序可以预测新的microRNA。新microRNA能通过miRDeep软件进行预测。根据microRNA成熟过程中,dicer酶对microRNA前体的加工原理,miRDeep构建了一种简单的预测模型。通过这个模型,它可以根据高通量测序结果准确的预测样品中的新microRNA。
7.差异表达microRNA的靶基因预测及功能富集分析
它们通过诱导mRNA降解或转录抑制在基因表达调控中起着一种重要的作用。每个microRNA通过部分序列互补的方式结合到基因的3'UTR,平均靶向几百个基因。Aksomics microRNA靶基因预测可以分析top10差异miRNA(上下调各10个)的潜在靶基因,并构建microRNA-靶基因调控网络,以推断这些microRNA的功能。
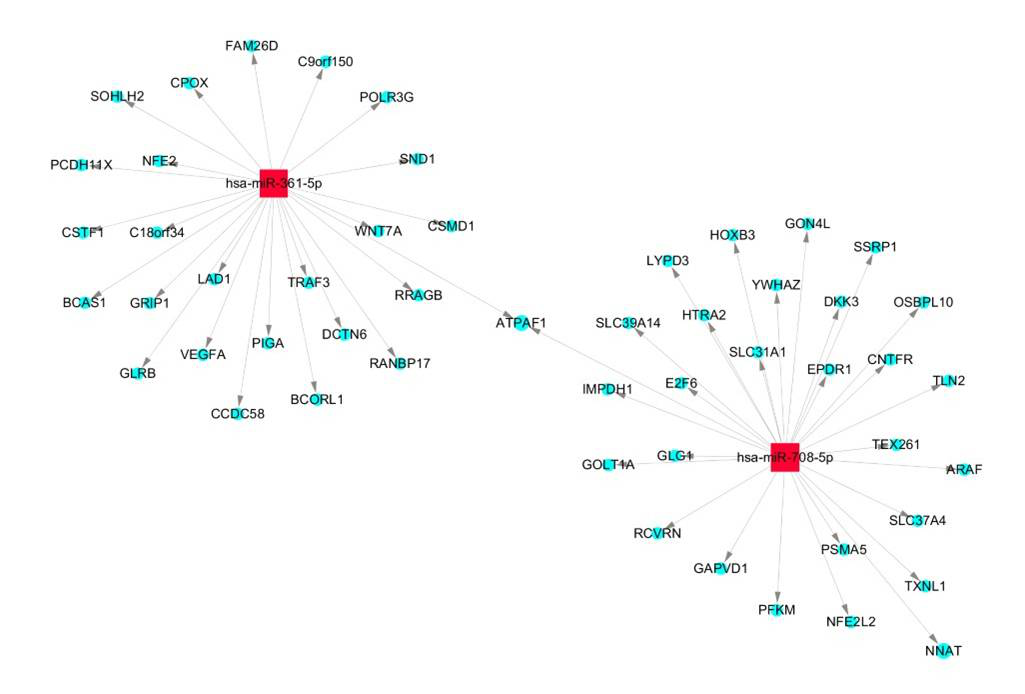
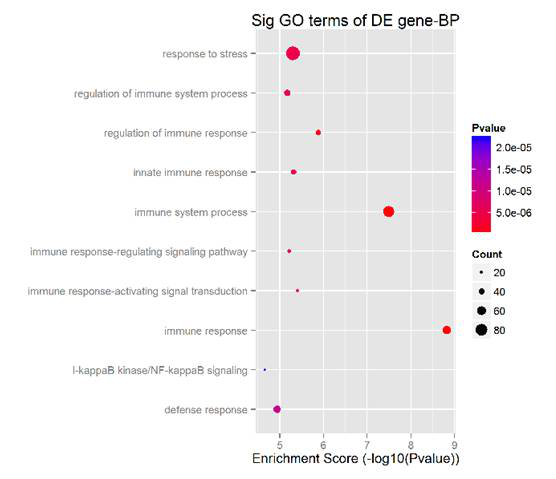
*图释:上图:通过靶基因预测构建的差异表达microRNA-靶基因网络(红色矩形:microRNA;蓝色圆形:靶基因);下图:预测得到的靶基因的GO富集结果。根据排名前10条的GO-term来展示结果,点颜色代表P-value,点大小代表靶基因和该GO条目中均存在的基因个数。
猪传染性胃肠病毒感染引起的microRNA转录组应答(The Porcine MicroRNA Transcriptome Response to Transmissible Gastroenteritis Virus Infection. PLoS One, 2015)
TGEV(猪传染性胃肠病毒)是一种具有高传染性、具有多种传播途径并在全球广泛分布的病毒,它所引起的猪胃肠系统疾病每年都会给养猪产业带来巨大的经济损失。为了揭示病毒感染过程中的的microRNA分子调控机制,作者对TGEV感染实验组和对照细胞进行了microRNA高通量测序,测序数据比对注释结果发现了其中存在284个已知microRNA成熟体和241个novel microRNA。随后的组间比较结果显示其中已知microRNA中分别出现了15个上调和44下调。从中挑选了10个microRNA进行qPCR验证,结果证实测序数据与qPCR结果相符。进一步通过生物信息学手段进行分析发现其中4个差异表达microRNA可能会靶作用于TGEV基因组;而对其中包括miR-146b和miR-155-5p在内的几个差异表达microRNA的靶基因进行功能富集分析,结果显示这些microRNA可能会通过作用于相应的靶基因来参与对代谢加工、细胞免疫反应、T细胞发育、细胞凋亡和细胞因子产生等过程的调控。该课题研究成果提供了一种全新的应对策略方向来治疗和预防TGEV感染。
结果展示

*图释:microRNA靶位点预测及靶基因功能富集。上图:ssc-miR-28-3p、ssc-miR-126-5p、ssc-miR-306-5p和ssc-miR-2411在TGEV基因组上预测的结合位点;下图:差异microRNA预测靶基因的GO注释结果。
→ EBV-miR-BART10-3p and EBV-miR-BART22 promote metastasis of EBV-associated gastric carcinoma by activating the canonical Wnt signaling pathway . Cellular Oncology . 2020
→ A miR-146a-5p/TRAF6/NF-kB p65 axis regulates pancreatic cancer chemoresistance: functional validation and clinical significance . Theranostics . 2020
→ MiR-19a suppress apoptosis of myocardial cells in rats with myocardial ischemia/reperfusion through PTEN/Akt/P-Akt signaling pathway . European Review for Medical and Pharmacological Sciences . 2020
→ Effects of miR-150-5p on cerebral infarction rats by regulating the Wnt signaling pathway via p53 . European Review for Medical and Pharmacological Sciences . 2020
→ Mesenchymal stem cell-derived exosomes protect beta cells against hypoxia-induced apoptosis via miR-21 by alleviating ER stress and inhibiting p38 MAPK phosphorylation . Stem Cell Research & Therapy . 2020
→ Regulation of cancer stem cell properties, angiogenesis, and vasculogenic mimicry by miR-450a-5p/SOX2 axis in colorectal cancer . Cell Death & Disease . 2020
→ MiR-214 Attenuates the Osteogenic Effects of Mechanical Loading on Osteoblasts . International journal of sports medicine . 2019
→ D2 Receptor-Mediated miRNA-143 Expression is Associated with the Effects of Antipsychotic Drugs on Phencyclidine-Induced Schizophrenia-Related Locomotor Hyperactivity and with Neuregulin-1 Expression in Mice . Neuropharmacology . 2019
→ miR-412-5p targets Xpo1 to regulate angiogenesis in hemorrhoid tissue . Gene . 2019
→ ZBTB7A, a miR-663a target gene, protects osteosarcoma from endoplasmic reticulum stress-induced apoptosis by suppressing LncRNA GAS5 expression . Cancer Letters . 2019
→ miRNA-3473b contributes to neuroinflammation following cerebral ischemia. Cell death & disease. 2018
→ Circulating microRNA signature for the diagnosis of childhood dilated cardiomyopathy. Scientific reports. 2018

