

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
相关服务
AGO APP-seq /AGO APP芯片相关产品
相关资源
smallRNA(包括miRNA和tRF&tiRNA等)是小型非编码RNA分子,其通过序列互补配对引导Argonaute(AGO)蛋白与mRNA 3‘UTR或CDS区结合,介导mRNA降解或翻译抑制[1,2]。尽管smallRNA在多种疾病及生物过程中具有重要作用,但关于AGO与smallRNA靶向作用的调控规则仍未被完全解析,精确鉴定smallRNA靶标仍面临重大挑战。比如利用生信预测small RNA靶基因通常具有以下局限性,其预测原理是通过分析smallRNA 2-8位种子区是否能与mRNA 3‘UTR互补配对[1]。一个smallRNA可以预测到成百上千个靶基因,预测结果太多导致假阳性率高,影响靶点挑选和验证效果。经典生信预测也难以分析非常规结合模式或CDS区域结合的靶点。
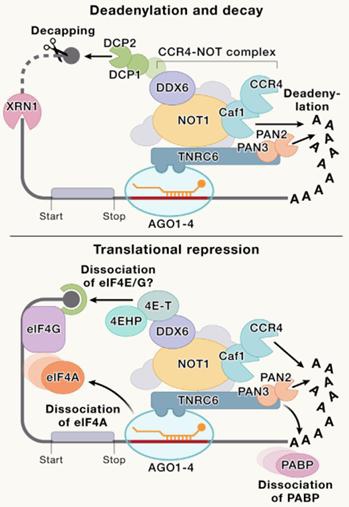
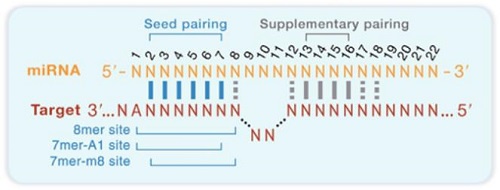
通过CLEAR-CLIP-seq技术鉴定smallRNA-靶标RNA嵌合体。
1)组织/细胞经过紫外线照射诱导small RNA与AGO蛋白交联;
2)组织裂解、DNA酶/RNA酶处理、AGO IP纯化AGO-small RNA-靶基因RNA复合物;
3)对RNA进行5’端磷酸化处理,RNA连接酶连接,产生small RNA-靶基因嵌合体;
4)3’端去磷酸化、加接头、标记、构建文库;
5)高通量测序鉴定small RNA及其结合的靶标RNA嵌合体序列。
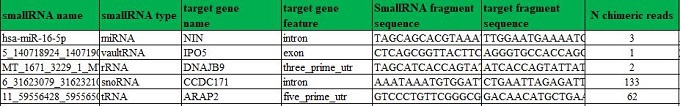
1.smallRNA-靶标RNA结合序列表格
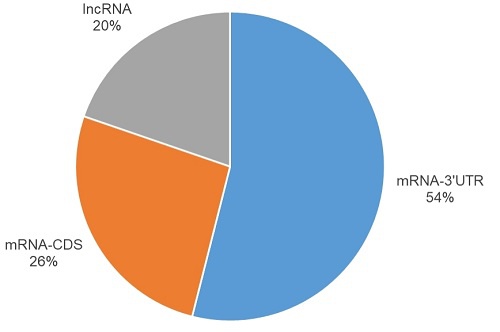
2.smallRNA结合靶点分类统计饼形图
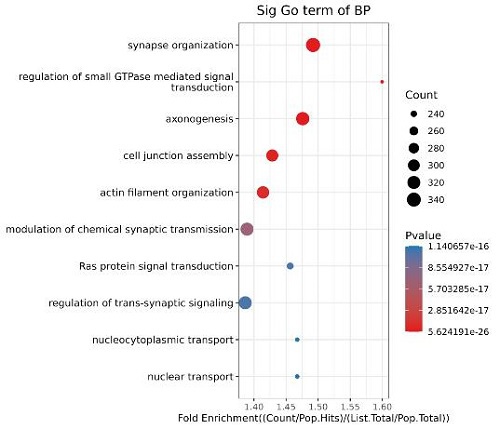
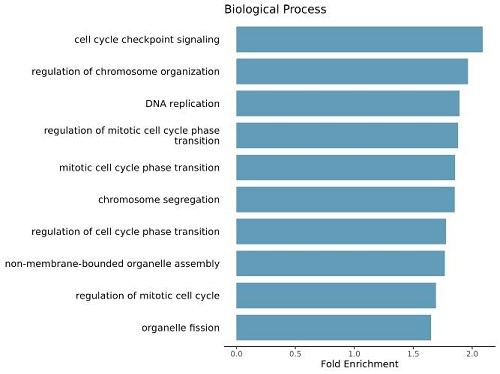
3.smallRNA结合靶标RNA GO/pathway分析
1.Chen LL, Kim VN. Small and long non-coding RNAs: Past, present, and future. Cell. 2024;187(23):6451-6485.
2.Jouravleva K, Zamore PD. A guide to the biogenesis and functions of endogenous small non-coding RNAs in animals. Nat Rev Mol Cell Biol. 2025;26(5):347-370.
3.Moore MJ, Scheel TK, Luna JM, et al. miRNA-target chimeras reveal miRNA 3'-end pairing as a major determinant of Argonaute target specificity. Nat Commun. 2015;6:8864.
4.Helwak A, Kudla G, Dudnakova T, Tollervey D. Mapping the human miRNA interactome by CLASH reveals frequent noncanonical binding. Cell. 2013;153(3):654-665.

