

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
相关服务
ChIP-qPCR N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 基因下游RNA(DoG RNA)芯片 RIME-MS相关产品
相关资源
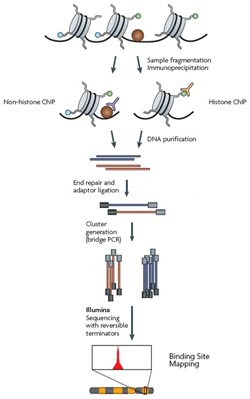
染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP)是研究体内蛋白质与DNA相互作用的有力工具,利用该技术不仅可以检测体内反式因子与DNA的动态作用,还可以用来研究组蛋白的各种共价修饰以及转录因子与基因表达的关系。
结合高通量的新一代测序技术,通过对染色质免疫共沉淀(ChIP)富集得到的DNA片断进行大规模测序,研究人员可获得数百万条序列标签,并能把所关注的蛋白的DNA结合位点精确定位到基因组上,从而获得全基因组范围内组蛋白各种修饰状态、转录因子结合位点的高分辨率分布图。染色质免疫共沉淀测序(ChIP-Seq)是继ChIP-chip之后,蛋白/核酸相互作用研究领域的又一技术突破。高质量、高通量、低成本的数据产出,为表观遗传组学研究提供了一套全新高效的技术工具。
染色质免疫共沉淀测序应用领域:
组蛋白修饰,转录因子,RNA polymerase II,转录因子等。
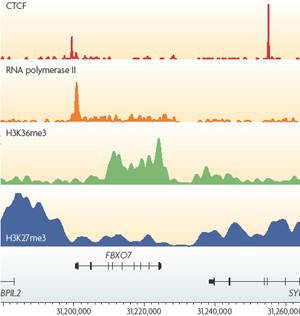
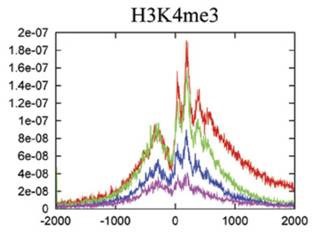
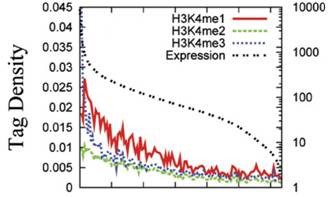
上图:染色质免疫共沉淀技术已应用于检测组蛋白各种共价修饰、CTCF、RNA polymerase II以及各种转录因子等DNA结合蛋白在基因组上的精确定位,为表观遗传组学与基因转录调控机制研究提供了又一有力工具。(摘自Park P.J, 2009,Nature Reviews Genetics)
● 一站式服务:客户只需提供样品,Aksomics的技术服务人员就可为您完成全部实验操作,包括染色质免疫共沉淀、IP与 Input DNA文库构建、高通量测序和数据分析、并提供完整的实验报告。
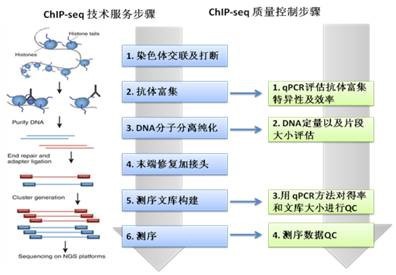
1. 染色质免疫共沉淀(ChIP)
*富集区注释(peak annotation)
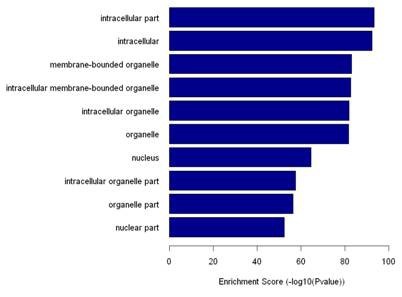
* 富集区相关基因的GO和Pathway
ChIP-seq 基本数据分析展示
1. Reads统计
|
|
Sample A
|
|
测序类型(单端/双端)
|
单端
|
|
reads长度
|
100bp
|
|
物种
|
Homo sapiens
|
|
基因组版本
|
HG19
|
|
Clean Reads(经过 CHASTITY过滤)
|
31,345,987
|
|
比对到基因组唯一位置的Reads
|
26,121,682
|
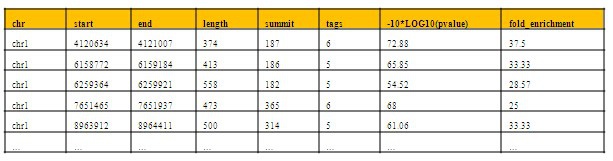
*图释:富集区域识别结果。
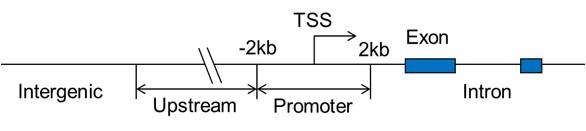
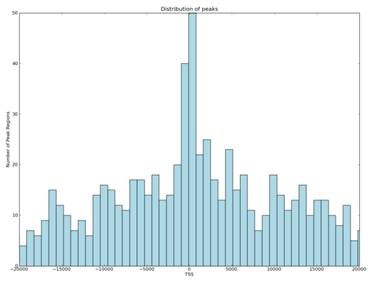
*图释:五种类型峰位置
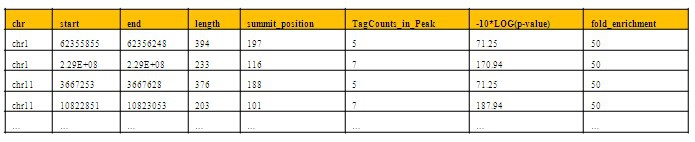
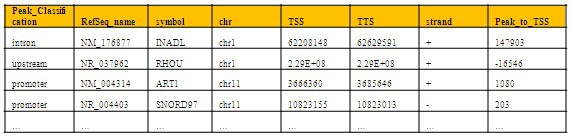
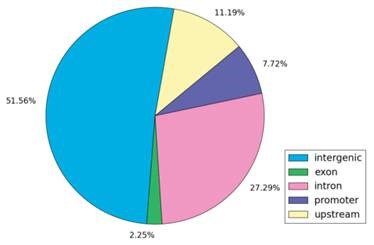
*图释:ChIP-Seq富集峰注释
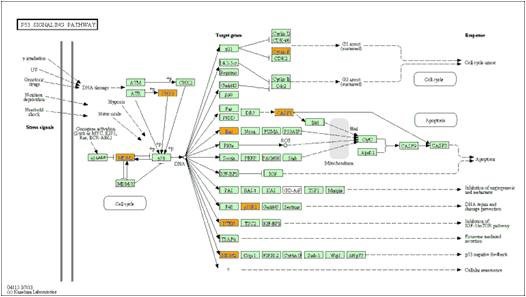

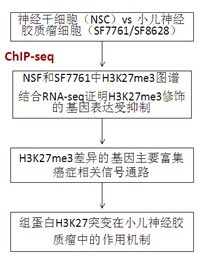
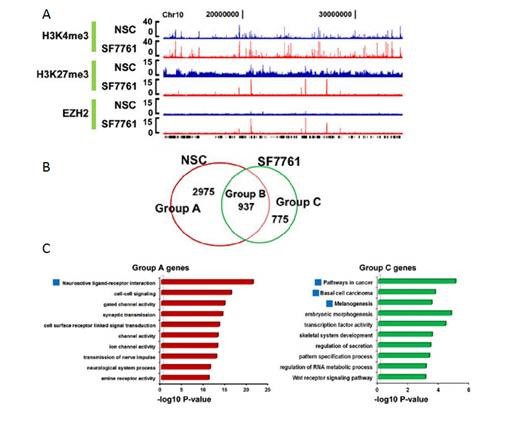
小儿神经胶质瘤的组蛋白修饰和基因表达调控(Chan, Fang et al. 2013)
→ Histone demethylase KDM6A promotes somatic cell reprogramming by epigenetically regulating the PTEN and IL‐6 signal pathways . STEM CELLS . 2020
→ Multi-generational obesogenic effects of sulfomethoxazole on Caenorhabditis elegans through epigenetic regulation . Journal of Hazardous Materials . 2019
→ MyoD1 suppresses cell migration and invasion by inhibiting FUT4 transcription in human gastric cancer cells . Cancer gene therapy . 2019
→ GATA3-dependent epigenetic upregulation of CCL21 is involved in the development of neuropathic pain induced by bortezomib. . Molecular Pain . 2019
→ Targeting TRPV1 on cellular plasticity regulated by Ovol 2 and Zeb 1 in hepatocellular carcinoma . Biomedicine & Pharmacotherapy . 2019
→ Targeting TRPV1 on cellular plasticity regulated by Ovol 2 and Zeb 1 in hepatocellular carcinoma . Biomedicine & Pharmacotherapy . 2019

