

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
转运RNA(tRNA)是一类广泛存在于生物体内,在肽链合成中负责氨基酸转运的非编码小RNA分子[1]。最新研究表明,tRNA通过多种方式主动参与mRNA翻译调控[2-4]。
tRNA表达水平影响mRNA翻译过程。在疾病发展的不同背景下,细胞内的mRNA密码子具有不同的使用偏好性,结合到这些密码子上的tRNA差异表达可直接影响翻译效率和准确性[2],细胞的增殖、分化和凋亡等状态均受到tRNA表达水平的动态调控。
tRNA分子的修饰状态对翻译过程也具有决定性影响,特定修饰(如m7G、m1A等)能够增强tRNA与核糖体的结合效率,从而提升mRNA的翻译速率和稳定性;而修饰异常则可导致核糖体停滞或错误解码、影响蛋白质合成[3, 5]。
此外,tRNA氨基酸负载状态也会影响mRNA翻译效率。由于只有已负载氨基酸的tRNA才能进入核糖体与密码子配对,因此氨酰化水平的变化直接决定翻译活性。当氨基酸负载比例下降时,翻译效率显著降低,甚至触发mRNA降解导致细胞状态异常或引起病变[4, 6]。
越来越多的研究表明,tRNA表达、修饰与氨基酸负载的异常与肿瘤[5, 7]、心血管疾病[8, 9]、神经退行性疾病[10]以及病毒感染[11]等密切相关。然而,由于tRNA分子具有高度稳定的二级结构和丰富的修饰,这些特性常常阻碍常规tRNA测序方法的文库构建与比对分析,导致检测效率低下和定量不准。
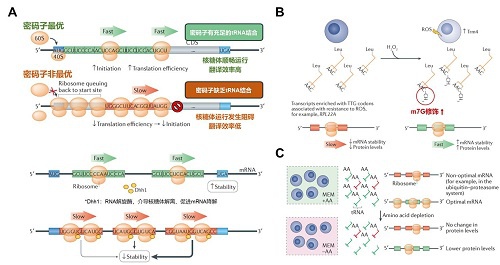
图1. tRNA表达水平(A)、修饰水平(B)、氨基酸负载比例(C)影响mRNA翻译和稳定性
Aksomics全新推出mim-tRNA-Seq,一种基于修饰介导cDNA错配的创新型tRNA测序方法,可在一次实验中同时检测tRNA的表达水平、修饰状态以及氨基酸负载比例[12]。该方法利用TGIRT酶进行全长tRNA的高效反转录、实现对tRNA表达水平的全面定量,通过3’末端CCA序列特征区分已负载与未负载氨基酸的tRNA,并通过识别cDNA错配碱基定位定量tRNA修饰水平。相比传统的tRNA测序方法,mim-tRNA-seq不仅能够克服修饰阻碍反转录的难点,还能在高通量条件下实现对tRNA表达水平、修饰水平、氨基酸负载比例三个维度的全面解析,为研究tRNA在肿瘤耐药、心肌纤维化及其他疾病中的作用机制提供了强有力的工具。
● tRNA研究一网打尽:仅进行一次实验即可同时检测tRNA表达水平、修饰状态及氨基酸负载比例。
● 准确的表达检测:通过高效的全长测序方法,实现对tRNA的精准识别和全面定量。
● 全面的修饰检测:精准识别多种修饰位点,支持同时检测m1A、 m1G,、m3C、m2, 2G等十余种修饰类型,覆盖全面。
● 无缝衔接翻译组学研究:通过3’端序列特征区分已负载与未负载氨基酸的tRNA并计算氨基酸负载比例,准确反映翻译活性。
● 结果丰富:高通量测序结合差异分析,提供表达、修饰与氨基酸负载的综合结果。
● 图表质量高:具备可直接用于文章发表的高质量数据与图表。
● 与Aksomics提供的其它服务平台联合分析:包括Ribo-seq、mRNA-seq、tRF&tiRNA-Seq、Arraystar small RNA表达芯片、Arraystar small RNA修饰芯片等。
mim-tRNA测序利用高效的全长测序方法、实现对tRNA的精准识别和表达水平的精确定量,并通过3’末端CCA序列特征计算tRNA氨基酸负载比例,同时实现tRNA多种修饰位点和修饰水平的全面检测。
1.tRNA表达水平定量和差异分析

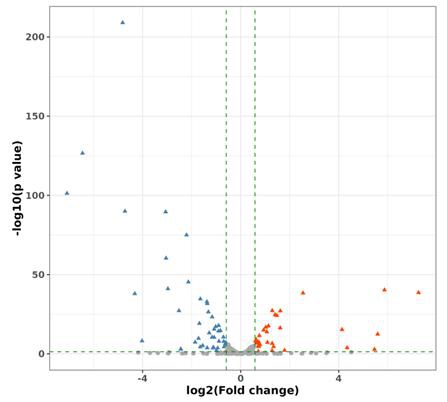
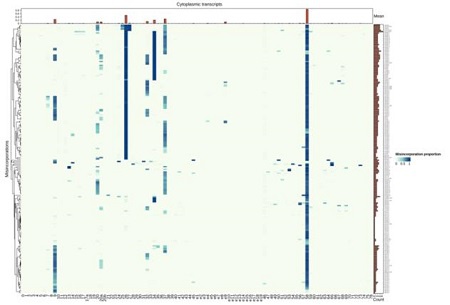
2.tRNA修饰水平定量和差异分析



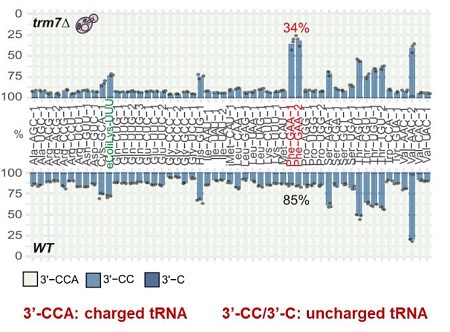
3.tRNA氨基酸负载状态定量和差异分析

1. Orellana, E.A., E. Siegal, and R.I. Gregory, tRNA dysregulation and disease. Nature Reviews Genetics, 2022. 23(11): p. 651-664.
2. El-Hachem, N., et al., Valine aminoacyl-tRNA synthetase promotes therapy resistance in melanoma. Nat Cell Biol, 2024. 26(7): p. 1154-1164.
3. Dai, Z., et al., N(7)-Methylguanosine tRNA modification enhances oncogenic mRNA translation and promotes intrahepatic cholangiocarcinoma progression. Mol Cell, 2021. 81(16): p. 3339-3355.e8.
4. Wu, J., et al., Glutamyl-Prolyl-tRNA Synthetase Regulates Proline-Rich Pro-Fibrotic Protein Synthesis During Cardiac Fibrosis. Circ Res, 2020. 127(6): p. 827-846.
5. Qi, L., et al., Dual Targeting of m(7)G tRNA Modification and Histone Acetylation using Carrier-Free Nano-Epidrugs to Evoke Osteosarcoma Chemosensitization. Adv Mater, 2025: p. e05951.
6. Duan, Y., et al., A glutamyl-tRNA reductase and its binding protein promote transitory starch biosynthesis and enhance grain quality and yield in rice. Plant Commun, 2025. 6(11): p. 101527.
7. Qian, Y., et al., ALKBH8-mediated codon-specific translation promotes colorectal tumorigenesis. Nat Commun, 2025. 16(1): p. 9075.
8. Nah, J., et al., Microprotein SMIM26 drives oxidative metabolism via serine-responsive mitochondrial translation. Mol Cell, 2025. 85(14): p. 2759-2775.e12.
9. Das, A.S., et al., AIMP3 maintains cardiac homeostasis by regulating the editing activity of methionyl-tRNA synthetase. Nat Cardiovasc Res, 2025. 4(7): p. 876-890.
10. Beharry, A., et al., High-fidelity and differential nonsense suppression in live cells and a frontotemporal dementia allele with human transfer RNAs. Nucleic Acids Res, 2025. 53(14).
11. Yang, X., et al., Anticodon Engineered Transfer RNA (tRNA(SUAG)) Inhibits Hepatitis B Virus Replication by Promoting the Degradation of Core Protein. Adv Sci (Weinh), 2025: p. e03534.
12. Behrens, A., G. Rodschinka, and D.D. Nedialkova, High-resolution quantitative profiling of tRNA abundance and modification status in eukaryotes by mim-tRNAseq. Mol Cell, 2021. 81(8): p. 1802-1815.e7.

