|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
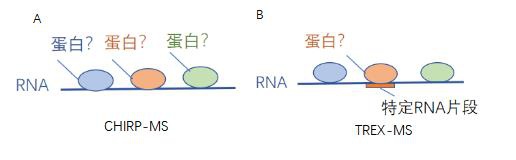
1、技术独特性:
RNase H酶切高效性:利用RNase H特异性切割“RNA-DNA”杂交链中的RNA链,释放此RNA链杂交区域的结合蛋白,反应具有高效性。
靶RNA区域选择性:可针对性研究目标转录本上某一特定区域的RNA互作蛋白。2、技术优势:
高特异性:通过设计特异性DNA探针,精准靶向目标RNA序列,释放并富集靶RNA区域的结合蛋白,有效避免非特异性结合蛋白的污染。
高灵敏度:与传统的RNA亲和捕获法相比,单次实验需要的起始细胞量减少。2.细胞裂解和相分离:
采用有机相分离技术在变性条件下裂解样本,可维持RNA-蛋白质复合物的完整性并实现有效回收。
3.靶RNA降解:采用液相色谱-串联质谱(LC-MS/MS)进行蛋白质组学分析,鉴定RBPs。
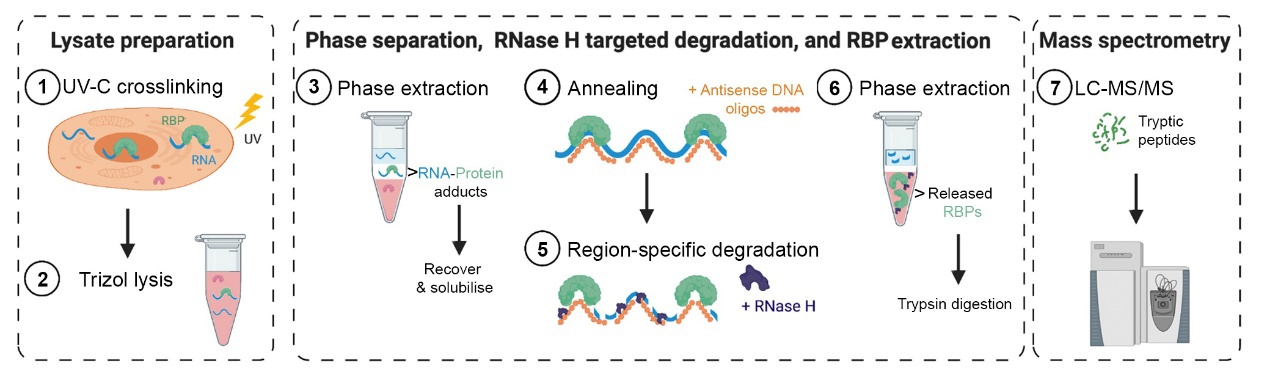
图 2 TREX实验流程
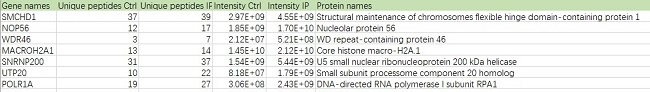
1.质谱检测结果
2.差异蛋白GO分析

3.差异蛋白KEGG pathway分析
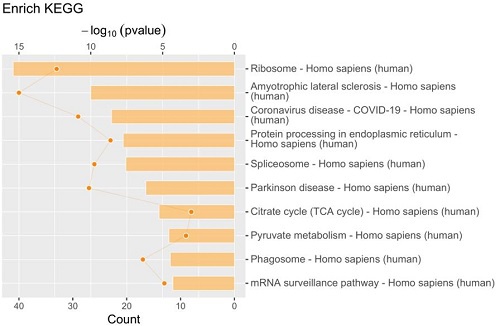
4.String蛋白质互作网络分析
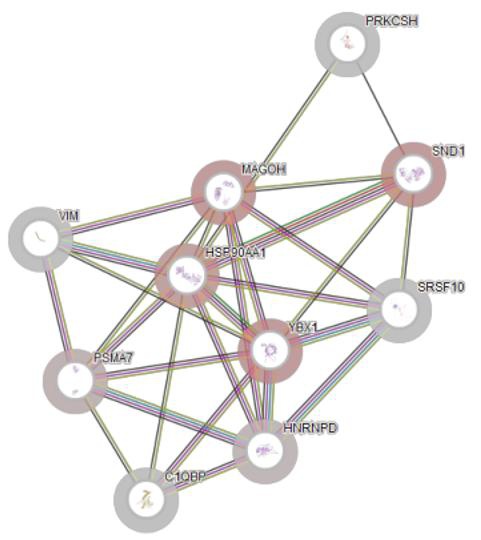
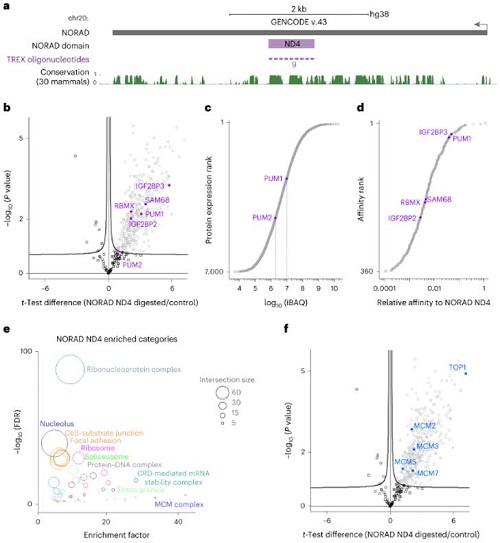
NORAD是一种长度为5.3 kb的长链非编码RNA,在大多数人体组织和细胞中呈现高表达[1]。研究表明,NORAD在DNA损伤条件下表达显著上调,其主要生物学功能是维持基因组稳定性[2]。NORAD包含5个重复区域(ND1-ND5),其中ND4区域呈现高度保守性,然而其直接互作蛋白尚未完全阐明。研究人员采用TREX-MS技术,采用9条反义寡核苷酸探针对ND4区域进行全覆盖杂交,成功鉴定出360个相互作用蛋白,包括已知的PUM1蛋白以及新型相互作用因子RBMX、TOP1等[3]。