

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
DNA(羟)甲基化是表观遗传调控的重要组成部分,在胚胎发育、神经分化以及肿瘤发生等生命活动中起着重要的作用,是当前研究的热点之一。DNA(羟)甲基化测序((h)MeDIP-seq)通过抗体富集高(羟)甲基化片段,并结合高通量测序平台,对所有富集的DNA片段进行高通量测序,研究人员能够获得全基因组范围内高精度的5mC/5hmC/6mA状态,为深入的表观遗传调控分析提供更有利的切入点。
服务名称
表观修饰
5mC- DNA甲基化测序(5mC-MeDIP-seq )
5mC
5hmC- DNA羟甲基化测序(5hmC-hMeDIP-seq )
5hmC
6mA - DNA甲基化测序(6mA-MeDIP-seq )
6mA
Aksomics(原康成生物)为您提供 (h)MeDIP-seq技术服务,您只需要提供保存完好的组织或细胞标本,Aksomics的技术服务人员就可为您完成全部实验操作,包括超声打断基因组、(羟)甲基化DNA免疫共沉淀、(h)MeDIP与 Input DNA文库构建、高通量测序和数据分析、并提供完整的实验报告。
● 灵活度高:能够直接对任意物种的高甲基化片段进行测序,无需已知的基因组序列信息。
● 检测范围广:覆盖整个基因组范围的甲基化区域,包括mRNA和、lncRNA和small ncRNA基因启动子的(羟)甲基化分析。
● 精确度高:能够在实际结合位点50个碱基范围内精确定位。
● 数字化信号:直接对甲基化片段进行测序和定量,不存在传统芯片杂交的荧光模拟信号带来的交叉反应和背景噪音问题。● 联合分析:能够与我司lncRNA芯片联合分析
Aksomics服务优势(以MeDIP为例)
1.MeDIP富集效果特异性佳:MeDIP是获得准确测序数据的关键。AksomicsMeDIP平台经过不断地优化,抗体富集效率高和特异性好。
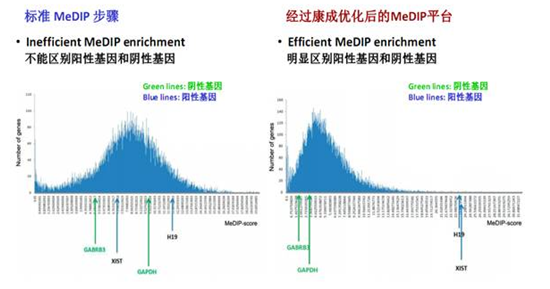
*图释:Aksomics优化后的MeDIP平台效果。横坐标为MeDIP-score其数值大小代表基因启动子甲基化水平,纵坐标为基因的数目。左图为用经典MeDIP方法构建测序文库的效果,阳性基因H19和Xist的信号无法和阴性基因GAPDH进行区分。右图为优化后的结果展示,阳性基因的MeDIP-score值远高于阴性基因,证明抗体富集的特异性。
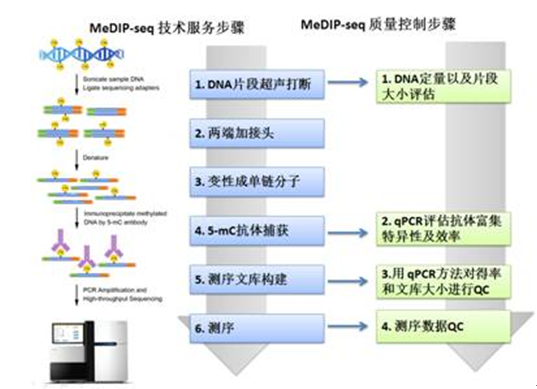
● 严格的质控体系:Aksomics在MeDIP-seq每个关键步骤都加入了质控实验。这些QC数据能够评估每个步骤的质量。如果达不到标准,我们会重复实验步骤或者优化实验体系,使得每个样品都能够顺利进入下个实验环节。
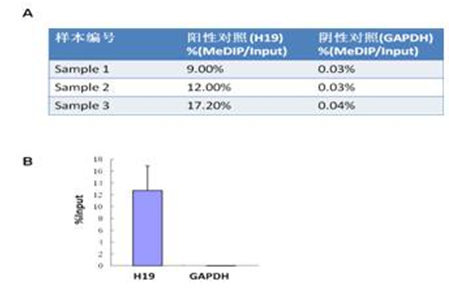
*图释:MeDIP-qPCR 质控。测序实验之前先对MeDIP实验效果进行评估,确定抗体富集的特异性。图中H19的启动子区域为阳性对照,管家基因GAPDH启动子区域为阴性对照,检测结果以MeDIP样品和Input样品的qPCR数据的比值表示。可以看出:MeDIP特异地富集甲基化的DNA区域,且阳性信号远高于背景噪音。
*图释:MeDIP-seq技术服务流程和关键步骤的质量控制
● 对DNA甲基化水平进行数字化定量分析:通过对测序得到的大量序列进行深入分析,MeDIP-seq得到的序列信息能够转换为可定量的甲基化水平信息。对基因组任意区域的甲基化水平给出定量的结果,并分析差异甲基化的区域。
● 可视化分析: 提供50bp精度的全基因组DNA甲基化信号可视化数据。通过UCSC基因组浏览器(UCSC genome browser),任意区域的甲基化水平能够直观的进行展示和比较。
1. (羟)甲基化DNA免疫共沉淀
2. 测序文库构建
3. DNA成簇(Cluster)扩增
4. 高通量测序
5. 数据分析
6. 提供实验报告
全基因组(羟)甲基化谱
1. (羟)甲基化峰识别:通过MACS软件,可以利用与基因组匹配的read来识别(羟)甲基化峰,常用的MACS筛选参数:q-Value≤10-4。
*mRNA相关的(羟)甲基化峰识别
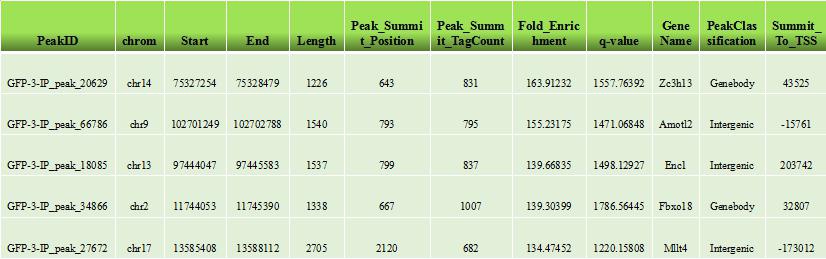
* lncRNA相关的(羟)甲基化峰识别
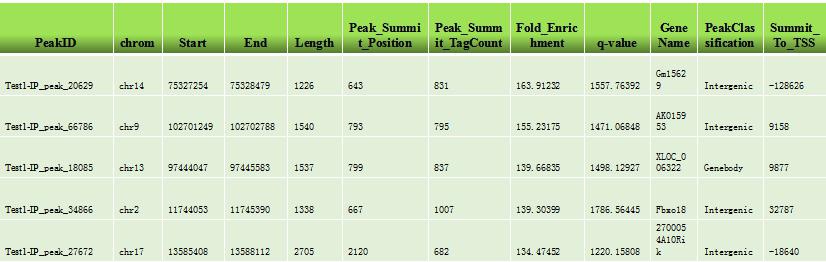
* Small_ncRNA相关的(羟)甲基化峰识别
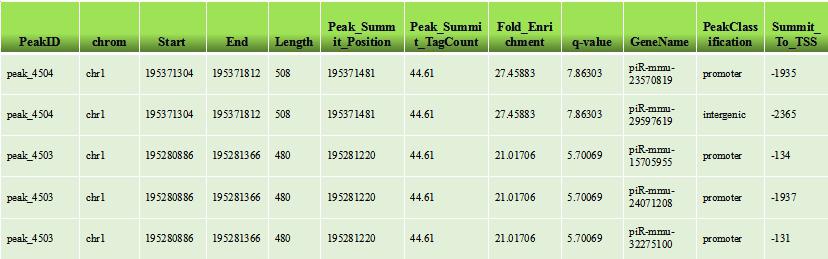
1. 识别(羟)差异甲基化基因
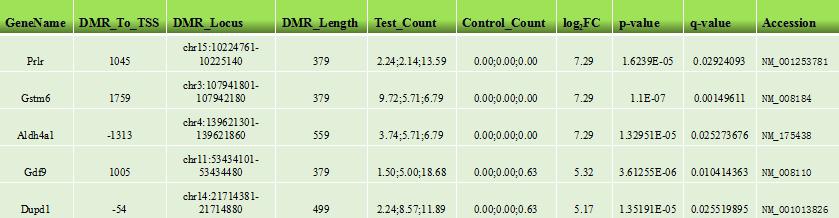
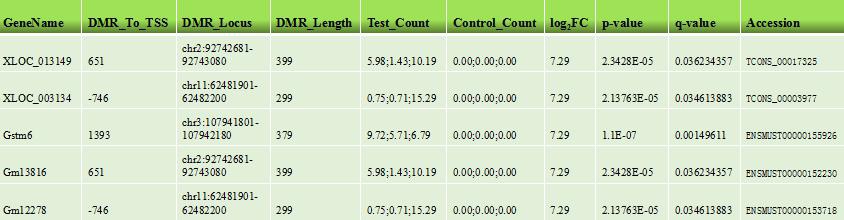
通过diffReps软件,Aksomics对不同样品或样品组间的基因启动子区域的差异(羟)甲基化进行了分析。为了对(羟)甲基化水平进行定量分析,处理组和对照组的样品分别通过相应的input样品进行标准化。根据处理组样品(羟)甲基化水平相对对照组的上下调情况,这些在启动子区域差异(羟)甲基化的基因被分成了“高(羟)甲基化和“低(羟)甲基化”基因。
* mRNA相关的启动子区域差异(羟)甲基化基因列表
* lncRNA相关的启动子区域差异(羟)甲基化基因列表
* Small_ncRNA相关的启动子区域差异(羟)甲基化基因列表
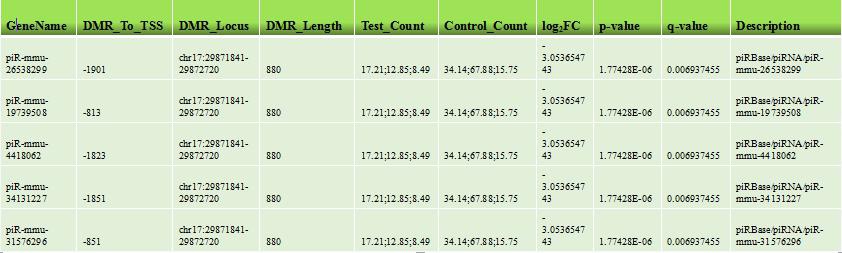
上表: 全基因组启动子区域差异甲基化数据列表
2. Enhancer & Super-enhancer相关的基因间差异(羟)甲基化列表
*Enhancer 关联的基因间差异(羟)甲基化基因列表
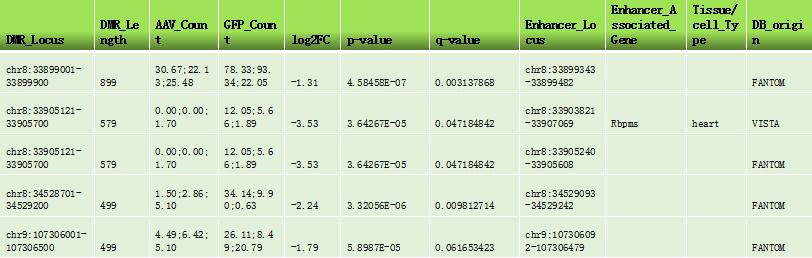
*Super-Enhancer 关联的基因间差异(羟)甲基化基因列表
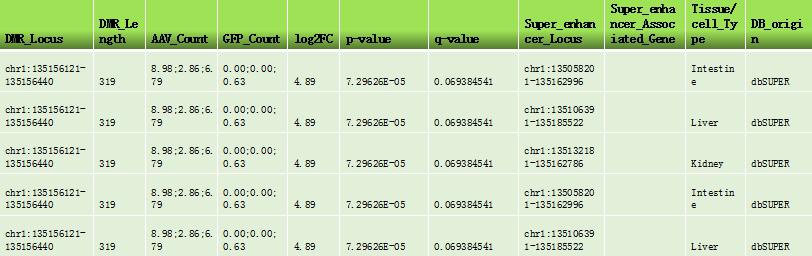
上表:增强子&超级增强子相关的基因间差异(羟)甲基化列表
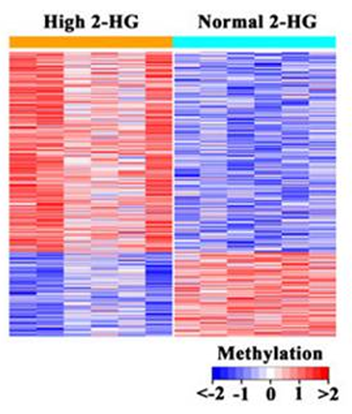
*图释:DMR区域聚类分析应用实例。AML病人按照血清中2-羟基戊二酸的含量分成2-HG高和2-HG正常两组。分离病人早期造血干细胞,检测DNA甲基化图谱,筛选组间差异甲基化的基因进行聚类分析。聚类结果表明两类病人呈现不同的甲基化特征。
4. 差异(羟)甲基化基因的GO分析
为了方便客户了解启动子差异(羟)甲基化基因的功能,Aksomics还分别提供了去(羟)甲基化基因和超(羟)甲基化基因的GO分析。
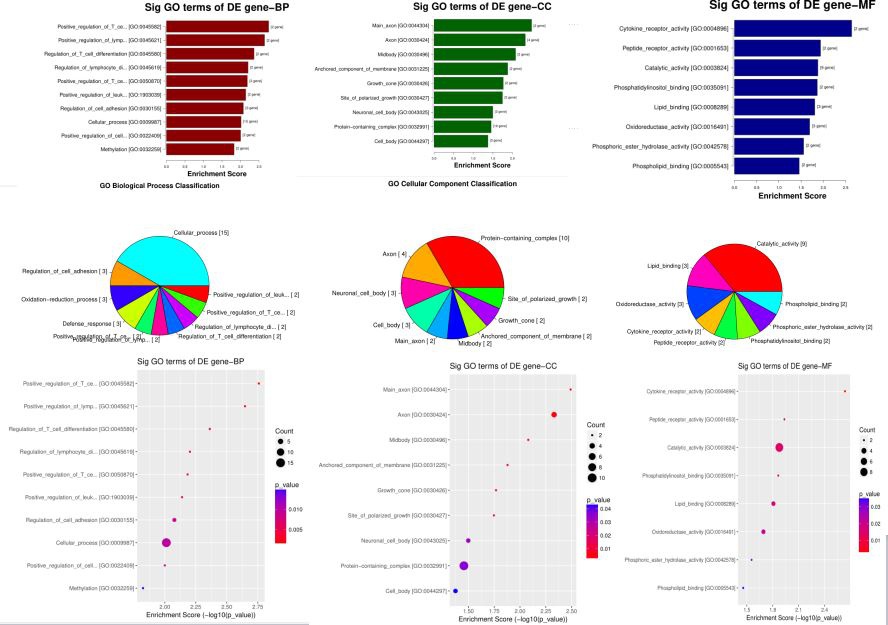
5. 差异(羟)甲基化基因的Pathway分析

可视化
Aksomics提供WIG格式的 (h)MeDIP-Seq(羟)甲基化谱数据,可以通过UCSC浏览器或IGB浏览器进行可视化。通过可视化图,客户可以直观的了解具体区域或基因的(羟)甲基化情况,以及样品间的差异(羟)甲基化状况。

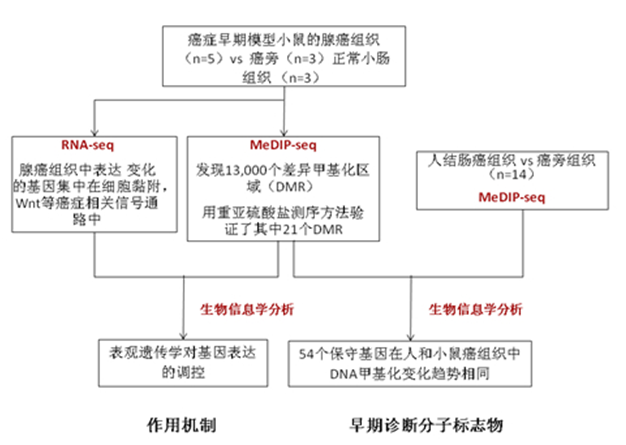
癌症早期诊断分子标记物筛选(DNA–Methylome Analysis of Mouse Intestinal Adenoma Identifies a Tumour-Specific Signature That Is Partly Conserved in Human Colon Cancer. Plos Genet. 2013)
小鼠模型可以模拟癌症发生的最初状态,找到疾病早期发生的表观遗传变化,这些变化位点可以作为肠腺癌早期诊断的分子标志物。研究者用MeDIP-seq检测疾病小鼠模型(APC基因缺失)的正常组织(n=3),腺癌组织(n=3)以及正常小鼠的小肠组织(n=3)样品。测序量为每个样品20 M reads, 平台为illumina。 比对腺癌组织和正常组织的数据,筛选得到13,000个差异甲基化区域(DMR)。
针对MeDIP-seq的数据进行生物信息分析,发现小鼠癌组织中DNA甲基化水平升高的基因显著地富集在PRC2靶标基因中,因此推测在癌组织中PRC2引起的组蛋白H3K27me3修饰变化和DNA甲基化之间存在互作关系。进一步通过表达谱的数据发现,DNMT3b,EZH2等表观修饰酶在癌组织中表达量发生改变,说明了小鼠腺癌组织发生表观遗传修饰改变的分子机制。
同时,研究者用MeDIP-seq方法对14对人结肠癌和癌旁组织行检测。综合两部分数据,找到54个保守基因在人和小鼠癌组织中呈现相同的DNA甲基化变化模式(图4),这些基因启动子的变化是在临床样本中的表现非常均一,是结肠癌中的核心表观遗传改变,可以作为早期诊断的候选标志物。
技术路线:
结果展示:
对小鼠组织的MeDIP-seq数据进行分析,发现腺癌组织和正常小肠相比有1,3000个区域发生甲基化水平的变化(DMR)。
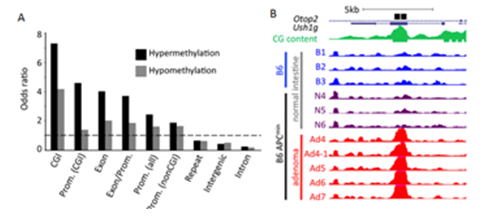
图1. A. 根据DMR在基因组的位置,分类进行统计分析。发现差异的甲基化区域主要集中在CpG岛以及包含CpG岛的启动子区域。CGI: CpG岛; Prom: 基因启动子; Exon:外显子; Exon/Prom: 外显子和启动子交界处; Repeat:重复序列; Intron: 内含子。
B. 应用UCSC browser可视化文件查看Ush1g基因的DMR,该区域在腺癌(Ad4, Ad4-1, Ad5, Ad6, Ad7)与正常小肠(B1, B2, B3),癌旁组织(N4,N5,N6)相比DNA甲基化水平升高。
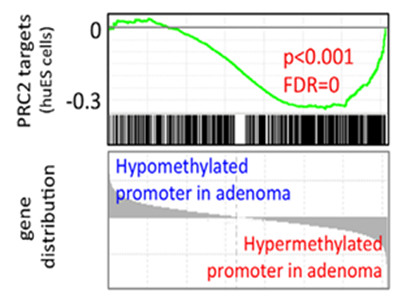
图2. GSEA分析:证明腺癌中甲基化水平升高的基因(hypermethylated promoter)显著富集在PRC2的靶标基因中,p<0.001, FDR=0。
小鼠腺癌和正常组织相比表达水平的差异基因和DNA甲基化水平的差异基因的联合分析,用韦恩图表示,如图3。
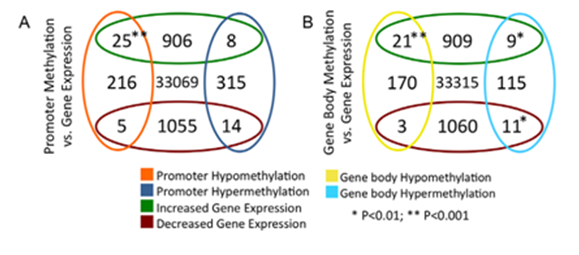
图3. A:启动子区域DNA甲基化水平变化的基因;启动子DNA甲基化水平降低的基因有25个表达上调,5个表达下调;启动子DNA甲基化水平升高的基因中有14个表达下调,8个表达上调。
B: Gene body的DNA甲基化水平发生变化的基因;Gene body DNA甲基化水平降低的基因中有21个表达上调,3个表达下调;Gene body DNA甲基化水平升高的基因中有11个表达下调,9个表达上调。
早期癌症小鼠模型的MeDIP-seq数据和14例临床样品的MeDIP-seq数据进行联合分析。54个保守基因在临床样本和小鼠模型中甲基化水平变化趋势相同。聚类分析表明这些基因的甲基化水平能够区分癌组织和正常组织,因此可以作为结肠癌早期诊断的标志物的候选分子。(图4)
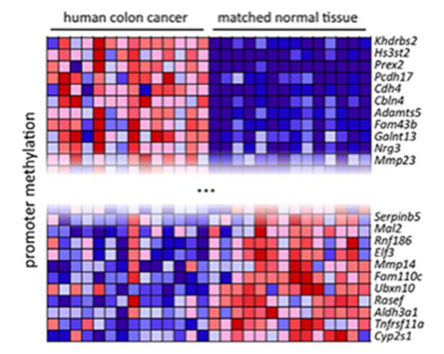
图4.聚类分析
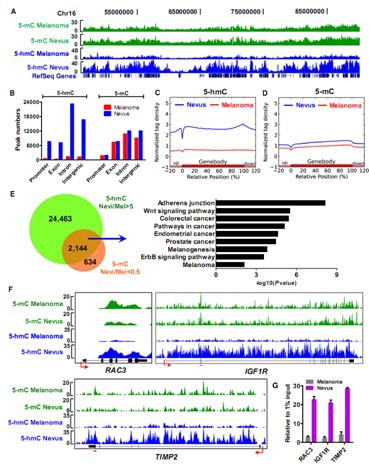
上图:黑色素瘤细胞中关键基因发生羟甲基化水平变化。A:UCSC可视化结果展示。黑色素瘤和痣组织相比整体羟甲基化水平减低,羟甲基化水平不变。B:对黑色素瘤和痣组织中检测到的DNA羟甲基化位点和羟甲基化位点进行分类统计。黑色素瘤与痣组织相比,在启动子,外显子,内含子,基因间等区域羟甲基化水平下降,羟甲基化水平基本不变。 C: 基因内部(Gene body)及周边(向两端各延伸20%)区段内的羟甲基化和羟甲基化reads(读段)密度分布。可以看到在黑色素瘤中,Gene body 内羟甲基化水平降低。E: 韦恩图显示癌组织中在Gene body区域羟甲基化水平降低同时羟甲基化水平升高的基因有2,144个。对这些基因进行pathway分析,发现变化的基因主要聚集在癌症相关的信号通路中。F: UCSC可视化结果展示,在RAC3, IGF1R, TIMP2中羟甲基化水平降低,羟甲基化水平升高。 G:hMeDIP-PCR对RAC3,IGF1R,TIMP2进行验证,证明在黑色素瘤中这些基因的羟甲基化水平降低,验证了测序的结果。
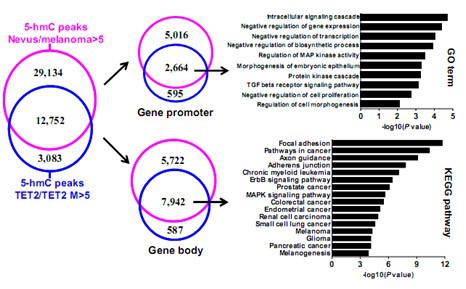
上图:过表达TET2后部分关键基因的羟甲基化水平得到恢复。构建TET2过表达的黑素素瘤细胞系,同时构建TET突变的细胞系作为对照。通过hMeDIP-seq检测,发现过表达TET2的细胞vs对照细胞 80%(12,752/15835)的羟甲基化升高的位点与黑色素瘤组织vs痣组织羟甲基化水平降低的位点重叠。将重叠的位点按照与基因的相对位置分成启动子和Gene body两个部分,分别进行GO和pathway分析。启动子重叠的基因聚集在信号传导,TGF-beta等重要的功能分类中;Gene body重叠的基因集中在MAPK, 细胞黏附等重要的通路中。
1.TRIM22 promotes the proliferation of glioblastoma cells by activating MAPK signaling and accelerating the degradation of Raf-1(Experimental & Molecular Medicine.2023)
2.High-temperature adaptation of anOsNRT2.3 allele is thermoregulated by small RNAs(Science Advances.2022)
3.EGR1 is crucial for the chlorogenic acid–provided promotion on liver regeneration and repair after APAP‑induced liver injury (Cell Biology and Toxicology.2023)
4.α5 Integrin regulates hepatic tight junctions through SRC-TET1-mediated DNA hydroxymethylation (iScience.2022)

