

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
DoGs形成了在癌症中反复检测到的嵌合RNA
嵌合RNA是通过相邻基因之间的顺式剪接(cis-SAGe)产生的,当RNA聚合酶II跳过终止信号时,产生两个相邻基因的前体转录本通读(图1)。 据估计,人类基因组中约5%的串联基因对可以转录成单一前体RNA,并最终被剪接成嵌合RNA。
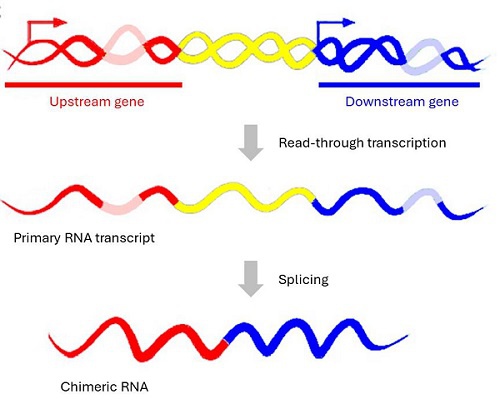
图1:嵌合RNA。同一链上的两个相邻基因通过转录通读产生一个前体RNA转录本,然后被剪接成嵌合RNA[1]。
嵌合RNA通常具有一些共同特征:(1)母基因是同一染色体上转录方向一致的相邻基因;(2)5’端的母基因处于活跃转录状态;(3)母基因之间的基因间距在30kb以内;(4)嵌合的接合点倾向于是5’端母基因的倒数第二外显子与3’端母基因的第二外显子融合,这被称为“2-2规则”;(5)嵌合体倾向于使用外显子的边缘被剪接在一起的经典剪接方式[2]。
嵌合RNA存在于癌症和正常生理状态中,且它们在癌症中可能存在异常表达。例如,与配对的正常前列腺样本相比,SLC45A3-ELK4在前列腺癌中的表达明显更高[3]。CHFR-GOLGA3在膀胱癌中的检测频率高于正常膀胱组织[4]。在前列腺癌细胞中,SLC45A3-ELK4的e1e2形式的嵌合RNA是通过相邻基因通读的顺式剪接产生的,而这不伴随着相应的DNA重排。CTCF与绝缘子序列的结合程度与嵌合转录本的表达呈负相关。SLC45A3-ELK4控制前列腺癌细胞增殖,其水平与前列腺癌的进展相关[2, 5, 6]。
SLC45A3-ELK4作为一个lncRNA发挥功能
SLC45A3-ELK4编码与ELK4相同的蛋白质。由于融合RNA水平低于野生型ELK4的1%,基本不影响ELK4的蛋白质总量。然而,当沉默融合RNA时,雄激素依赖性和去势抵抗性前列腺癌细胞中细胞增殖被抑制。外源过表达融合或突变体来阻止ELK4蛋白翻译可以挽救这种生长停滞。这个突变体也可以抑制CDKN1A和其他SLC45A3-ELK4的靶点。此外,与许多lncRNA类似,融合RNA在核分离物中富集。总的来说,这些结果表明SLC45A3-ELK4通过其RNA转录本而不是其翻译的蛋白来调节癌细胞增殖[7]。在没有转录终止的情况下,上游基因末端3’ss缺失,而其末端5’ss与下游基因的第一个3’ss剪接在一起,这是当RNAPII到达第二个外显子时从新生转录本中产生的(图2)。
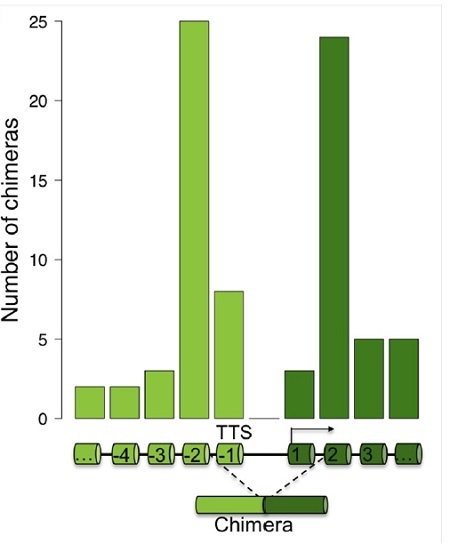
图2:通过基因间外显子剪接形成的通读RNA嵌合体的数量统计。
RNA嵌合体的一个显著特征是有些会在不同肿瘤样本中反复检测到。例如,CTSC-RAB38在20%的TCGA样本中检测到,但在任何配对的正常样本中都没有[8]。ccRCC细胞系中的嵌合RNA可以通过RNAi敲除后进行定量PCR验证(图3)。
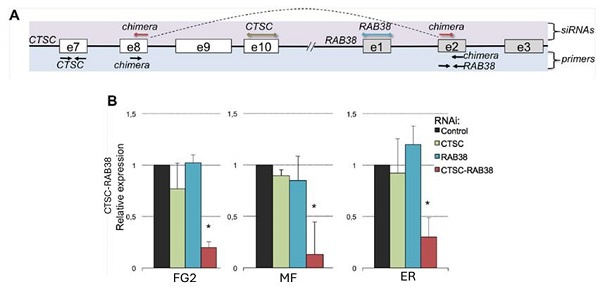
图3:CTSC-RAB38嵌合RNA位点示意图显示siRNA靶向CTSC、RAB38和嵌合的CTSC-RAB38转录本。这些转录本通过qPCR引物进行定量。虚线曲线表示产生嵌合RNA的剪接。(B)CTSC、RAB38和CTSC-RAB38区域在三种不同的ccRCC细胞系(FG2、MF和ER)中经过RNAi敲除后的相对CTSC-RAB38嵌合RNA表达。通过T检验与对照组相比较*p<0.05 [8]。
DoG形成环状RNA
通读转录会在下游区域生成环状RNA
环状RNA可以通过通读转录高效生成。一旦两个相邻的倒置重复被转录,反向剪接就会发生,从而形成RNA环并与新生转录本分离(图4)[7]。例如,MATR3-PAIP2通读转录,从下游基因PAIP2出产生circPAIP2(图5)[9]。
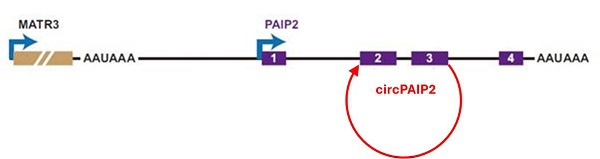
图4:MATR3-PAIP2的通读转录导致从下游PAIP2的外显子3到2处反向剪接产生circPAIP2[9]。
通读环状RNA(rt-circRNAs)
最近,发现了一种新型环状RNA,被称为通读环状RNA(rt-circRNAs)。它们是混合环状RNA,包含了来自两个相邻且转录方向相同的基因的编码外显子。Vo等人描述了1,359种人类rt-circRNAs[10]。
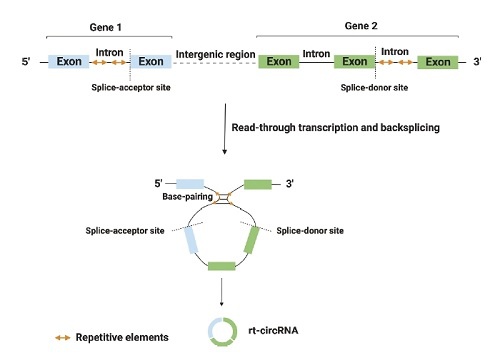
图5:通读转录产生rt-circRNAs的生物发生过程。通读转录导致形成混合环状RNA(rt-circRNAs),其中包括来自两个相邻且转录方向相同基因的编码外显子。环状化过程通过携带重复序列的长内含子之间的碱基配对来介导[11]。
相关服务
Downstream-of-Gene Transcript (DoG RNA)芯片
相关资源
什么是基因下游(DoG)RNA?
在疾病中DoGs对基因转录的调节功能探究
参考文献
1. Shi X, Singh S, Lin E, Li H: Chimeric RNAs in cancer. Adv Clin Chem 2021, 100:1-35.[PMID: 33453863]
2. Qin F et al: Discovery of CTCF-sensitive Cis-spliced fusion RNAs between adjacent genes in human prostate cells. PLoS Genet 2015, 11(2):e1005001.[PMID: 25658338]
3. Rickman DS et al: SLC45A3-ELK4 is a novel and frequent erythroblast transformation-specific fusion transcript in prostate cancer. Cancer Res 2009, 69(7):2734-2738.[PMID: 19293179]
4. Zhu D et al: The landscape of chimeric RNAs in bladder urothelial carcinoma. Int J Biochem Cell Biol 2019, 110:50-58.[PMID: 30818082]
5. Zhang Y et al: Chimeric transcript generated by cis-splicing of adjacent genes regulates prostate cancer cell proliferation. Cancer Discov 2012, 2(7):598-607.[PMID: 22719019]
6. Kumar-Sinha C, Kalyana-Sundaram S, Chinnaiyan AM: SLC45A3-ELK4 chimera in prostate cancer: spotlight on cis-splicing. Cancer Discov 2012, 2(7):582-585.[PMID: 22787087]
7. Qin F, Zhang Y, Liu J, Li H: SLC45A3-ELK4 functions as a long non-coding chimeric RNA. Cancer Lett 2017, 404:53-61.[PMID: 28716526]
8. Grosso AR et al: Pervasive transcription read-through promotes aberrant expression of oncogenes and RNA chimeras in renal carcinoma. Elife 2015, 4.[PMID: 26575290]
9. Liang D et al: The Output of Protein-Coding Genes Shifts to Circular RNAs When the Pre-mRNA Processing Machinery Is Limiting. Mol Cell 2017, 68(5):940-954 e943.[PMID: 29174924]
10. Vo JN et al: The Landscape of Circular RNA in Cancer. Cell 2019, 176(4):869-881 e813.[PMID: 30735636]
11. Vidal AF: Read-through circular RNAs reveal the plasticity of RNA processing mechanisms in human cells. RNA Biol 2020, 17(12):1823-1826.[PMID: 32783578]

