

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
组蛋白修饰H3K36me3的失调
在肾透明细胞癌(ccRCC)中,由于转录终止缺陷和随之而来的异常剪接,通读转录和DoG形成是广泛存在的[1] 。值得注意的是,H3K36me3修饰酶基因SETD2经常发生突变。在ccRCC细胞中敲除SETD2会诱导通读转录[1]。通读转录的现象也已在多种癌症中观察到,凸显了这种转录缺陷作为细胞转化的普遍原因[2, 3]。
组蛋白H2A.Z的缺乏
组蛋白H2A.Z是与DNA修复相关的一种替代组蛋白变体。在H2A.Z缺失的非增殖性衰老细胞中检测到DoGs(图1)[4]。DoGs通过作为反义转录本发挥关键作用来控制基因表达。
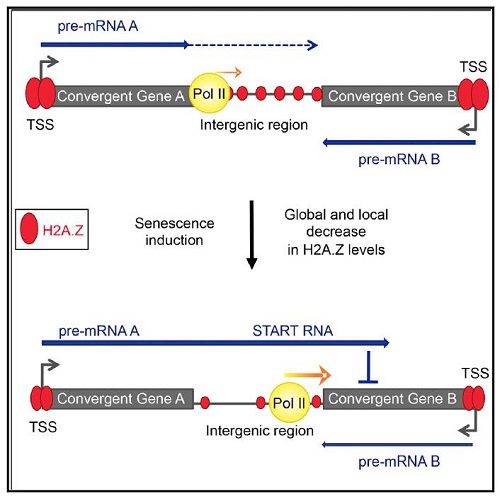
图1:通过转录通读调控衰老中基因表达。在衰老过程中,一类功能性反义RNA家族,称为START RNA(Senecence-Triggered Antisense Read-through RNAs),是通过在相反方向编码基因下游的转录通读产生的。这些RNA通过依赖于POL II延伸速率控制和H2A.Z的局部占据机制来被激活[4]。
RNA修饰
RNA修饰,如m6A,会影响转录终止[5, 6] 。在植物中,m6A修饰酶相关因子FIP37的突变体诱导了部分基因的通读转录和嵌合mRNA的形成[7] 。m6A辅助多聚腺苷化(m-ASP)通路的m6A修饰酶相关因子FIP37和m6A识别蛋白CPSF30L发挥功能,来确保转录组完整性[7] 。靶向FIP37和CPSF30L缺失植物的m-ASP通路会导致转录通读和mRNA嵌合体的形成(图2)。此外,m-ASP通路还可以限制嵌合基因/转座元件转录本的形成,并可能控制特定位点的转座元件。总的来说,对3’-UTR m6A的选择性识别作为一种保障机制,限制不恰当的基因表达并确保转录组的完整性[7]。
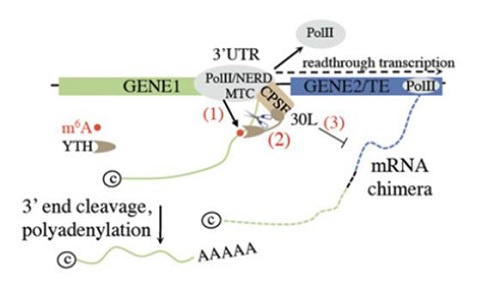
图2:DoG形成中的m6A辅助多聚腺苷化(m-ASP)通路模型[7]。 (1) m6A修饰酶相关因子FIP37对于基因1的3’-UTR位置的m6A修饰是必需的。 (2) m6A被CPSF30L识别蛋白的YTHDC型结构域识别,促进了在基因1的3’-UTR位置的切割和多聚腺苷化。 (3) 因此,mRNA嵌合体的形成受到限制。
m6A修饰和R-loop
新生RNA的m6A修饰促进了基因终止区域的R-loop形成,进而促进转录终止(图3)[8]。m6A甲基转移酶METTL3的敲除显著减少了m6A修饰的基因TES区域的R-loop积累,导致终止缺陷和通读转录[8]。METTL3甲基转移酶对于恢复受影响的TES区域的R-loop形成,并抑制随之而来的转录通读非常重要[8]。
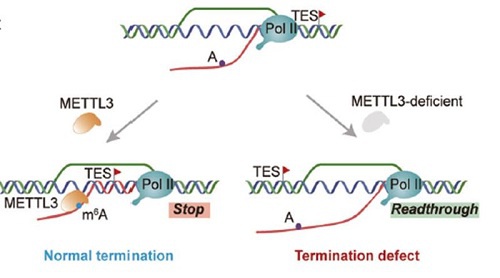
图3:m6A修饰通过促进终止区域R-loop的形成来实现高效的转录终止[8]。
最近发现,R-loop作为染色质锚点,招募DDX21和METTL3,二者共同促进m6A在新生转录本上的共转录修饰。新生RNA上的m6A对于招募核5’到3’外切酶XRN2起着关键作用。在转录终止的鱼雷模型中,XRN2进入多A裂解位点,降解正在进行转录的RNA,追逐RNAPII并使其从DNA上脱落,进而有效终止转录。m6A识别蛋白,例如YTHDC1可以结合并招募XRN2到转录终止位点以促进终止。任何这些步骤的破坏,包括DDX21、METTL3的缺失或酶活性的降低,都会导致终止缺陷和转录通读[9]。
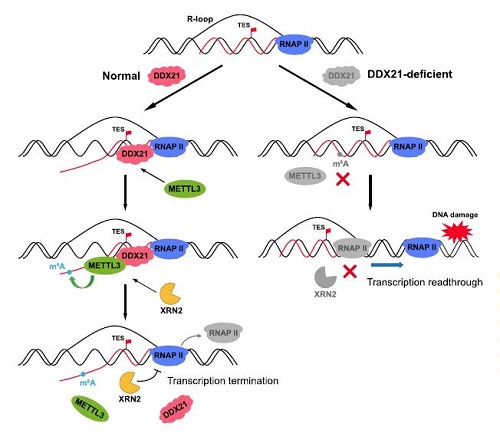
图4. DDX21-METTL3-m6A轴将m6A修饰添加到新生RNA上,在TES处消除R-loop结构,并招募XRN2终止转录。它们活性的缺失可能导致转录终止缺陷,DoG形成以及DNA损伤[9]。
相关服务
Downstream-of-Gene Transcript (DoG RNA)芯片
相关资源
什么是基因下游(DoG)RNA?
在疾病中DoGs对基因转录的调节功能探究
参考文献
1. Grosso AR et al: Pervasive transcription read-through promotes aberrant expression of oncogenes and RNA chimeras in renal carcinoma. Elife 2015, 4.[PMID: 26575290]
2. Maher CA et al: Transcriptome sequencing to detect gene fusions in cancer. Nature 2009, 458(7234):97-101.[PMID: 19136943]
3. Kannan K et al: Recurrent chimeric RNAs enriched in human prostate cancer identified by deep sequencing. Proc Natl Acad Sci U S A 2011, 108(22):9172-9177.[PMID: 21571633]
4. Muniz L et al: Control of Gene Expression in Senescence through Transcriptional Read-Through of Convergent Protein-Coding Genes. Cell Rep 2017, 21(9):2433-2446.[PMID: 29186682]
5. Frye M, Harada BT, Behm M, He C: RNA modifications modulate gene expression during development. Science 2018, 361(6409):1346-1349.[PMID: 30262497]
6. Anreiter I et al: New Twists in Detecting mRNA Modification Dynamics. Trends Biotechnol 2021, 39(1):72-89.[PMID: 32620324]
7. Pontier D et al: The m(6)A pathway protects the transcriptome integrity by restricting RNA chimera formation in plants. Life Sci Alliance 2019, 2(3).[PMID: 31142640]
8. Yang X et al: m(6)A promotes R-loop formation to facilitate transcription termination. Cell Res 2019, 29(12):1035-1038.[PMID: 31606733]
9. Hao JD et al: DDX21 mediates co-transcriptional RNA m(6)A modification to promote transcription termination and genome stability. Mol Cell 2024, 84(9):1711-1726 e1711.[PMID: 38569554]

