

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
鉴定和检测体内 RNA G-四联体(rG4)是研究细胞生物学和人类疾病中 rG4功能的必要步骤。基于二代测序(NGS)的rG4检测技术,比如 G4RP-seq[1]、BG4 uvRIP-seq 或DMS-seq联合RT 停滞位点检测[2]等,已被用于研究整个转录组中的 rG4 表达情况,并评估各种条件下的定量变化。然而,这些方法在准确定量检测 rG4 上均存在一定的局限性。
在G4RP-seq 和 uvRIP-seq 中,需要对样本进行交联以保留细胞内瞬时形成的rG4结构。然而,间接的 RNA-RNA 相互作用也可能被交联,从而可能导致假阳性结果[3].
此外,在捕获 rG4 的过程中,rG4抗体或配体与 rG4 的结合可能会受到 rG4 结合蛋白(如 G3BP1 和 nucleolin)的阻碍,这些蛋白在体内优先与 rG4 结构相互作用并占据结合位点。例如,G3BP1 通过其 C 端 RGG 结构域直接与 rG4 结合,并调节 mRNA 的稳定性[4]。这些蛋白对 rG4 的占位及其交联会阻碍 rG4 抗体或小分子配体的后续结合,从而导致假阴性检测结果。
Arraystar (in vivo)rG4芯片 vs DMS-seq 联合 RT停滞位点检测
DMS-seq 联合 RT停滞位点检测方法包含了体内 DMS 处理步骤和 RT 停滞位点分析。处于展开状态的 rG4 区域中的鸟嘌呤(G)在其 N7 位置被 DMS 甲基化,而折叠的 rG4 结构中的鸟嘌呤(G)受G4结构保护不会被 DMS 甲基化[2]。受体内 rG4 结构保护而未被 DMS 甲基化的鸟嘌呤可以在体外重新折叠成 rG4,而被甲基化的鸟嘌呤则不能,因此可以间接推断出体内 rG4 的折叠状态。
不过,DMS 同时也能在 N1 位甲基化腺苷(A)形成m1A和在 N3 位甲基化胞嘧啶(C)形成m3C(图 1)。在 DMS-seq 加RT停滞位点检测分析中,反转录酶不仅会在遇到折叠的 rG4 时停滞和链终止,也会在遇到 m1A 或 m3C 时停滞和链终止。因此,检测到的RT停滞位点既可能是折叠的 rG4 位点,也可能是 m1A 或 m3C 位点,这会导致对真正的 rG4 位点的定量不准确。
Arraystar (in vivo)rG4芯片平台使用 G4 特异性抗体 BG4 来亲和性捕获含有 rG4 结构的 RNA 片段,这样产生的 rG4 信号比依靠逆转录酶停滞所产生的信号更加真实。然后,通过去甲基化处理去除所捕获 RNA 的m1A与m3C甲基化,以实现无障碍、无偏倚的反转录,并进行 cRNA合成 和 rG4芯片检测。体内 DMS 处理和体外重折叠保留并恢复了真正的原生 rG4 构象,再加上抗 G4 抗体 BG4,就能高效、特异地捕获真正的含 rG4 的 RNA 片段。最后,rG4-RNA 通过 Arraystar (in vivo)rG4芯片进行检测,比测序更加灵敏。因此,Arraystar (in vivo)rG4芯片平台是最准确、最灵敏的体内 rG4 分析平台。
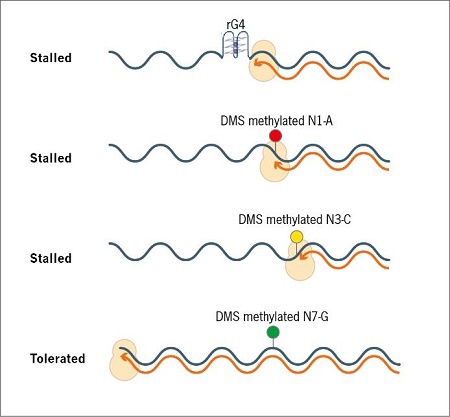
图 1 DMS-seq联合 RT停滞分析时,无论是在遇到折叠的 rG4 时,还是在遇到 DMS 甲基化的 m1A 或 m3C 时,反转录酶(RT)都会停止,cDNA 链也会终止,从而导致对 rG4 位点的错误识别。
RNA-seq无法准确定量低丰度的rG4
活细胞(体内)中 rG4 的形成情况是研究 rG4功能的必要信息。其中一个主要难点是,rG4s 在体内的折叠和展开是快速和动态的,而且其自然平衡是倾向于展开状态的。因此,细胞中含 rG4 的 RNA 数量非常少,其丰度较低,很难捕捉。
在 RNA-Seq 中,大多数测序reads来自高丰度 RNA(如管家基因),而低丰度 RNA 的覆盖率较低。低测序深度降低了检测低丰度RNA 片段的灵敏度和定量的可靠性。然而,增加测序深度并不能线性地提高低丰度转录本的灵敏度和准确性,而且随着测序深度增加到一定程度,其边际收益会迅速降低。因此,rG4-RNA 本身的低丰度水平导致基于 RNA-seq 的方法难以准确检测和量化它们。
例如,lncRNA-MALAT1 是一种因其 rG4 形成而被广泛研究的 RNA,但在通过测序(G4RP-Seq)绘制其rG4图谱时,就遇到了这一问题 [3]。G4RP-Seq 需要添加 G4 稳定配体 BRACO-19 和 RHPS4 (它们可以人为地诱导和稳定 G4),才能检测到来自 MALAT1 的显著 rG4 信号[1]。然而,这种方法并不适合在正常生理条件下观察自然的、瞬时的rG4。
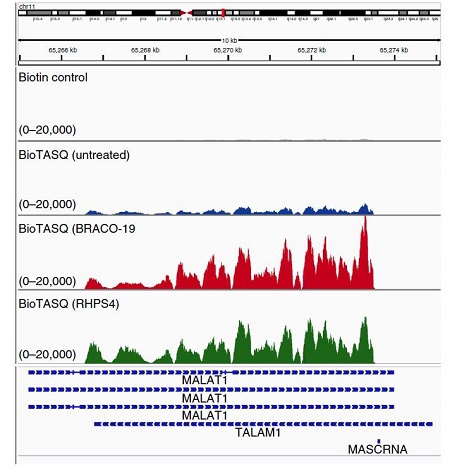
图 2 G4RP-Seq中,生物素对照、未处理、BRACO-19 处理和 RHPS4 处理样本 [3] 的 MALAT1 rG4 信号。虽然用 G4 稳定配体 BRACO-19 或 RHPS4 处理会产生较高的 rG4 信号,但这些 rG4s 是人工诱导和稳定的 rG4s [1],不能代表体内原生的瞬时 rG4 状态,也不适合观察正常生理条件下的天然 rG4s。
此外,在RNA测序中,RPKM(Reads Per Kilobase of transcript per Million reads mapped)用于对文库大小和基因长度的差异进行均一化处理。然而,RPKM 值反映的是样本中转录本的相对丰度,RNA 成分的变化会影响整个 RNA 图谱,使用 RPKM 比较不同样本的转录本表达水平可能会产生误导。在进行跨样本比较时,这些差异会扭曲 RNA 表达水平。因此,RNA-seq 的 rG4 图谱不适合用于比较不同样本间的 rG4 丰度。
而使用Arraystar (in vivo)rG4芯片分析rG4时,RNA 目标与其序列特异性探针杂交,不受其他 RNA 序列的影响,甚至不受高丰度 RNA 的影响。在 RNA 测序中,高丰度 RNA(如管家基因 RNA)会挤占低丰度 RNA 的覆盖度,但它们对芯片上低丰度转录本的检测几乎没有影响。因此,基于芯片的分析平台特别适合定量检测低水平表达的 rG4-RNA。
相关服务
参考文献
1.Yang SY et al: Transcriptome-wide identification of transient RNA G-quadruplexes in human cells. Nat Commun 2018, 9(1):4730.[PMID: 30413703]
2. Guo JU, Bartel DP: RNA G-quadruplexes are globally unfolded in eukaryotic cells and depleted in bacteria. Science 2016, 353(6306).[PMID: 27708011]
3. Yang SY, Monchaud D, Wong JMY: Global mapping of RNA G-quadruplexes (G4-RNAs) using G4RP-seq. Nat Protoc 2022, 17(3):870-889.[PMID: 35140410]
4. He X, Yuan J, Wang Y: G3BP1 binds to guanine quadruplexes in mRNAs to modulate their stabilities. Nucleic Acids Res 2021, 49(19):11323-11336.[PMID: 34614161]

