

R-loop是由转录的RNA链与打开的双链DNA其中一条模板链发生碱基互补配对,形成RNA-DNA杂合链,同时使未配对的另一条DNA链游离在外组成的三方结构 [1]。它们分布广泛,占哺乳动物基因组的 5% [2,3]。R-loop经常出现在基因组的启动子和转录终止位点, 影响R-loop形成的结构因素包括高度的GC偏倚(GC skew,在TSS下游的非模板链上,G比C富集)、G四联体、DNA缺口和DNA/RNA修饰等[4]。除了mRNA新生转录本外,lncRNA和circRNA均可形成R-Loop,在调节基因表达、DNA 复制以及 DNA 和组蛋白修饰方面发挥着重要作用,具有重要的生物学功能。


图1 R-loop的结构[5]。
R-loop积累是包含未甲基化CpG岛的启动子的一个特征,这些启动子的序列同时也具有GC偏倚的特点[6]。全基因组范围的检测显示,R-loops在DNA甲基化降低和DNA酶超敏性(与染色质可及性相关)增加的位点上富集[7]。R-loops可以通过阻止DNA (胞嘧啶-5)甲基转移酶(DNMTs)[8,9]与DNA的结合,来抑制启动子区的DNA甲基化。图1展示了一个R-loops介导的基因调控的例子,其中R-loops通过阻止DNA与DNMTs的结合,保护BAMBI的基因启动子免受DNA甲基化修饰的影响[9]。
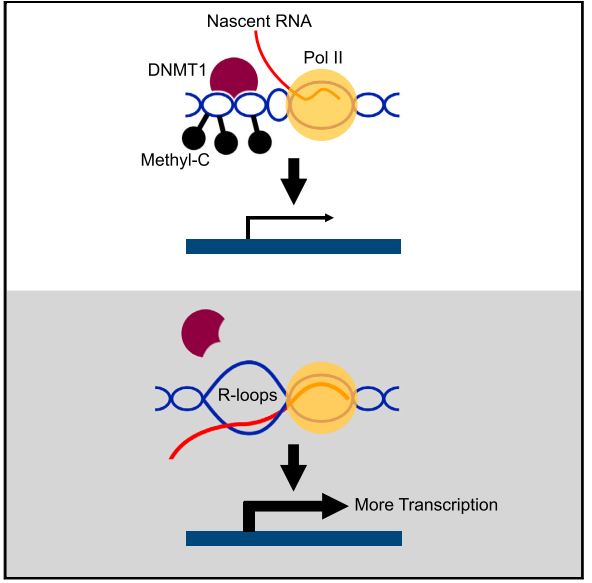
• mRNA形成R-loops发挥“类启动子”功能促进基因转录
R-loops可以通过促进/阻碍转录因子的结合,从而调控转录起始过程。这种调控作用主要发生在启动子区,通过阻碍转录因子的结合抑制转录起始,促进转录因子结合或封闭转录抑制因子的结合位点激活转录发生(图3)。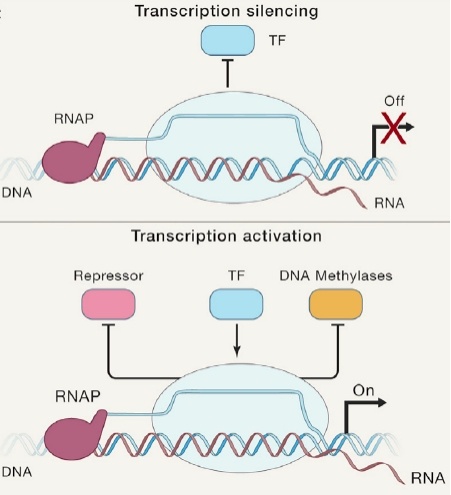
图 3 R-loop通过促进/阻碍转录因子的结合,进而调控基因表达。(上图)通过阻碍转录因子结合沉默转录过程,(下图)通过促进转录因子结合或封闭转录抑制因子结合位点激活转录过程。
由于RNA聚合酶II(Pol II)以无核小体的RNA为模板促进转录起始,正如在基因启动子区发现的现象相同。与此相似,R-loops结构中的单链DNA成分,具有直接促进Pol II反义RNA(Antisense RNA,AS)转录的潜力,而不需要常规转录因子(General Transcription Factors,GTFs)将双链DNA打开成为单链DNA。当新生的编码RNA转录本侵入DNA双链时即可形成R-loops结构,此时在延伸中的RNA Pol II可以作为反义lncRNA的启动子元件。R-loops结构中的单链DNA作为模板用来转录反义RNA(图4)。
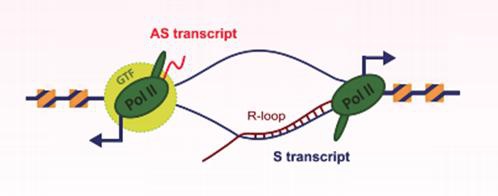
图 4 R-loops作为反义lncRNA的启动子元件。R-loop结构通常形成于人蛋白编码基因的启动子区域,并且可以作为反义lncRNA的启动子元件促进反义lncRNA的生成。S transcript:编码RNA;AS transcript:反义RNA;GTF:常规转录因子;Pol II:RNA聚合酶II[15]。
R-loops促进DNA低甲基化的另一种机制是通过吸引DNA去甲基化酶TET (ten-eleven translation DNA demethylases)。例如图5所示,在mESCs细胞中,lncRNA TARID在TCF21基因的富含CpG岛的启动子区生成一个R-loop,同时lncRNA TARID的转录方向与TCF21相反。GADD45A识别并结合到TCF21启动子的R-loop上,招募DNA去甲基化因子TET1,导致TCF21的转录激活[10]。
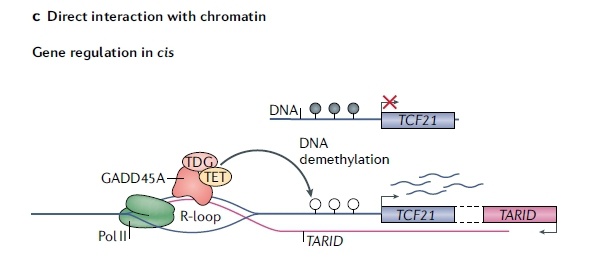
图 5 GADD45A与R-loops结合并招募TET1到TCF21启动子区的CpG岛。
• lncRNA通过形成R-loops直接招募染色质修饰复合物影响染色质构象变化
案例 1:在小鼠胚胎干细胞的分化过程中,有两个与lncRNA结合的染色质修饰复合物起了非常重要的作用,一个是多梳复合物PRC2(与基因抑制有关),一个是组蛋白乙酰转移酶复合物TIP60(也称为KAT5)- p400(与基因激活有关),这些lncRNA也能够参与R-loops的形成[6](图6)。倾向于形成R-loops的启动子区域能够通过形成的R-loops招募TIP60-p400复合物并激活临近基因转录。事实上,RNase H1在小鼠胚胎干细胞中的过表达能够降低R-loops的水平,因而降低了TIP60和p400在大多数靶基因中的定位,使这些靶基因能够招募PRC2,改变基因表达状态。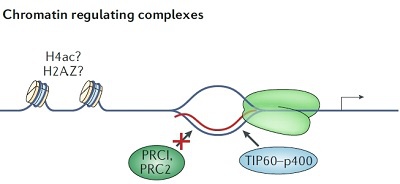
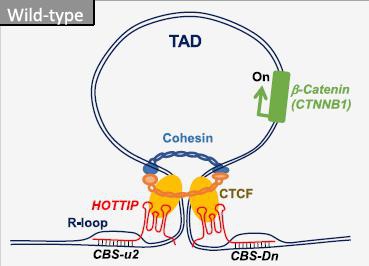
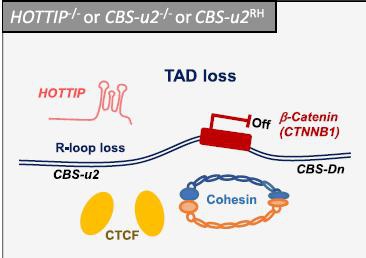
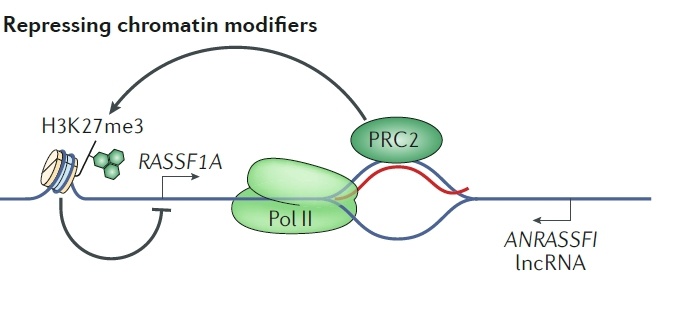
图 8 lncRNA ANRASSF1能够在RASSF1A转录本的启动子区形成R-loop结构,这一结构能够作为一个招募平台招募染色质修饰复合物PRC2,通过该对H3组蛋白的27位赖氨酸进行三甲基化修饰(H3K27me3)抑制RASSF1A转录本的表达[12]。
• R-loops作为lncRNA锚点,使部分单链lncRNA招募染色质重构复合物
案例1:部分lncRNAs在细胞中作为单链RNA存在,能够与染色质重构复合物结合。此外另一部分则存在于RNA-DNA杂合链中(参与R-loop形成),这些lncRNA因此被锚定在特异的基因组位点上。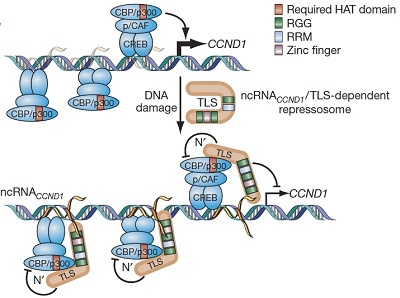
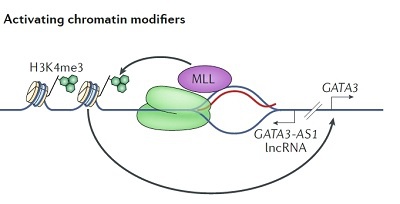
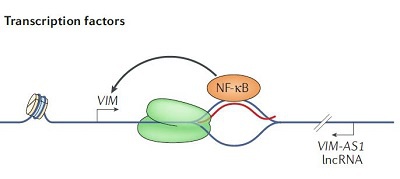
• lncRNA形成驻留R-loop抑制转录延伸
案例 1:R-loop的驻留是一个在多种生物中影响转录延伸的常规机制,在拟南芥中,寒冷环境诱导FLOWERING基因区域产生的lncRNA COOLAIR,转录后停留在其转录位点上,并促进该位点的PRC2依赖的H3K27me3修饰。详细来讲,COOLAIR是来源于拟南芥FLOWERING基因位置C(FLOWERING LOCUS C)的3’端一系列反义转录本。体内实验发现,单链DNA是COOLAIR启动子区的RNA-DNA杂合链或R-loop结构的一部分。同源结构域蛋白AtNDX可以结合在R-loop的单链DNA链上稳定R-loop结构,而后可能导致Pol II暂停或COOLAIR转录阻滞。AtNDX促进R-loop的稳定驻留,进而抑制COOLAIR转录,从而调控FLC表达[17](图12)。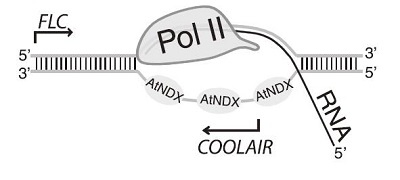
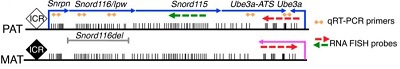
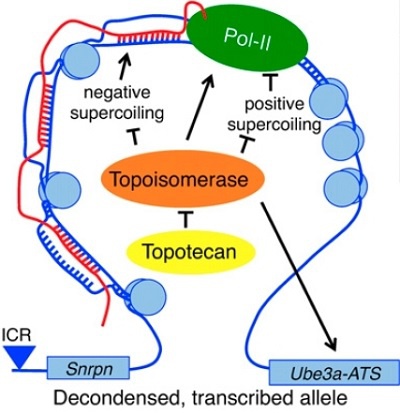
案例 2:Snord116 或 Ube3a 变异与神经发育遗传病 Prader-Willi 综合征 (PWS) 和 Angelman 综合征 (AS) 相关。在非编码 RNA Snord116 基因区域 R-loop 结构的形成抑制了转录在非编码 RNA Ube3a-ATS 基因处的延伸,从而抑制 Ube3a-ATS 的表达。由于 Snord116 基因处存在 GC 偏倚的重复单元,使该区域更倾向于形成 R-loop。在生理条件下,R-loop 的形成促进转录复合体通过 Ube3a-ATS 进而顺式沉默蛋白编码基因 Ube3a 在神经元中表达。然而,拓扑异构酶抑制剂拓扑替康处理等条件下,会形成过量的 R-loop,R-loop 的形成导致过量的转录复合体停滞,从而抑制 Ube3a-ATS 的表达,反过来促进蛋白编码基因 Ube3a 的表达 [18](图13)。
• lncRNA形成的R-loops通过反式诱导作用调控基因表达
R-loop的形成或许并不需要相同区域的共转录。富含G的RNA通过分子内的G四链体持续与富含C的DNA碱基互补配或许有助于反式诱导的R-loop形成。反式诱导的R-loop比顺式形成的R-loop更可能威胁到基因组的完整性。顺式诱导的R-loop只能形成于RNA转录区域,然而反式诱导的R-loops可以形成于基因组中的多个位置,产生多个基因组不稳定的“热点”区域。
lncRNA APOLO(auxin-regulated promoter loop,生长素调控的启动子环)负责拟南芥中生长素反应基因的激活。APOLO靶向的基因通常都处于沉默状态,且被多梳因子样异染色质蛋白1(Polycomb factor like heterochromatin protein 1,LHP1)维持在沉默状态。对生长素响应的APOLO转录激活后,APOLO识别其靶基因启动子区的特异性基序,结合在靶基因的启动子区形成R-loop,并锚定在靶基因的启动子区,单链的APOLO RNA作为LHP1的诱饵,从而促进靶基因表达(图14)。
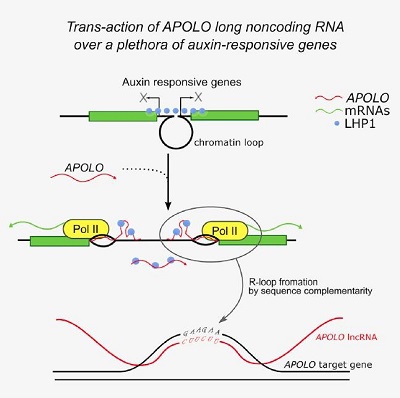
图 14 拟南芥中,lncRNA APOLO通过反式作用形成R-loop,成为生长素反应基因的广泛调控方式中的一部分。长链非编码RNA APOLO通过碱基互补配对形成DNA-RNA双链体(R-loop结构)识别多个远端的靶基因。APOLO与其靶基因的互作是基于核酸序列的直接互补配对。APOLO靶基因的共有序列是GAAGAA(G/C)。APOLO包含两个TTCTTC核心,可以完美地通过序列互补配对识别靶基因的共有序列。APOLO的RNA水平影响R-loop的形成,和这些远端基因的转录活性,包括一系列的生长素响应基因(比如WAG2和AZG2),进而在拟南芥侧根形成中协调生长素响应基因的表达[19]。
circRNA形成的R-Loops
SMARCA5是DNA损伤修复过程中的重要蛋白,参与DNA损伤区域的染色质重塑,促进DNA损伤修复因子的招募。SMARCA5的外显子15和16发生back-splicing,形成circSMARCA5。在乳腺癌细胞中,circSMARCA5表达出现明显下调,过表达circSMARCA5可以诱导乳腺癌细胞系的药物敏感性[21]。机制上,circSMARCA5能与其母基因座结合,形成一个R-loop,从而导致SMARCA5的第15外显子的转录暂停,进而导致产生截断的无功能蛋白和SMARCA5表达的下调(图15)。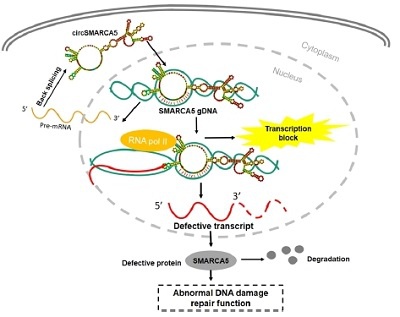
图 15 circSMARCA5在母基因mRNA SMARCA5 gene body区形成R-loop,导致转录暂停,产生有缺陷的转录本,翻译出截断的无功能蛋白,进而被降解,使得DNA损伤修复过程出现异常[20]。
参考文献
1. S. Hamperl, K.A. Cimprich, The contribution of co-transcriptional RNA:DNA hybrid structures to DNA damage and genome instability, DNA Repair 19 (2014) 84–94.
2. L.A. Sanz, et al., Prevalent, dynamic, and conserved R-Loop structures associate with specific epigenomic signatures in mammals, Mol. Cell 63 (1) (2016) 167–178.
3. Li. Miaomiao, et al., Modifications and interactions at the R-loop, DNA Repair 96 (2020) 102958.
4.Christof Niehrs and Brian Luke, Regulatory R-loops as facilitators of gene expression and genome stability.Nat Rev Mol Cell Biol. 2020 Mar;21(3):167-178.
5. A.H. Youssef, et al., The balancing act of R-loop biology: The good, the bad, and the ugly, J. Biol. Chem. (2020) 295(4) 905–913.
6. Ginno, P. A., Lim, Y. W., Lott, P. L., Korf, I. & Chedin, F. GC skew at the 5’ and 3’ ends of human genes links R-loop formation to epigenetic regulation and transcription termination. Genome Res. 23, 1590–1600 (2013).
7. Nadel, J. et al. RNA:DNA hybrids in the human genome have distinctive nucleotide characteristics, chromatin composition, and transcriptional relationships. Epigenetics Chromatin 8, 46 (2015).
8. Ginno, P. A., Lott, P. L., Christensen, H. C., Korf, I. & Chedin, F. R- loop formation is a distinctive characteristic of unmethylated human CpG island promoters. Mol. Cell 45, 814–825 (2012)
9. Grunseich, C. et al. Senataxin mutation reveals how R- loops promote transcription by blocking DNA methylation at gene promoters. Mol. Cell 69, 426–437 (2018).
10. Arab, K. et al. GADD45A binds R- loops and recruits TET1 to CpG island promoters. Nat. Genet. 51, 217–223 (2019).
11. Chen, P. B., Chen, H. V., Acharya, D., Rando, O. J. & Fazzio, T. G. R loops regulate promoter- proximal chromatin architecture and cellular differentiation. Nat. Struct. Mol. Biol. 22, 999–1007 (2015).
12. Beckedorff, F. C. et al. The intronic long noncoding RNA ANRASSF1 recruits PRC2 to the RASSF1A promoter, reducing the expression of RASSF1A and increasing cell proliferation. PLOS Genet. 9, e1003705 (2013).
13. Wang et al., Induced ncRNAs allosterically modify RNA-binding proteins in cis to inhibit transcription. Nature 454 (2008)
14. Gibbons, H. R. et al. Divergent lncRNA GATA3–AS1 regulates GATA3 transcription in T- helper 2. Cells. Front. Immunol. 9, 2512 (2018).
15. Tan-Wong et al., R-Loops Promote Antisense Transcription across the Mammalian Genome. Molecular Cell 76, 600–616 (2019)
16. Boque- Sastre, R. et al. Head- to-head antisense transcription and R- loop formation promotes transcriptional activation. Proc. Natl Acad. Sci. USA 112, 5785–5790 (2015).
17. Sun, Q., Csorba, T., Skourti- Stathaki, K., Proudfoot, N. J. & Dean, C. R- loop stabilization represses antisense transcription at the Arabidopsis FLC locus. Science 340, 619–621 (2013).
18. Powella et al., R-loop formation at Snord116 mediates topotecan inhibition of Ube3a-antisense and allele-specific chromatin decondensation Proc. Natl Acad. Sci. USA 110, 13938-13943 (2015).
19. Ariel, F. et al. R-loop mediated trans action of the APOLO long noncoding RNA. Mol. Cell 77, 1055–1065.e4 (2020).
20. Luo H, et al., HOTTIP-dependent R-loop formation regulates CTCF boundary activity and TAD integrity in leukemia. Mol Cell. 17;82(4):833-851.e11. (2022).
21. Xu X et al: CircRNA inhibits DNA damage repair by interacting with host gene. Mol Cancer 19(1):128 (2020).