

背景:
5hmC- 哺乳动物DNA中的“第六碱基”
早在上世纪70年代,Penn等人首次发现在哺乳动物DNA中存在5-羟甲基化胞嘧啶(5hmC)(Penn et al., 1972)。然而该发现并未得到反复证实(Kothari and Shankar, 1976),因而5hmC也没有受到应有的重视。直到2009年,Kriaucionis 和Tahiliani两位科学家证实小鼠的Purkinje细胞,颗粒神经元以及胚胎干细胞存在5hmC (Kriaucionis and Heintz, 2009; Tahiliani et al., 2009)。此外,在哺乳动物的其他组织中同样检测到大量5hmC的存在 (Globisch et al., 2010)。其中5hmC在中枢神经和脊髓中含量最高(0.3-0.7%),在肾脏、鼻上皮细胞、膀胱、心脏、骨骼肌和肺部的含量居中(0.15 -0.17%),而肝脏、脾脏和内分泌腺中的含量最低(0.03-0.06%)。宋红军等人利用敏感度更高的5hmC化学标记法对HeLa和HEK293细胞检测发现,5hmC大约占总核苷酸的0.001% (Song et al., 2011)。
5甲基胞嘧啶转变为5羟甲基胞嘧啶的途径
DNA氧化损伤能产生各种修饰碱基,如鸟嘌呤可被氧化成为8-氧桥鸟嘌呤,5甲基胞嘧啶可被氧化生成5羟甲基胞嘧啶。因此,某些细胞中的5hmC很可能就是环境氧化压力所造成的。但是Kriaucionis和Heintz的发现表明5hmC的存在并未伴随有其他损伤产物的积累,并且Purkinje细胞和颗粒细胞中的5hmC总量与小鼠的年龄也不存在必然的联系(Kriaucionis and Heintz, 2009)。新近发现的TET蛋白家族使人们对5hmC有了更深入的了解:TET是一种能够催化5甲基胞嘧啶产生5羟甲基胞嘧啶的加氧酶。TET蛋白家族有3个成员,分别是Tet1,Tet2和Tet3。它们都含有一个序列保守的C-端催化结构域 (Ito et al., 2010; Tahiliani et al., 2009),并具有2-氧化戊二酸和2价铁原子依赖型双加氧酶的典型特征(Aravind and Koonin, 2001; Loenarz and Schofield, 2009)。
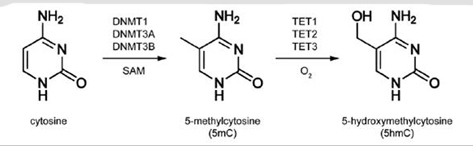
图释:哺乳动物DNA中的胞嘧啶种类。胞嘧啶是DNA复制的原料之一;5甲基胞嘧啶则是DNA甲基化转移酶DNMT以S-腺苷甲硫氨酸(SAM)为甲基供体催化胞嘧啶形成的;5羟甲基胞嘧啶是在TET蛋白作用下形成的,TET利用氧分子使羟基转移到5甲基胞嘧啶上。
5hmC的生物学作用
5hmC可影响长期和短期的基因表达调控,因此在体内可能具有重要的生物学意义。由于维持性甲基化酶DNMT1不能识别羟甲基化胞嘧啶,甲基化胞嘧啶所转化的羟甲基胞嘧啶可能有助于DNA进行被动去甲基化。维持性甲基化的保真度即使只发生微弱的降低就足以导致多次细胞分裂的过程中CpG的甲基化水平呈指数级降低,从而实现被动性去甲基化。
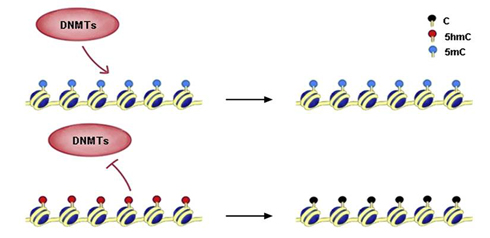
5hmC潜在的生物学功能-A:DNA甲基转移酶(DNMT)无法识别5hmC,因而5hmC在DNA复制过程中可以阻碍DNMT的维持性甲基化作用,最终导致DNA的被动去甲基化。
在光氧化实验或高pH条件下,5羟甲基胞嘧啶可通过去醛基产生胞嘧啶 (Privat and Sowers, 1996,Alegria, 1967; Flaks and Cohen, 1959),这使得在特定细胞环境中5羟甲基胞嘧啶有可能转变为胞嘧啶。一种可能性就是5羟甲基胞嘧啶作为主动去甲基化途径中的一个关键中间体存在,而这种去甲基化途径通常涉及到DNA的损伤修复机制(Tahiliani et al., 2009)。这种观点也得到了一些证据的支持:早在1988年,cannon等人就报道了牛胸腺提取物存在特异修饰5hmC的糖基化酶(Cannon et al., 1988)。此外,还有报道表明一些DNA糖基化酶如TDG和MBD4参与了DNA去甲基化过程,但目前体外酶活性试验尚未证实这些酶具有催化甲基胞嘧啶的活性。除此之外,hmC可以通过脱氨基作用生产去甲基化过程的中间产物hmU。因为在成纤维细胞中已经检测到了高活性的hmU:G的糖基化酶(Rusmintratip and Sowers, 2000)。
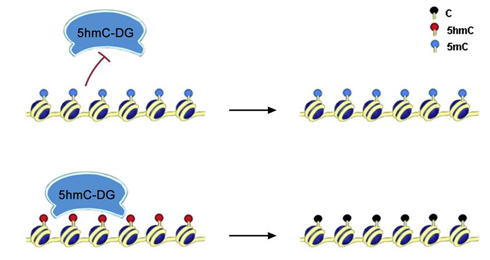
5hmC可能的生物学作用-B:5hmC可以被DNA修复酶识别,例如5hmC特异DNA糖基化酶(5hmC-DG)可以把羟甲基胞嘧啶转变为胞嘧啶,从而引起DNA的主动性去甲基化。
作为一个潜在稳定的碱基,5hmC也有可能通过募集5hmC结合蛋白或者排斥甲基化结合蛋白(MBPs)来影响染色质结构和转录活性。而实验结果也确实证明了甲基化结合蛋白MeCP2无法识别5hmC (Valinluck et al., 2004)。
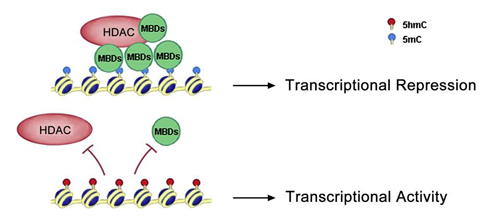
5hmC可能的生物学作用-C:由于5hmC不能被甲基化结合蛋白(MBD1,MBD2,MBD4,MeCP2)所识别,这样羟甲基化修饰的DNA就不能募集组蛋白去乙酰化酶,从而可以形成具有转录活性的染色质结构。
5hmC在基因组的分布以及与基因表达水平的关系:
基因体(gene body)上的5hmC及其与基因表达水平的关系
大量研究表明,5hmC特异性地富集在基因体上(Jin et al., 2011; Pastor et al., 2011; Song et al., 2011; Xu et al., 2011)。进一步深入的研究发现基因内部的5hmC含量往往可以表征基因表达的活性,这也与以前报道5hmC能够促进或维持基因表达的结论一致(Jin et al., 2011; Song et al., 2011)。在一些没有发生实质性去甲基化(5mC转变为5C)的过程中,5mC转换成5hmC可能是起到类似DNA去甲基化的作用,即可以解除5mC对基因表达的抑制效应(Wu and Zhang, 2010)。然而,基因体5hmC的水平与基因表达高低也不是简单的线性关系,Xu等人发现在组成型表达的基因如看家基因的基因体上5hmC含量很低。
启动子区的5hmC及其与基因表达水平的关系
Song等人观察到5hmC主要富集在如转录起始位点,转录终止位点和基因远端等区域的上游和下游(Song et al., 2011)。进一步的分析揭示基因附近富含5hmC意味着这个基因表达水平较高。Ficz 等人的实验结果也显示出启动子区5hmC的含量与转录水平具有明显的正相关性。而且发现这种关系部分依赖于启动子的CpG密度。5hmC似乎可以减轻部分5mC的沉默效应,因为启动子区含有5hmC和5mC的基因转录活性比启动子只有5mC的基因高。与这些观察相符的是,富含5hmC的启动子都有组蛋白激活的标志H3K4me3 ,而富含5mC的启动子缺乏H3K4me3 标志(Ficz et al., 2011)。另外有趣的是,随着胚胎干细胞分化,多能性相关基因包括Esrrb, Prdm14, Dppas, Klfz, TCL1 和ZFP43的启动子区5hmC水平显著下降,而5mC水平却明显增加(Ficz et al., 2011)。
二价结构域的5hmC及其与表达水平的关系
研究发现5hmC 在H3K27me3 和H3K4me3双修饰启动子的基因上有较高的富集度(Pastor et al., 2011)。均衡态(poised)的染色质往往也具有H3K27me3 和H3K4me3的二价修饰。另外Williams等人证明Tet1可以结合polycomb复合体的一大部分靶基因(Williams et al., 2011)。因此,胚胎干细胞中许多发育调控的基因所处的染色质都表现为均衡态,5hmC或许参与了染色质的这种均衡态的维持以及基因的转录调控。
增强子区的5hmC及其基因调控作用
在含有H3K4me1但缺少H3K4me3修饰的增强子区,5hmC的含量比5mC高,这暗示了5hmC与基因调控元件存在一定联系(Pastor et al., 2011)。Stroud等人观察到5hmC的存在与组蛋白发生H3K4me1 和 H3K27ac的修饰正相关,同时5hmC的含量反映了增强子的状态是处于激活还是静息状态。一般而言,5hmC含量高意味着增强子处于活化状态,反之亦然(Stroud et al., 2011)。
睾丸特异性基因启动子中5hmC的分布
在大脑DNA中,睾丸特异性基因的启动子区域一般都是高甲基化的。事实上,正是因为启动子区域发生高甲基化,所以睾丸特异性基因和其他生殖细胞特异表达的基因在体细胞通常是表达受抑制的 (De Smet et al., 1999),但是与高丰度的5mC不同的是,这些生殖相关基因中的5hmC含量几乎可以忽略不计。
胚胎干细胞和分化过程中的5hmC动态调控
由于染色质均衡态的构象及基因的组织特异性表达都与5hmC存在特定的联系,因此研究人员猜测5hmC的存在使一些特定的基因座能够快速响应适当的生物学信号刺激从而被激活。例如组织特异性基因的激活可能通过5mC去甲基化途径(5mC-5hmC-C)或者募集特异识别5hmC的转录调控因子来响应分化信号(Pastor et al., 2011)。又如,胚胎干细胞分化为拟胚体(EBs)过程中,多能性相关基因的启动子区的5hmC丰度下降,而这些位置的5mC丰度却在增加(Ficz et al., 2011)。此外,有人提出在产生诱导干细胞过程中,5hmC可以清除干性相关基因启动子区域的甲基化修饰 (Meissner et al., 2008)同时也能大规模地清除原生殖细胞的甲基化修饰(Iqbal et al., 2011; Ito et al., 2010)。
氧气感知与调控
Song等人发现基因中5hmC富集程度与低氧环境下的血管生成密切相关。Tet蛋白催化5mC变成5hmC的过程需要氧分子(Ito et al., 2010; Tahiliani et al., 2009)。HIF蛋白是哺乳动物系统中一个著名的氧气感知器,主要参与了缺氧诱导的血管生成等生理过程,而且研究表明HIF蛋白与Tet蛋白催化结构域均属于单铁双加氧超家族。因此,有人猜测在哺乳在动物细胞中,Tet蛋白氧化5mC成5hmC可能组成了一个新的氧气感受和调节通路。
受精卵细胞的表观重编程
哺乳动物的早期胚胎获得发育全能性时候就在表观基因组水平发生了大规模的重编程。而研究表明哺乳动物受精卵中的5hmC与表观重编程有密切联系。据报道,受精卵的雄原核会在基因组范围内发生DNA去甲基化事件,这种去甲基化又明显依赖于DNA的复制。Wosido等人发现伴随着5mC水平的下降,雄原核5hmC的含量却在逐渐地积累。这些雄原核的5hmC可能是由氧化酶Tet3氧化5mC产生的。
然而目前对5hmC在雄原核中的作用了解的还是不很清楚。5mC氧化生成5hmC的一个直接效应可能就是中和5mC对基因表达的抑制。我们知道,小鼠胚胎基因组的活化发生在两细胞时期,而那些在精子发生过程被DNA甲基化所抑制的许多基因如Oct4和Nanog都需要重新激活才能使得发育继续进行。5mC氧化生成5hmC后,5mC抑制蛋白不能够识别并结合该DNA序列,从而解除了5mC的抑制作用。(Jin et al., 2010; Valinluck et al., 2004)。另一种可能就是,5hmC替换5mC后就不再作为维持性甲基化酶DNMT1的底物(Valinluck and Sowers, 2007),这就意味着在早期胚胎的DNA复制过程中,即使DNMT酶存在活性,5hmC的形成也能够稀释DNA中甲基化CpG的含量。
神经元的发育和成熟
Song等人实验观察到小鼠小脑中的5hmC含量随发育的时期而增加。与出生7天后的小鼠小脑相比,成年小鼠的5hmC水平显著提高,这表明5hmC有可能直接参与了神经元的发育和成熟。
5hmC在疾病中的作用:
5hmC与神经退行性疾病
研究人员通过对衰老过程中含有5hmC的5425个基因进行GO(Gene Ontology)富集分析和信号通路(Pathway)富集分析发现,显著富集的信号通路都是与神经退行性失调,血管生成和缺氧应答过程密切相关。而且对富集基因列表的分析发现成年小鼠大约有15/23的基因发生了5羟甲基化修饰,而这些基因都是以前鉴定可能会引起小鼠和人的Purkinje细胞退化的(Lim et al., 2006)。总之,5hmC修饰经常发生在一些参与神经退行性失调的基因上面,因此5hmC可能对人神经失调疾病的发生有促进作用。
5hmC与癌症
实验表明,5hmC在结肠癌组织中的含量显著下降,甚至在结肠癌细胞株当中几乎无法检测到。多样本(38例结肠癌组织和8例正常结肠组织)的检测进一步证实结肠癌组织中的5hmC含量远低于正常组织。这些结果表明至少在结肠组织中,5hmC对肿瘤形成和发生可能具有负调控作用。其中可能的机制是DNA通过5hmC影响5mC的总量,所以结肠癌组织5hmC含量减少后将导致受到高度甲基化所抑制的肿瘤抑制基因和凋亡相关基因无法被重新激活,从而使这些肿瘤细胞免受凋亡基因或抑制基因的调控(Li and Liu, 2011)。
5hmC还可能直接参与了白血病的发生。TET2位于染色体的4q24,这个区域在脊髓发育不全病人中经常发生染色体缺失或重排(Viguie et al., 2005)。Tet2错义突变主要发生C-端包含2OGFeDO的结构域,进而干扰了Tet2催化5mC变成5hmC的活性(Ko et al., 2010)。与正常样本相比,TET2突变的急性髓性白血病人的骨髓样本中5hmC含量非常低(Ko et al., 2010),所有这些结果表明5hmC可能参与急性髓性白血病的发生。
参考文献:
Alegria, A.H. (1967). Hydroxymethylation of pyrimidine mononucleotides with formaldehyde. Biochim Biophys Acta 149, 317-324.
Aravind, L., and Koonin, E.V. (2001). The DNA-repair protein AlkB, EGL-9, and leprecan define new families of 2-oxoglutarate- and iron-dependent dioxygenases. Genome Biol 2, RESEARCH0007.
Cannon, S.V., Cummings, A., and Teebor, G.W. (1988). 5-Hydroxymethylcytosine DNA glycosylase activity in mammalian tissue. Biochem Biophys Res Commun 151, 1173-1179.
De Smet, C., Lurquin, C., Lethe, B., Martelange, V., and Boon, T. (1999). DNA methylation is the primary silencing mechanism for a set of germ line- and tumor-specific genes with a CpG-rich promoter. Mol Cell Biol 19, 7327-7335.
Ficz, G., Branco, M.R., Seisenberger, S., Santos, F., Krueger, F., Hore, T.A., Marques, C.J., Andrews, S., and Reik, W. (2011). Dynamic regulation of 5-hydroxymethylcytosine in mouse ES cells and during differentiation. Nature 473, 398-402.
Flaks, J.G., and Cohen, S.S. (1959). Virus-induced acquisition of metabolic function. I. Enzymatic formation of 5-hydroxymethyldeoxycytidylate. J Biol Chem 234, 1501-1506.
Globisch, D., Munzel, M., Muller, M., Michalakis, S., Wagner, M., Koch, S., Bruckl, T., Biel, M., and Carell, T. (2010). Tissue distribution of 5-hydroxymethylcytosine and search for active demethylation intermediates. PLoS One 5, e15367.
Iqbal, K., Jin, S.G., Pfeifer, G.P., and Szabo, P.E. (2011). Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine. Proc Natl Acad Sci U S A 108, 3642-3647.
Ito, S., D'Alessio, A.C., Taranova, O.V., Hong, K., Sowers, L.C., and Zhang, Y. (2010). Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification. Nature 466, 1129-1133.
Jin, S.G., Kadam, S., and Pfeifer, G.P. (2010). Examination of the specificity of DNA methylation profiling techniques towards 5-methylcytosine and 5-hydroxymethylcytosine. Nucleic Acids Res 38, e125.
Jin, S.G., Wu, X., Li, A.X., and Pfeifer, G.P. (2011). Genomic mapping of 5-hydroxymethylcytosine in the human brain. Nucleic Acids Res 39, 5015-5024.
Kangaspeska, S., Stride, B., Metivier, R., Polycarpou-Schwarz, M., Ibberson, D., Carmouche, R.P., Benes, V., Gannon, F., and Reid, G. (2008). Transient cyclical methylation of promoter DNA. Nature 452, 112-115.
Ko, M., Huang, Y., Jankowska, A.M., Pape, U.J., Tahiliani, M., Bandukwala, H.S., An, J., Lamperti, E.D., Koh, K.P., Ganetzky, R., et al. (2010). Impaired hydroxylation of 5-methylcytosine in myeloid cancers with mutant TET2. Nature 468, 839-843.
Kothari, R.M., and Shankar, V. (1976). 5-Methylcytosine content in the vertebrate deoxyribonucleic acids: species specificity. J Mol Evol 7, 325-329.
Kriaucionis, S., and Heintz, N. (2009). The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain. Science 324, 929-930.
Li, W., and Liu, M. (2011). Distribution of 5-hydroxymethylcytosine in different human tissues. J Nucleic Acids 2011, 870726.
Lim, J., Hao, T., Shaw, C., Patel, A.J., Szabo, G., Rual, J.F., Fisk, C.J., Li, N., Smolyar, A., Hill, D.E., et al. (2006). A protein-protein interaction network for human inherited ataxias and disorders of Purkinje cell degeneration. Cell 125, 801-814.
Loenarz, C., and Schofield, C.J. (2009). Oxygenase catalyzed 5-methylcytosine hydroxylation. Chem Biol 16, 580-583.
Meissner, A., Mikkelsen, T.S., Gu, H., Wernig, M., Hanna, J., Sivachenko, A., Zhang, X., Bernstein, B.E., Nusbaum, C., Jaffe, D.B., et al. (2008). Genome-scale DNA methylation maps of pluripotent and differentiated cells. Nature 454, 766-770.
Metivier, R., Gallais, R., Tiffoche, C., Le Peron, C., Jurkowska, R.Z., Carmouche, R.P., Ibberson, D., Barath, P., Demay, F., Reid, G., et al. (2008). Cyclical DNA methylation of a transcriptionally active promoter. Nature 452, 45-50.
Pastor, W.A., Pape, U.J., Huang, Y., Henderson, H.R., Lister, R., Ko, M., McLoughlin, E.M., Brudno, Y., Mahapatra, S., Kapranov, P., et al. (2011). Genome-wide mapping of 5-hydroxymethylcytosine in embryonic stem cells. Nature 473, 394-397.
Penn, N.W., Suwalski, R., O'Riley, C., Bojanowski, K., and Yura, R. (1972). The presence of 5-hydroxymethylcytosine in animal deoxyribonucleic acid. Biochem J 126, 781-790.
Privat, E., and Sowers, L.C. (1996). Photochemical deamination and demethylation of 5-methylcytosine. Chem Res Toxicol 9, 745-750.
Rusmintratip, V., and Sowers, L.C. (2000). An unexpectedly high excision capacity for mispaired 5-hydroxymethyluracil in human cell extracts. Proc Natl Acad Sci U S A 97, 14183-14187.
Song, C.X., Szulwach, K.E., Fu, Y., Dai, Q., Yi, C., Li, X., Li, Y., Chen, C.H., Zhang, W., Jian, X., et al. (2011). Selective chemical labeling reveals the genome-wide distribution of 5-hydroxymethylcytosine. Nat Biotechnol 29, 68-72.
Stroud, H., Feng, S., Morey Kinney, S., Pradhan, S., and Jacobsen, S.E. (2011). 5-Hydroxymethylcytosine is associated with enhancers and gene bodies in human embryonic stem cells. Genome Biol 12, R54.
Tahiliani, M., Koh, K.P., Shen, Y., Pastor, W.A., Bandukwala, H., Brudno, Y., Agarwal, S., Iyer, L.M., Liu, D.R., Aravind, L., et al. (2009). Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science 324, 930-935.
Valinluck, V., and Sowers, L.C. (2007). Endogenous cytosine damage products alter the site selectivity of human DNA maintenance methyltransferase DNMT1. Cancer Res 67, 946-950.
Valinluck, V., Tsai, H.H., Rogstad, D.K., Burdzy, A., Bird, A., and Sowers, L.C. (2004). Oxidative damage to methyl-CpG sequences inhibits the binding of the methyl-CpG binding domain (MBD) of methyl-CpG binding protein 2 (MeCP2). Nucleic Acids Res 32, 4100-4108.
Viguie, F., Aboura, A., Bouscary, D., Ramond, S., Delmer, A., Tachdjian, G., Marie, J.P., and Casadevall, N. (2005). Common 4q24 deletion in four cases of hematopoietic malignancy: early stem cell involvement? Leukemia 19, 1411-1415.
Williams, K., Christensen, J., Pedersen, M.T., Johansen, J.V., Cloos, P.A., Rappsilber, J., and Helin, K. (2011). TET1 and hydroxymethylcytosine in transcription and DNA methylation fidelity. Nature 473, 343-348.
Wu, S.C., and Zhang, Y. (2010). Active DNA demethylation: many roads lead to Rome. Nat Rev Mol Cell Biol 11, 607-620.
Xu, Y., Wu, F., Tan, L., Kong, L., Xiong, L., Deng, J., Barbera, A.J., Zheng, L., Zhang, H., Huang, S., et al. (2011). Genome-wide Regulation of 5hmC, 5mC, and Gene Expression by Tet1 Hydroxylase in Mouse Embryonic Stem Cells. Mol Cell 42, 451-464.
Zhu, B., Zheng, Y., Hess, D., Angliker, H., Schwarz, S., Siegmann, M., Thiry, S., and Jost, J.P. (2000). 5-methylcytosine-DNA glycosylase activity is present in a cloned G/T mismatch DNA glycosylase associated with the chicken embryo DNA demethylation complex. Proc Natl Acad Sci U S A 97, 5135-5139.