

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
Small RNA测序已被用于分析microRNA和其它small RNA。然而,RNA修饰的干扰、文库扩增的偏差、数据分析不准确….等诸多问题限制了Small RNA测序的效率和准确性。Arraystar Small RNA芯片能有效解决这些挑战,在总RNA需求量更低的情况下,依旧以高灵敏度和准确度同时分析全谱small RNA。
通过测序进行small RNA定量检测的挑战
·来自RNA修饰的干扰
在small RNA测序文库构建过程中,small RNA内部的修饰(例如m1A、m3C和m1G)会阻碍反转录反应进而中止cDNA合成,导致获得的序列偏向启动端,最终导致构建无偏倚全长序列文库的失败。例如,由于tRF&tiRNA TΨC环区域中存在m1A修饰的阻碍,small RNA-seq通常识别18nt 3'tRF&tiRNA,但遗漏了由Northern Blot检测到的更主要的22nt tRF&tiRNA异构体。
·文库扩增的偏差
在small RNA定量过程中,为了产生足够量的cDNA进入测序仪,small RNA测序需要对文库进行PCR扩增[1]。然而,如图1所示,RNA的G/C含量、序列组成、二级结构、长度、PCR的启动以及反应条件都可能导致PCR产物丰度的偏差和扭曲[2]。因此,small RNA测序并不能对RNA进行有效的绝对定量,其结果还需使用正交方法进行验证。
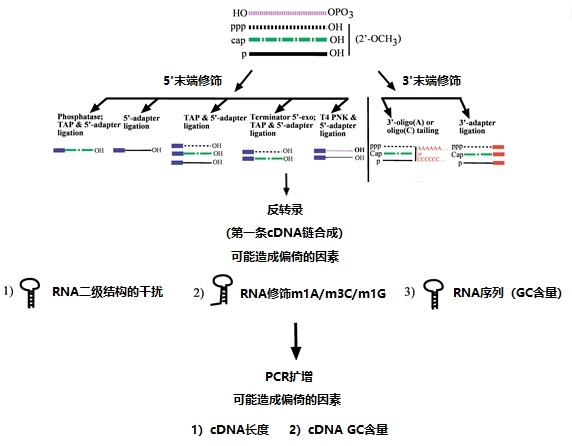
图 1. cDNA构建所涉及步骤的图示,包括潜在的偏差来源。 (A) 具有不同5’ 端和3’ 端修饰的非蛋白质编码RNA的起始池,由不同的线型示意性表示。 (B) 左图描述了在RNA 5’ 端连接之前的不同酶预处理,以富集不同的RNA亚型。在各自的5’ 端预处理后可用于接头连接的RNA类别在每个预处理下示意性表示。右图描述了可用于3’ 端(-oligo(A)或-oligo(C)尾)和接头连接的RNA类别的亚型。 (C)与RNA 5’(左)和3’(右)末端修饰以及cDNA构建的后续步骤相关的可能偏差
·采用TPM衡量small RNA表达水平的潜在问题
在small RNA测序分析中,不同的实验条件或不同研究的数据集中,small RNA整体表达水平变化很大,这导致使用TPM值衡量small RNA在样本间的表达差异存在偏差[3-4]。如图2[5]所示,在其它RNA的绝对丰度没有变化的情况下,一个RNA的绝对丰度变化,就会使得其它所有RNA的TPM值成比例变化。TPM值完全依赖于样本中的RNA组成,且少数高表达的基因表达变化就可以影响整体TPM定量的准确性。
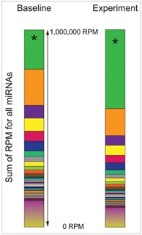
图2 .miRNA测序的TPM值堆积图。除了*代表的RNA,其它所有miRNA在Baseline和Experiment条件下表达不变。然而,*代表的RNA丰度在Experiment条件下上升至2倍时,会同时导致其它所有miRNA的TPM值相应下降,即使这些miRNA的绝对表达水平没有变化[5]
·样本要求量高
对于tRNA和tRF&tiRNA测序,在文库构建之前,目标RNA的分离和预处理需要5 ug甚至更多的总RNA 量[6],这使得样品量有限的研究项目更加难以进行。
多种Small RNA同时准确定量的解决方案
Arraystar small RNA芯片结合直接末端标记和巧妙的探针设计微阵列技术,有效弥补了small RNA测序的各项短板,在一张芯片上可同时检测和量化包括miRNA、pre-miRNA、tRF&tiRNA、tRNA和snoRNA在内的小RNA,为研究疾病中small RNA的调控功能和挖掘生物标志物潜力提供重要的表达信息。与small RNA测序相比,该芯片具有以下优势:
• 提高Small RNA表达谱的门槛,达到高灵敏度、特异性和准确性。实验流程简单直接,克服了small RNA-seq中由于RNA修饰、RNA二级结构、反转录阻碍、PCR扩增和数据分析不准确造成的偏差。
• 所需的RNA样本量低至100ng,为许多研究创造了机会。
• 可以检测低质量RNA样本,如降解的RNA、血清/血浆等体液RNA、FFPE RNA。
参考文献:
[1] Kapranov, P., et al. (2012) Methods Mol Biol [PMID: 22144202]
[2] Dabney, J. and Meyer, M. (2012) Biotechniques [PMID: 22313406]
[3] Zhao, Y., et al. (2021) J Transl Med [PMID: 34158060]
[4] Zhao, S., et al. (2020) RNA [PMID: 32284352]
[5] Witwer, K. W. and Halushka, M. K. (2016) RNA Biol [PMID: 27645402]
[6] Shi, J., et al. (2021) Nat Cell Biol [PMID: 33927371]

