|
单细胞测序技术服务 单细胞全转录组测序 |
|
NGS测序技术服务 DRIP-seq |
|
基因芯片技术服务 Small RNA表达芯片 Small RNA修饰芯片 m6A单碱基分辨率芯片 mRNA&lncRNA表观转录组芯片 circRNA表观转录组芯片 |
NGS测序技术服务 RNA m6A甲基化测序(MeRIP Seq) |
LC-MS mRNA碱基修饰检测 tRNA碱基修饰检测 |
PCR技术服务 MeRIP-PCR技术服务 m6A绝对定量RT-PCR技术服务 m6A单碱基位点PCR(MazF酶切法)技术服务 |
|
基因芯片技术服务 DNA甲基化芯片 DNA羟甲基化芯片 ChIP-chip |
NGS测序技术服务 DNA甲酰基胞嘧啶(5fc)修饰测序 DNA 5hmC 测序(化学法) DNA甲基化测序 DNA羟甲基化测序 染色质免疫共沉淀测序 |
PCR技术服务 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR |
|
蛋白相对定量 TMT标记定量技术 非标定量技术 |
蛋白修饰 TMT标记定量磷酸化 非标定量磷酸化 |
RNA/蛋白-蛋白相互作用 RNA-蛋白相互作用 蛋白-蛋白相互作用 |
相关服务
Small RNA芯片 tsRNA(tRF&tiRNA)测序相关产品
相关资源
Arraystar Small RNA芯片结合直接末端标记和巧妙的探针设计技术,实现对主要small RNA类群,包括miRNAs、pre-miRNAs、tsRNA、tRNAs和snoRNAs的同时检测与精确定量,为研究疾病中small RNA的调控功能和挖掘生物标志物潜力提供重要的表达信息。
芯片优点
• 同时检测主要的small RNA类群:miRNA、pre-miRNA、tRNA、tsRNA和snoRNA
• 直接末端标记和巧妙的探针设计,提高small RNA检测的灵敏度、特异性和准确性
• 直接和简化的实验步骤: 克服在small RNA测序中存在的RNA修饰、RNA折叠阻碍、逆转录阻断、PCR扩增偏倚以及分析不准确的问题
• 适用于低质量RNA样本:如降解RNA、血清/血浆/体液RNA、FFPE RNA
• 所需的RNA样本量低至100 ng
Arraystar Small RNA表达芯片列表
| 服务名称 | 描述 | 规格 |
|---|---|---|
| Arraystar Human Small RNA 表达芯片 | 定量miRNA,pre-miRNA,snoRNA,tRNA & tsRNA | 8 x 15K |
| Arraystar Mouse Small RNA 表达芯片 | 定量miRNA,pre-miRNA,snoRNA,tRNA & tsRNA |
8 x 15K |
通过测序进行small RNA定量检测的挑战
Small RNA测序已被用于分析microRNA和其他small RNA。然而,利用small RNA测序数据量化的small RNA的相对丰度与qPCR / Northern Blot结果不一致。例如,通过small RNA-seq测得的miR-143的水平比miR-145高40倍,但通过qPCR或Northern Blot测得它们的表达水平相当 ADDIN EN.CITE ADDIN EN.CITE.DATA [8-10]。Small RNA测序的不准确性主要是由于small RNA测序文库制备和数据分析过程的偏差对结果造成的误导 ADDIN EN.CITE ADDIN EN.CITE.DATA [1,2],在某些情况下,这些与真实丰度的偏差可高达10,000倍 ADDIN EN.CITE ADDIN EN.CITE.DATA [3-8]。
•来自RNA修饰的干扰
在测序文库构建和small RNA定量过程中,small RNA内部的修饰(例如m1A、m3C和m1G)会阻碍反转录反应进而中止cDNA合成,导致获得的序列偏向启动端,最终导致构建无偏倚全长序列文库的失败。例如,由于tsRNA TΨC环区域中存在m1A修饰的阻碍,small RNA-seq通常识别18nt 3'tsRNA,但遗漏了由Northern Blot检测到的更主要的22nt tsRNA异构体。
•文库扩增的偏差
为了产生足够的cDNA量进入测序仪,small RNA测序需要对文库进行PCR扩增[9]。然而,RNA的G/C含量、序列组成、二级结构、长度、PCR的启动以及反应条件都可能导致PCR产物丰度的偏差和扭曲。也就是说,当转录组中的不同模板在PCR反应中一起扩增时,某些模板的优先或阻滞扩增总是会导致获得的产物无法呈现真实的RNA丰度[10]。因此,需要多轮PCR扩增的small RNA测序定量从来都不能对RNA进行有效的绝对定量,其结果需要使用正交方法进行验证。
•采用TPM衡量small RNA表达水平的潜在问题
在small RNA测序中,TPM(Reads Per Million reads)值是代表small RNA 转录本的相对丰度的指标,即测到的样本中总体small RNA的reads数标准化至1百万后,每个small RNA的reads数占比。
然而,如(图1)[1]所示,即使其他RNA的绝对丰度没有变化,一个RNA的绝对丰度变化,就会使得其他所有RNA的TPM值成比例变化。因此,TPM值完全依赖于样本中的RNA组成,且少数高表达的基因表达变化就可以影响整体TPM定量的准确性。实际上,在不同的实验条件或不同研究的数据集中,small RNA整体表达水平变化很大,导致使用TPM值比较small RNA在不同样本间的表达差异存在偏差[11,12]。
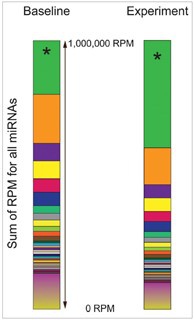
图1 miRNA测序的TPM值堆积图。除了*代表的RNA,其他所有miRNA在Baseline和Experiment条件下表达不变。然而,*代表的RNA丰度在Experiment条件下上升至2倍时,会同时导致其他所有miRNA的TPM值相应下降,即使这些miRNA的绝对表达水平没有变化[1]。
•样本要求量高
对于tRNA和tsRNA测序,在文库构建之前,目标RNA的分离和预处理需要5 ug甚至100 ug的总RNA [13],这使得样品量有限的研究项目难以进行。
Small RNA准确定量的解决方案
为了克服这些挑战,Arraystar设计了Arraystar Small RNA芯片。结合末端直接标记和巧妙的探针设计技术,克服了small RNA测序的诸多问题,在需要的总RNA量更少的情况下,依旧能够作为一种实用且有效的解决方案以高灵敏度和准确度分析全谱small RNA。
•提高了small RNA分析的高灵敏度、特异性和准确性的标准
通过末端直接标记法,small RNA通过T4 RNA连接酶直接将pCp-Cy3连接到3 '末端,在这一过程中每一个RNA分子都被一个Cy3标签所标记。这种方法消除了small RNA-seq中逆转录合成cDNA时由于RNA修饰干扰和RNA折叠阻碍而产生的偏差;避免了测序文库PCR扩增的偏好性;利用DMSO来减少small RNA存在的RNA结构和序列差异的影响。所有这些都有助于维持天然small RNA水平的保真度,从而实现比RNA-seq甚至qPCR更精确的无偏倚定量效果。
巧妙的探针设计采用5'发夹结构和标准化的序列靶向区域,特异性区分只有1~2个核苷酸差异的small RNA。此外,高亲和力探针杂交确保了即使是低丰度的small RNA检测也具有非常高的灵敏度。
•低样本RNA量要求
Arraystar Small RNA芯片需要的样本总RNA量低至100 ng,这比small RNA-seq所需的总RNA量要低一个数量级。由于其采用直接末端化学标记的方法,不需要进行RNA预处理从而避免了RNA损失,因此该芯片显著降低了RNA起始量的需求,特别是对于高度修饰的RNA分子类型(例如tRNA和tsRNA)。这一样品量要求低的特点,为样品稀有或供应有限的研究项目提供了机会。
•适用于低质量的样本
直接的末端标记对底物RNA序列中的核苷酸损伤相对不敏感,因为它不依赖于通过逆转录扩增的cDNA进行检测。此外,虽然芯片探针不受不相关序列存在的影响,但来自降解RNA样品中丰富rRNA的RNA片段可以污染目标RNA长度范围内的small RNA,从而降低small RNA测序结果中对small RNA的覆盖率。
由于这些原因,利用small RNA芯片检测保存、化学处理或降解的样品,例如血清/血浆/体液/FFPE RNA等是特别有利的。
•同时分析多个small RNA类群
通过测序分析不同的small RNA类群需要单独的测序方法和实验:miRNA-seq,tRNA-seq,tsRNA-seq和用于较长的snoRNA和small RNA前体的常规RNA-seq。Arraystar small RNA芯片使用统一的化学标记并与同一个芯片上的不同探针杂交,一次实现对所有主要的small RNA类群检测。
参考文献
[1] Witwer, K. W. and Halushka, M. K. (2016) RNA Biol [PMID: 27645402]
[2] Raabe, C. A., et al. (2014) Nucleic Acids Res [PMID: 24198247]
[3] Fuchs, R. T., et al. (2015) PLoS One [PMID: 25942392]
[4] Sorefan, K., et al. (2012) Silence [PMID: 22647250]
[5] Hafner, M., et al. (2011) RNA [PMID: 21775473]
[6] McCormick, K. P., et al. (2011) Silence [PMID: 21356093]
[7] Tian, G., et al. (2010) BMC Biotechnol [PMID: 20815927]
[8] Linsen, S. E., et al. (2009) Nat Methods [PMID: 19564845]
[9] Kapranov, P., et al. (2012) Methods Mol Biol [PMID: 22144202]
[10] Dabney, J. and Meyer, M. (2012) Biotechniques [PMID: 22313406]
[11] Zhao, Y., et al. (2021) J Transl Med [PMID: 34158060]
[12] Zhao, S., et al. (2020) RNA [PMID: 32284352]
[13] Shi, J., et
al. (2021) Nat Cell Biol [PMID: 33927371]
Arraystar Human Small RNA 表达芯片 V1.0
| 探针总数 | 14,707 |
|---|---|
| 探针设计原理 | 全长探针包括5’端帽子序列、与smallRNA互补配对的特异性结合序列及3’linker序列 |
| 探针位置 |
5-p-miRNAs and 5'tsRNAs: 针对small RNA的3’端序列设计 3-p-miRNAs and 3'tsRNAs: 针对small RNA的5’端序列设计 Pre-miRNA: 针对前体miRNA的环序列设计 tRNA: 针对成熟tRNA的反密码子环序列设计 snoRNA:针对snoRNA的特异性序列区域设计 |
| 探针特异性 | Small RNA高特异性探针 |
| miRNA | 2,627 (1,318个5-p-miRNA, 1,309个3-p-miRNA) |
| tsRNA | 4254 |
| miRNA前体 | 1745 |
| tRNA成熟体 | 346 |
| snoRNA | 955 |
| Small RNA来源 |
miRNA: miRBase(v22) tsRNA: tRFdb, MINTbase, GtRNADb (Updated to 18.1 2019.08) pre-miRNA: miRBase(v22) tRNA:GtRNADb(更新至18.1 2019.08), ENSEMBL(v99) snoRNA:ENSEMBL(v99) 发表的高分文章 |
| 芯片规格 | 8 x 15K |
Arraystar Mouse small RNA 表达芯片 V1.0
| 探针总数 | 14,895 |
|---|---|
| 探针设计原则 | 全长探针包括5’端帽子序列、与smallRNA互补配对的特异性结合序列及3’linker序列 |
| 探针设计位置 |
5-p-miRNAs and 5'tsRNAs: 针对small RNA的3’端序列设计 3-p-miRNAs and 3'tsRNAs:针对small RNA的5’端序列设计 Pre-miRNA:针对前体miRNA的环序列设计 tRNA:针对成熟tRNA的反密码子环序列设计 snoRNA:针对snoRNA的特异性序列区域设计 |
| 探针特异性 | small RNA高特异性探针 |
| miRNA | 1949 (966个5-p-miRNA, 983个3-p-miRNA) |
| tsRNA | 1809 |
| miRNA前体 | 1,122 |
| tRNA成熟体 | 270 |
| snoRNA | 1,323 |
| Small RNA来源数据库 |
miRNA: miRBase (v22) tsRNA: tRFdb, GtRNAdb(Updated to 18.1 2019.08) pre-miRNA:miRBase(v22) tRNA:GtRNAdb(更新至18.1 2019.08), ENSEMBL(v99) snoRNA:ENSEMBL(v99) 发表的高分文章 |
| 芯片规格 | 8 x 15K |
对于每种small RNA类群,数据分析结果包括检测指标的表达定量值、统计学分析、注释信息和可供发表的高质量图表。
•差异表达分析(以tsRNA和tRNA为例)
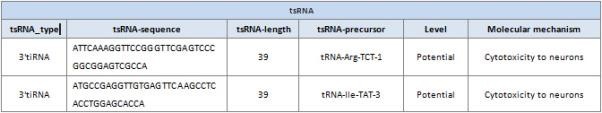
tsRNA_type: tsRNA类型(tsRNA-5, tsRNA-3, tsRNA-1, 5-Leader, 5-tiRNA, 3-tiRNA, and i-tsRNA)。
tsRNA-sequence: tsRNA序列。
tsRNA-length: tsRNA长度。
tsRNA-precursor: tsRNA前体的基因名。
Level: tsRNA的可信度。
Functional–文献报道的具有特定生物学功能或疾病关联性;
Reliable–tsRNAdb收录或文献报道,但未做深入功能机制研究;
Potential-由Arraystar根据RNA片段长度和tRNA中的切割位置预测的tsRNA。
Mechanism: tsRNA的分子机制。

Sequence: tRNA isodecoder的序列。
Gene name: tRNA isodecoder的基因名。
GenomeLocus: tRNA isodecoder在基因组上的定位。
tRNA promoter Locus: tRNA isodecoder的启动子在基因组上的定位。tRNA promoter–包括一个tRNA的基因位置加上基因上游100bp的序列(PMC6108506)。
pre-tRNA locus: tRNA isodecoder的前体在基因组上的定位。pre-tRNA – tRNA前体包括一个成熟tRNA加上基因上游100bp序列及3’trailer序列。
tRNA neighboring gene: tRNA isodecoder最邻近的基因名。
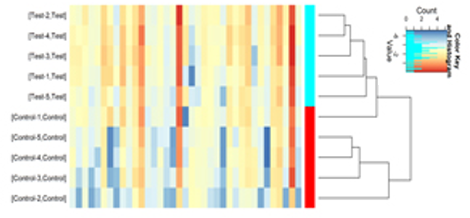
•差异表达Small RNAs的层次聚类热图
|
图4 差异表达的miRNA的层次聚类热图。 |