

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
相关服务
circRNA-qPCR circRNA表观转录组芯片相关产品
相关资源
为什么circRNA芯片比RNA-seq更适合于circRNA的表达分析? IF27.7!客户文章|Arraystar circRNA芯片助力结直肠癌抗PD-1治疗耐药性的分子机制研究 环状RNA的研究意义 喜报!Cell发表Arraystar circRNA芯片客户文章! 客户成果丨 影响因子23!circRNA促进缺血性脑卒中动物模型的功能修复 【SCI 22分客户成果】circRNA抑制由DNA感受器cGAS介导的长期造血干细胞耗竭 客户文章|揭示环状RNA作为心脏炎症调控与组织修复关键因子的全新机制 芯片更适用于低丰度RNA分析环状RNA(circRNA)是一类由特殊剪接机制形成的、具有闭合环状结构、大量存在于真核转录组中的非编码RNA,也是目前生命科学和医学领域的研究热点分子。circRNA分子的组织特异性、疾病特异性、时序特异性及高稳定性等特征,使得circRNA作为临床疾病的biomarker具有明显的优势。近来研究显示,环状RNA在不同物种中起到miRNA海绵的作用,称之为竞争性内源RNA(ceRNA),即能竞争性结合miRNA。而与疾病关联性miRNA的相互作用说明环状RNA对疾病的调控起着非常重要的作用。此外,一些内含子类型的环状RNA(ciRNA)会促进宿主基因的转录。
为了能够更好地研究circRNA,美国Arraystar公司在首款circRNA芯片基础上迅即升级版本为V2.0。其circRNA来源融合了环状RNA研究的高水平文献,所有cicrRNA都经过了严谨的实验验证,以便于对不同生理及病理条件下的circRNA进行系统的研究。同时我们对所有差异表达的circRNA用高匹配值的miRNA靶标位点进行了标注,这将有利于对circRNA作为天然miRNA海绵功能的进行研究。研究者使用该款芯片发表的超过350篇高水平SCI文章,点击此处了解。
芯片特点:
▪ 实用的高灵敏度和特异性的circRNA表达谱检测平台。
▪ 针对环状反向结合位点的特异性探针、线性RNA去除和高效的circRNA标记,确保circRNA 表达谱检测的高特异性、高准确度和高可信度。
▪ 针对circRNA生物学功能的详细注释信息,例如,miRNA结合位点,miRSVR 得分以及保守性,方便研究circRNA作为miRNA sponge的功能。
▪ 基于circRNA特殊的分子特征,芯片是优于测序的高通量检测选择。了解更多>>
Aksomics(原康成生物)是Arraystar公司中国地区唯一代理商,为您提供一站式circRNA芯片技术服务,您只需要提供保存完好的组织或细胞标本,Aksomics的技术服务人员就可为您完成全部实验操作,并提供完整的实验报告。
Arraystar公司circRNA芯片产品列表
| 服务 | 芯片 | 规格 | 描述 |
|---|---|---|---|
| Human circular RNA Microarray Service | Human circular RNA Array V2.0 | 8x15K | 13,617 human circular RNAs |
| Mouse circular RNA Microarray Service | Mouse circular RNA Array V2.0 | 8×15K | 14,236 Mouse circular RNAs |
| Rat circular RNA Microarray Service | Rat circular RNA Array V2.0 | 8×15K | 14,145 Rat circular RNAs |
可以很好区分对应的线性分子,确保circRNA分子检测的可靠性和准确度(图1)。
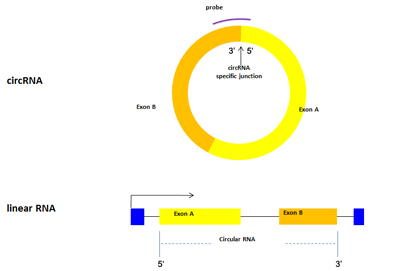
图1. Arraystar circRNA芯片 V2.0使用针对circRNA反向剪接位点设计探针进行检测,可以准确识别并定量单个circRNA的表达,并很好区分其对应的线性转录本。上图中的线性RNA可以通过可变剪切,其中一部分外显子环化形成上图中circRNA,检测circRNA的特异性探针靶向结合外显子B的3’端和外显子A的5’端连接的环化位点。
▪ 详细的circRNA-miRNA 注释信息
Arraystar circRNA芯片结果注释里提供circRNA分子上潜在的miRNA结合位点有助于探索其miRNA sponge的功能(图2)。
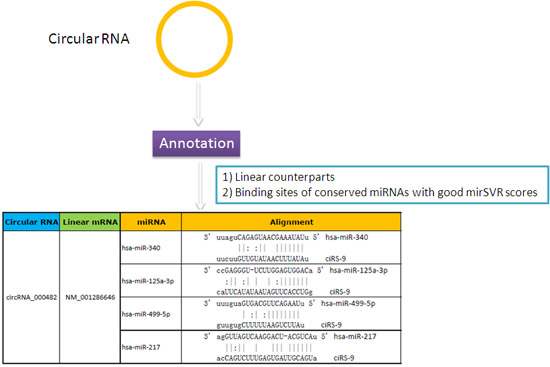
图2.circRNA与保守的miRNA结合位点信息的详细注释
▪ circRNA芯片更适合于circRNA的检测:鉴于circRNA分子的特性,circRNA芯片是比RNA测序更合适的方式 获取更多 >
1)比对到circRNA反向剪接位点附近序列仅占了circRNA数据量很少一部分的,远少于同等丰度的线性转录本。
2)对circRNA 定性仅需要很少的counts,但表达值的准确定量需要数百个counts以上。
3)结合大量测序数据和研究,大部分已知的circRNA仅检测到很少的counts,不能有效进行差异表达的分析(图3)。
简而言之,CircRNA测序适用于发现新的circRNA分子,但对于大部分的circRNA,测序无法准确分析其表达差异。
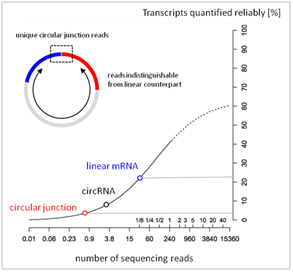
图3.定量可靠的转录本比例与测序深度关系图。 RNA测序中一般产生低于30M 的mRNA reads(蓝色的点),小于0.5M 的circRNA junction reads(红色的点)。能够准确定量的circRNA 少于5%。(Adopted from Labaj et al, (2011) Bioinformatics [PMID 21685096]. )
▪ 优质的检测结果
灵敏度更高:芯片检测范围跨越5个数量级,可以准确定量低丰度的circRNA(图4)。

图4:Arraystar circRNA芯片 可以准确检测5个数量级范围内的动态表达。
高重复性:Arraystar circRNA芯片的技术重复数据显示了高相关性(R2>0.9) (图5)
图5. Arraystar circRNA芯片的高重复性
Human Database
| 探针总数 | 13,617 |
| 探针长度 | 60 nt |
| 探针设计 | 靶向circRNA特异性反向剪接位点 |
| 探针特异性 | 转录本特异性 |
| 标记方法 | 随机引物+样本RNase R前处理确保circRNA被特异性且高效的标记Random primer labeling coupled with RNase R sample pretreatment to ensure specific and efficient labeling of circular RNAs. |
| Circular RNA 来源 | |
| Salzman's circRNAs [4] | 8,529 |
| Memczak's circRNAs [3] | 1,601 |
| Zhang's circRNAs [6] | 93 |
| Zhang's circRNAs [5] | 4,980 |
| Jeck's circRNAs [2] | 3,769 |
| Guo's circRNAs [1] | 5,536 |
| 芯片规格 | 8 * 15K |
Mouse Database
| 探针总数 | 14,236 |
| 探针长度 | 60 nt |
| 探针设计 | 靶向circRNA特异性反向剪接位点 |
| 探针特异性 | 转录本特异性 |
| 标记方法 | 随机引物+样本RNase R前处理确保circRNA被特异性且高效的标记Random primer labeling coupled with RNase R sample pretreatment to ensure specific and efficient labeling of circular RNAs. |
| CircRNA 来源 | |
| Memczak's circRNAs | 1,750 |
| Guo's circRNAs | 570 |
| You's circRNAs | 13,300 |
| 芯片规格 | 8 * 15K |
Rat Database
| 探针总数 | 14,145 |
| 探针长度 | 60 nt |
| 探针设计 | 靶向circRNA特异性反向剪接位点 |
| 探针特异性 | 转录本特异性 |
| 标记方法 | 随机引物+样本RNase R前处理确保circRNA被特异性且高效的标记Random primer labeling coupled with RNase R sample pretreatment to ensure specific and efficient labeling of circular RNAs. |
| CircRNA来源 | |
| You Xintian's circRNAs [7] | 12,298 |
| Mouse circRNA orthologs | 1,668 |
| Human circRNA orthologs | 179 |
| 芯片规格 | 8 * 15K |
1. Guo, J. U., V. Agarwal, et al. (2014). "Expanded identification and characterization of mammalian circular RNAs." Genome Biol 15(7): 409.
2. Jeck, W. R., J. A. Sorrentino, et al. (2013). "Circular RNAs are abundant, conserved, and associated with ALU repeats." RNA 19(2): 141-157.
3. Memczak, S., M. Jens, et al. (2013). "Circular RNAs are a large class of animal RNAs with regulatory potency." Nature 495(7441): 333-338.
4. Salzman, J., R. E. Chen, et al. (2013). "Cell-type specific features of circular RNA expression." PLoS Genet 9(9): e1003777.
5. Zhang, X. O., H. B. Wang, et al. (2014). "Complementary sequence-mediated exon circularization." Cell 159(1): 134-147.
6. Zhang, Y., X. O. Zhang, et al. (2013). "Circular intronic long noncoding RNAs." Mol Cell 51(6): 792-806.7. You X., I. Vlatkovic, et al. (2015). "Neural circular RNAs are derived from synaptic genes and regulated by development and plasticity." Nat Neurosci 18(4): 603-610. .
• 样品总RNA抽提
• RNA质量检测:使用Nanodrop测定总RNA在分光光度计260nm、280nm和230nm的吸收值,以计算浓度并评估纯度;使用甲醛变性琼脂糖凝胶电泳检测RNA完整性
• RNase R 处理
• cDNA 合成
• 标记
• 芯片杂交及扫描
• 数据分析及报告整理
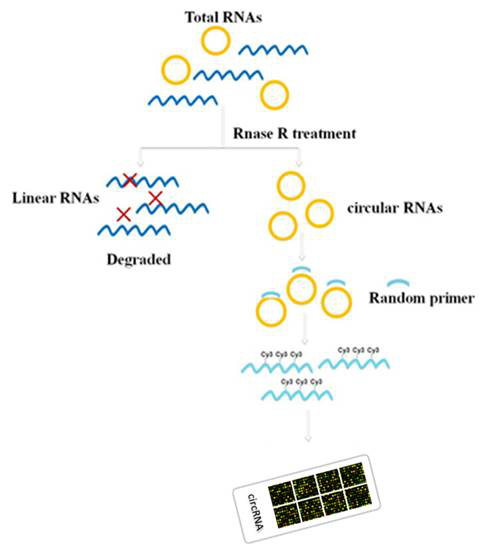
图释.:Arraystar circRNA芯片实验流程,样本RNase R前处理+随机引物标记体系确保去除线性转录本而特异性且高效地标记circRNA
circRNA抑制由DNA感受器cGAS介导的长期造血干细胞耗竭 (A Circular RNA Protects Dormant Hematopoietic Stem Cells from DNA Sensor cGAS-Mediated Exhaustion. Immunity,2018) IF:22.845
长期造血干细胞(LT-HSCs)作为潜能最高的干细胞系,具有最高的更新和分化能力,可以为短期造血干细胞、多能干细胞等提供了持续性细胞补给。大多数时候,LT-HSCs处于休眠的静息状态,其干性的维持受转录因子及骨髓内环境等多种因素的影响,比如TNF、CXCL4和I型IFN等,然而LT-HSCs在静息状态与激活状态之间的精细调控机制尚未完全解析。
作者所在的研究团队主要从事肿瘤干细胞、免疫细胞发育分化及肿瘤靶标发现与肿瘤个体化治疗等领域的研究,该研究中,作者应用Arraystar Mouse CircRNA Array研究了小鼠骨髓细胞(BM)中分离的LT-HSCs和多能干细胞(MPPs)的circRNAs表达谱。筛选出一个在LT-HSCs细胞核内高表达的circRNA cia-cGAS,通过一系列功能机制实验发现LT-HSCs中cia-cGAS可以与DNA敏感的cGAMP合成酶cGAS相互结合并抑制了其酶活性,阻碍cGAS结合基因组DNA,从而不能激活I型干扰素(IFN)的表达,维持LT-HSCs的静息状态。该研究同时也发现cia-cGAS是cGAS介导的自身免疫相关的有效抑制剂,为有效防治自身免疫性疾病与血液系统恶性肿瘤提供了新思路和潜在药物研发靶标。
• 提供详尽的circRNA-miRNA相互作用注释
Arraystar 环状RNA芯片提供circRNA可能结合的miRNA,并注释潜在的结合位点,这将有助于研究者进一步探索环状RNA作为miRNA吸附海绵的功能作用。
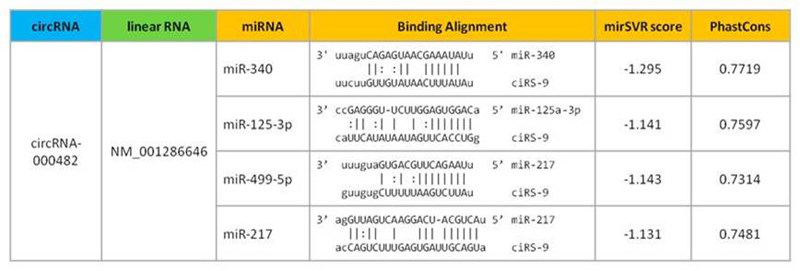
图1:circRNA及其可能结合的microRNA
• circRNA差异表达分析
差异表达circRNA是通过表达值变化倍数及统计学显著性指标p值筛选得到,这部分circRNA将通过火山图进行展示;circRNA表达模式则会通过聚类图进行展示。

图2. 差异表达circRNA、它们可能结合的microRNA及这些microRNA Response Elements (MREs)的详细展示。
→ Extracellular Vesicle-Mediated Delivery of CircSCMH1 Promotes Functional Recovery in Rodent and Nonhuman Primate Ischemic Stroke Models . Circulation . 2020
→ A Noncoding Regulatory RNAs Network Driven by Circ‐CDYL Acts Specifically in the Early Stages Hepatocellular Carcinoma . Hepatology . 2020
→ N6-methyladenosine modification of circNSUN2 facilitates cytoplasmic export and stabilizes HMGA2 to promote colorectal liver metastasis . Nature Communications . 2020
→ CircPTK2 (hsa_circ_0005273) as a novel therapeutic target for metastatic colorectal cancer . Molecular Cancer . 2020
→ Circ-AKT3 inhibits clear cell renal cell carcinoma metastasis via altering miR-296-3p/E-cadherin signals . Molecular cancer . 2020
→ circGSK3β promotes metastasis in esophageal squamous cell carcinoma by augmenting β-catenin signaling . Molecular cancer . 2020
→ circKIF4A acts as a prognostic factor and mediator to regulate the progression of triple-negative breast cancer . Molecular Cancer . 2020
→ Circular RNA hsa_circ_0000326 acts as a miR-338-3p sponge to facilitate lung adenocarcinoma progression . Journal of Experimental & Clinical Cancer Research . 2020
→ A novel circular RNA circENTPD7 contributes to glioblastoma progression by targeting ROS1 . Cancer Cell International . 2020
→ Circular RNA Circ100084 functions as sponge of miR‑23a‑5p to regulate IGF2 expression in hepatocellular carcinoma . Molecular Medicine Reports . 2020
→ circPRRC2A promotes angiogenesis and metastasis through epithelial-mesenchymal transition and upregulates TRPM3 in renal cell carcinoma . Theranostics . 2020
→ A Noncoding Regulatory RNAs Network Driven by Circ‐CDYL Acts Specifically in the Early Stages Hepatocellular Carcinoma . Hepatology . 2019
→ N6-methyladenosine modification of circNSUN2 facilitates cytoplasmic export and stabilizes HMGA2 to promote colorectal liver metastasis . Nature Communications . 2019
→ Circ-AKT3 inhibits clear cell renal cell carcinoma metastasis via altering miR-296-3p/E-cadherin signals . Molecular cancer . 2019
→ circGSK3β promotes metastasis in esophageal squamous cell carcinoma by augmenting β-catenin signaling . Molecular cancer . 2019
→ The circRNA circPTPRA suppresses epithelial-mesenchymal transitioning and metastasis of NSCLC cells by sponging miR-96-5p . EBioMedicine . 2019
→ circKDM4C suppresses tumor progression and attenuates doxorubicin resistance by regulating miR-548p/PBLD axis in breast cancer . Oncogene . 2019
→ Hsa_circ_101555 functions as a competing endogenous RNA of miR-597-5p to promote colorectal cancer progression . Oncogene . 2019
→ Circular RNA circ-RanGAP1 regulates VEGFA expression by targeting miR-877-3p to facilitate gastric cancer invasion and metastasis . Cancer Letters . 2019
→ A Circular RNA Protects Dormant Hematopoietic Stem Cells from DNA Sensor cGAS-Mediated Exhaustion . Immunity . 2018
→ PRMT5 Circular RNA Promotes Metastasis of Urothelial Carcinoma of the Bladder through Sponging miR-30c to Induce Epithelial–Mesenchymal Transition . Clinical Cancer Research . 2018

