

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 R-loop 测序(DRIPc-seq) |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
相关服务
hMeDIP-qPCR相关产品
相关资源
5羟甲基化的研究背景及与疾病的关系在人类基因组中,5mC占据总胞嘧啶的比例为2-8%,并且5mC修饰在很多生物学过程中,如基因表达、基因组稳定性维持、父母印记、X染色体失活、发育调控、衰老以及癌症的发生发展等生物学过程中发挥着不可替代的作用。5hmC作为5mC的氧化产物,主要存在于脑组织、神经细胞和胚胎干细胞中,在这些样本中的5hmC的含量在0.3-0.7%。5hmC是通过TET氧化5mC而得到,这种氧化作用依赖于亚铁离子和a-酮戊二酸。5hmC在胚胎干细胞的维持、正常血细胞生成以及恶性肿瘤和受精卵的发育等生物学过程中起着重要的作用,同时也在DNA的去甲基化过程中发挥着承上启下的作用。
为了更好更深入地了解5hmC的生物学功能,需要特异性强、方法成熟的DNA 5hmC富集技术。然而,传统的基于重亚硫酸盐的方法无法区分5-hmC和5-mC。 康成生物丨数谱生物DNA 5hmC测序(化学法)能特异性富集DNA 5hmC片段,同时有很高的富集效率,完全满足对DNA 5hmC特异性检测的需要。该方法是基于beta-GT能够特异性地将葡萄糖结合在5hmC这一原理而开展的。康成生物丨数谱生物DNA 5hmC测序(化学法)及数据分析,可以获得全基因组羟甲基化分布图,帮助研究者从一个新的角度解析胚胎发育、神经细胞分化以及疾病发生的分子机制。
Aksomics(原康成生物)为您提供一站式DNA 5hmC测序(化学法)技术服务,您只需要提供保存完好的组织或细胞标本,我司就可以为您完成从DNA提取、5hmC DNA富集、文库构建、高通量测序到数据分析的一整套服务,并提供完整的实验报告。
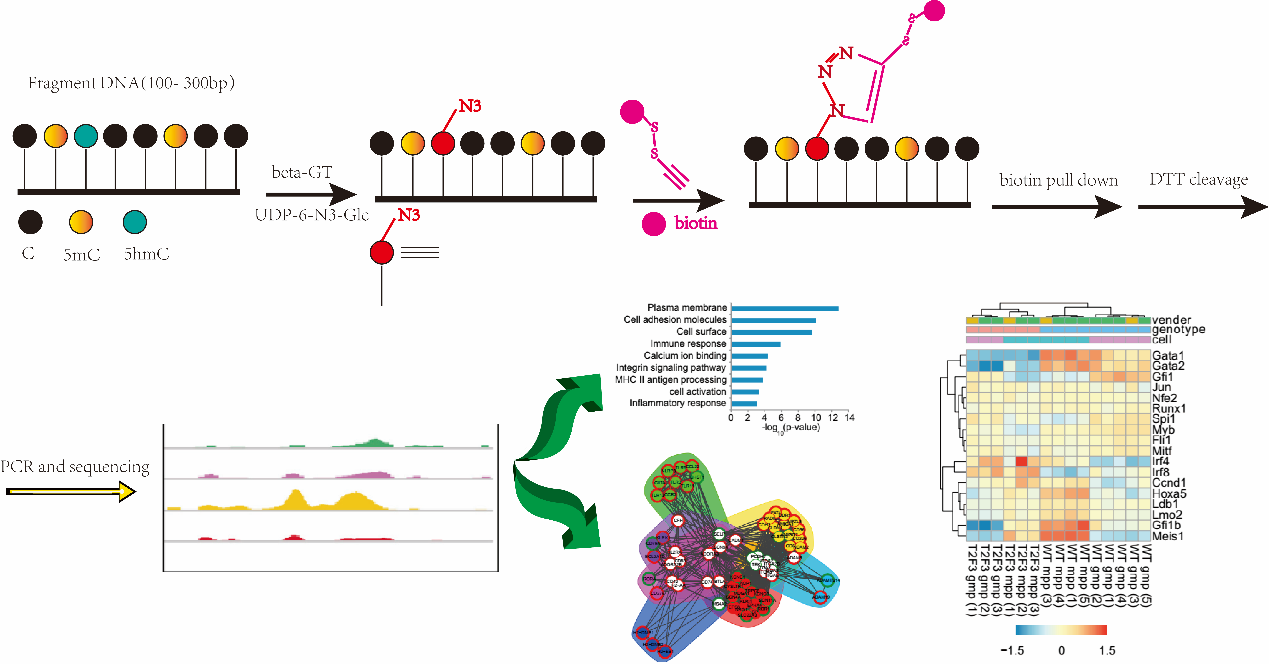
图释. DNA 5hmC测序(化学法)测序的流程示意图
高度特异性: 本方法可以从DNA修饰(C,5mC,5hmC)中特异性识别5hmC
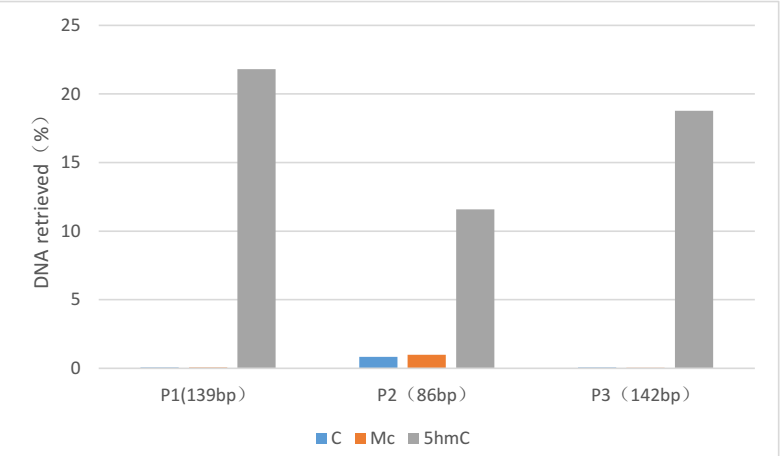
Figure 1. 分别对加入了含有3种不同修饰的spike in(分别为C、5mC和5 hmC)的DNA样品(10 ug)进行富集试验,富集完成后,分别针对spike-in的P1,p2,p3 不同位置进行PCR检测,结果显示该方法具有良好的5hmC富集特异性。
无密度偏好性: 该方法富集5hmC,不依赖于DNA的修饰水平
稳定性高:实验操作稳定性高,减少实验操作带来的误差。
灵活度高:能够直接对有基因组信息的任意物种的进行DNA 5hmC测序
精确度高:能够在实际结合位点50个碱基范围内精确定位。
提供数据可视化与文章发表级别的图谱
1. peak识别及注释
我司使用MACS2进行peak识别,并根据Ensembl数据库和Ehancer数据库(EnhancerAtlas)的注释信息,对peak进行详细注释。
(1)Peak分类
根据peak顶点与转录本的相对位置关系对peak进行分类,具体分类方法如下。
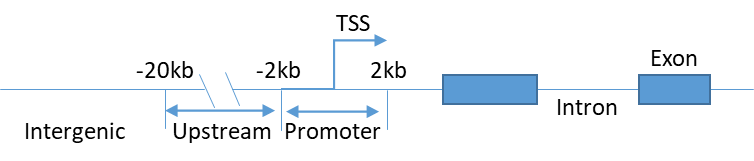
Figure1. peak分类
Table1. 各样本peaks区域

(2)peak统计
根据以上分类,对不同区域进行统计绘制饼状图。
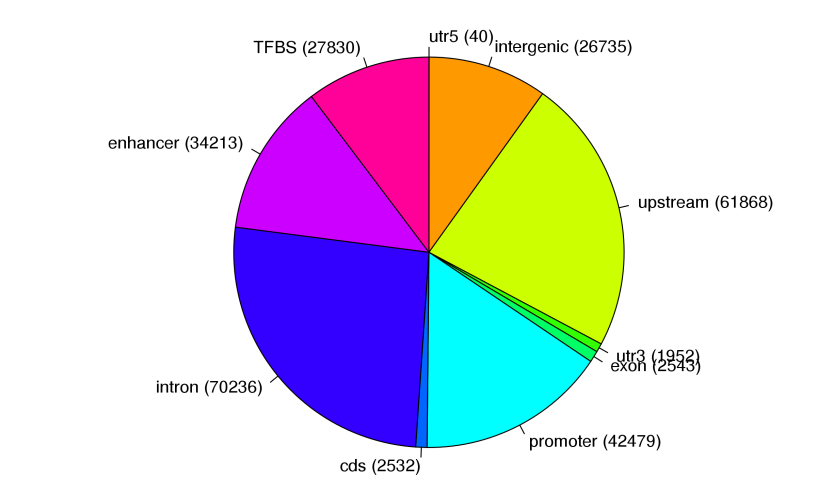
Figure2. peak统计图(TFBS: 转录因子结合位点)
对peaks和fragments在TSS(转录起始位点)及转录本周围的分布情况进行统计并绘图。
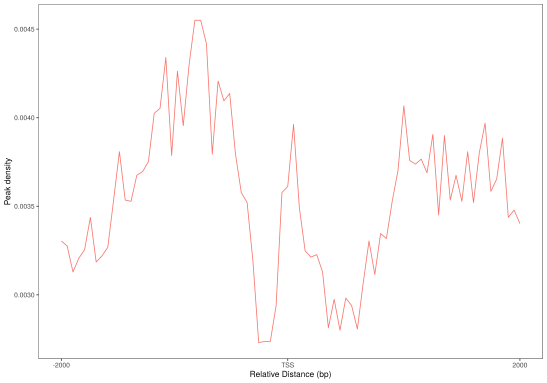

(a)Peaks密度在TSS周围的分布 (b) Peaks密度在转录本周围的分布
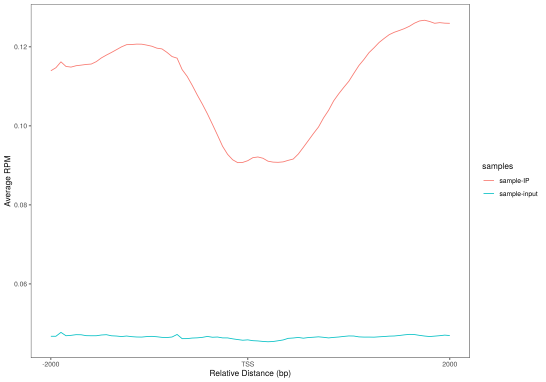
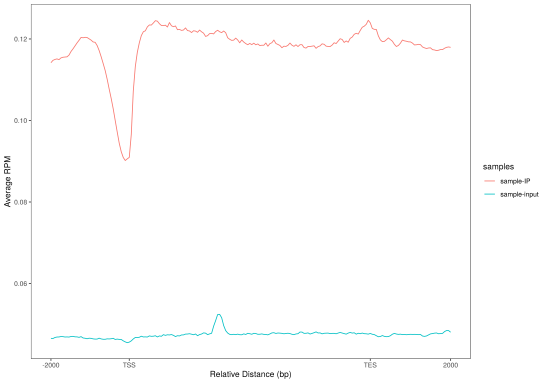
(c)标准化后的reads在TSS周围的分布 (d) 标准化后的reads在转录本周围的分布
Figure3. peak分布情况
修饰密度(peaks/C)用来展示修饰区域在整个染色体上的分布情况;将修饰水平分为低(0%~30%,绿色),中(30%~70%,蓝色),高(70%~100%,红色)三类,在Circos图中进行展示(bin=1Mbp)。
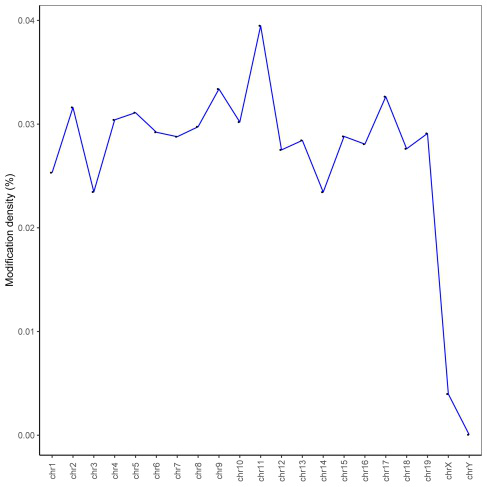
Figure4. 修饰密度在不同染色体上的分布
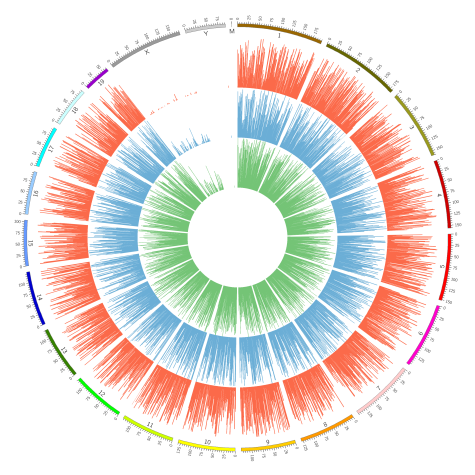
Figure5. 不同修饰水平peaks在染色体上的分布
2. 差异peak分析
康成生物丨数谱生物使用DiffBind进行组间或样本间比较的差异peak分析,并根据Ensembl数据库和Enhancer数据库(EnhancerAtlas)进行差异peak注释。
Table2. 差异peak区域

根据差异peak注释到的不同区域,对区域数量进行统计绘制条形图,并以10kb为单位对peak在染色体上的分布进行统计并绘图。


Figure6. 差异peak统计展示 (左):差异peak分布统计图; (右):差异peak染色体分布图
通过各样本在差异peak富集程度的系统聚类分析,可以了解各组样本间的关系。火山图通过-log10P_value和log2Fold_Change两个条件,对比较数据进行筛选,可以直观地展示两组数据之间富集差异的倍数变化及显著性的关系。
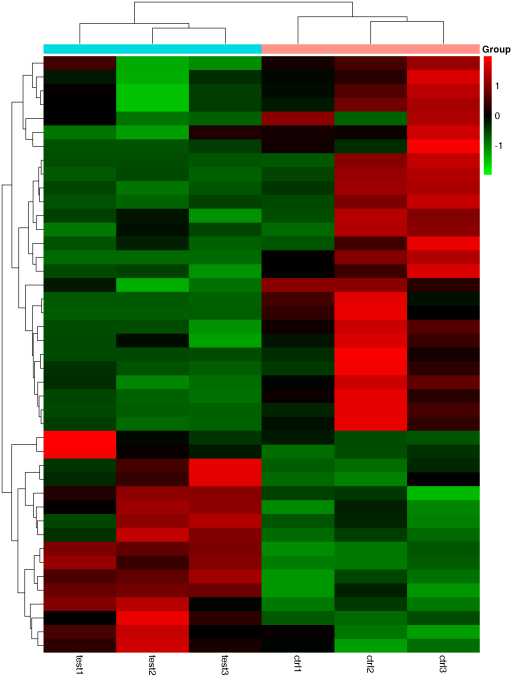
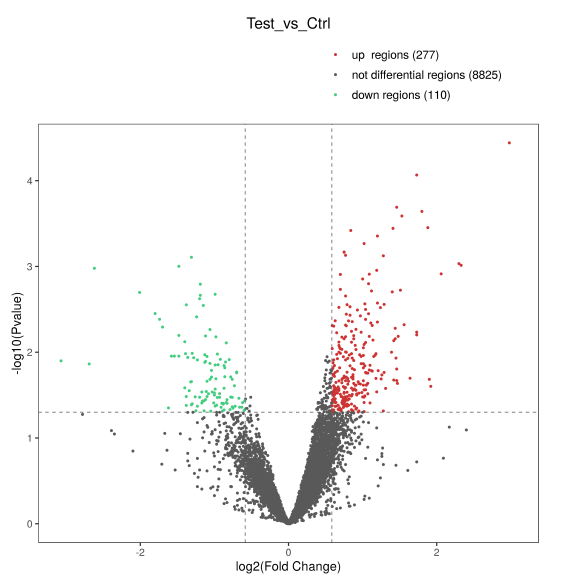
Figure 7. 差异peak聚类图和火山图 (左):聚类图; (右): 火山图
3.差异peak的GO & KEGG富集分析
(1)差异peak的GO富集分析
GeneOntology (GO)是一种基因功能分类条目,有三个子条目,分别描述每个基因的分子功能(MF: Molecular function)、细胞组成(CC: Cellular component)、参与的生物学过程(BP: Biological process)。通常的GO富集分析就是利用统计学算法来找出一组差异表达基因和哪些具体功能条目联系最大,每个GO条目都对应一个统计值P-value来表示显著性,P-value越小表示该GO条目和输入的差异表达基因联系越大,即该组差异表达基因大部分具有该GO条目对应的描述功能。
Table3. GO富集分析结果

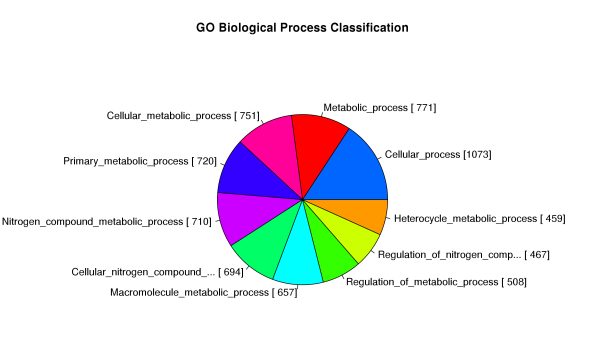
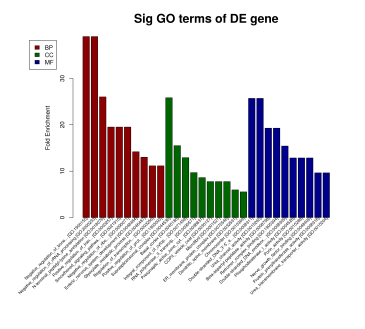
Figure 8. GO富集结果图 (左):各GO条目中出现的基因数;(右)MF、CC、BP前十富集条目柱状图
(2)差异peak的Pathway富集分析
根据KEGG database (Kyoto Encyclopedia of Genes and Genomes)中的生物学通路分类条目,来找出一组差异表达基因和哪些具体的功能条目联系最大。每个Pathway条目都对应一个统计值P-value来表示显著性,P-value越小表示该GO条目和输入的差异表达基因联系越大,即该组差异表达基因P-value越小表示该Pathway条目和输入的差异表达基因联系越大。
Table 4. KEGG富集分析结果

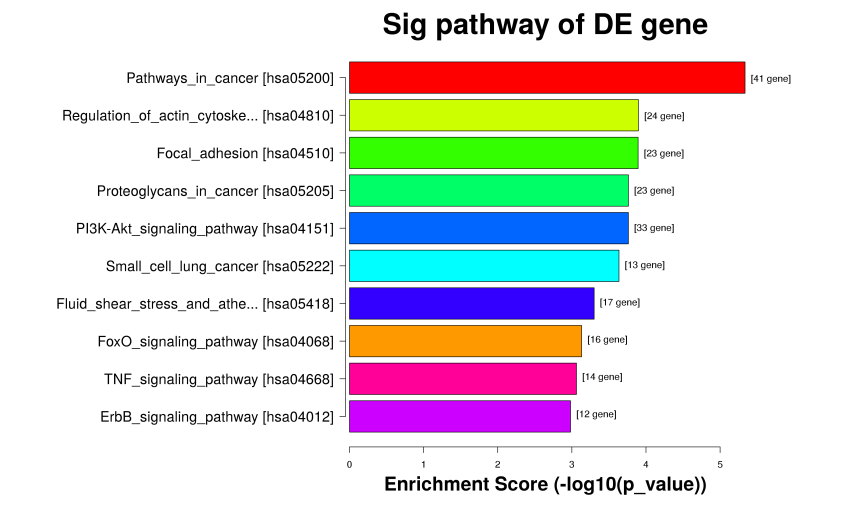
Figure 9. KEGG富集结果图
4. Motif分析
在遗传学中,motif是指一段短的且普遍存在的核苷酸或氨基酸序列。被认为有特定的生物学意义,比如DNA上的蛋白结合位点。当motif出现在基因外显子区域时,其对蛋白结构具有重要作用。我司使用MEME-ChIP(v4.9.1)中的DREME方法(适合于查找短的motif)来检测motif。将得到的motif与转录因子结合位点motif数据库(JASPARCORE2018)内的motif进行比较,从而得到相关motif的更多信息。
Table 5. Motif比较结果



Figure 10. Motif结果图 (左):Motif结果图;(右)左图中序列反向互补图
5. 比对结果可视化
我司将测序数据储存为bedGraph文件,可以在IGV或UCSC基因组浏览器中查看。
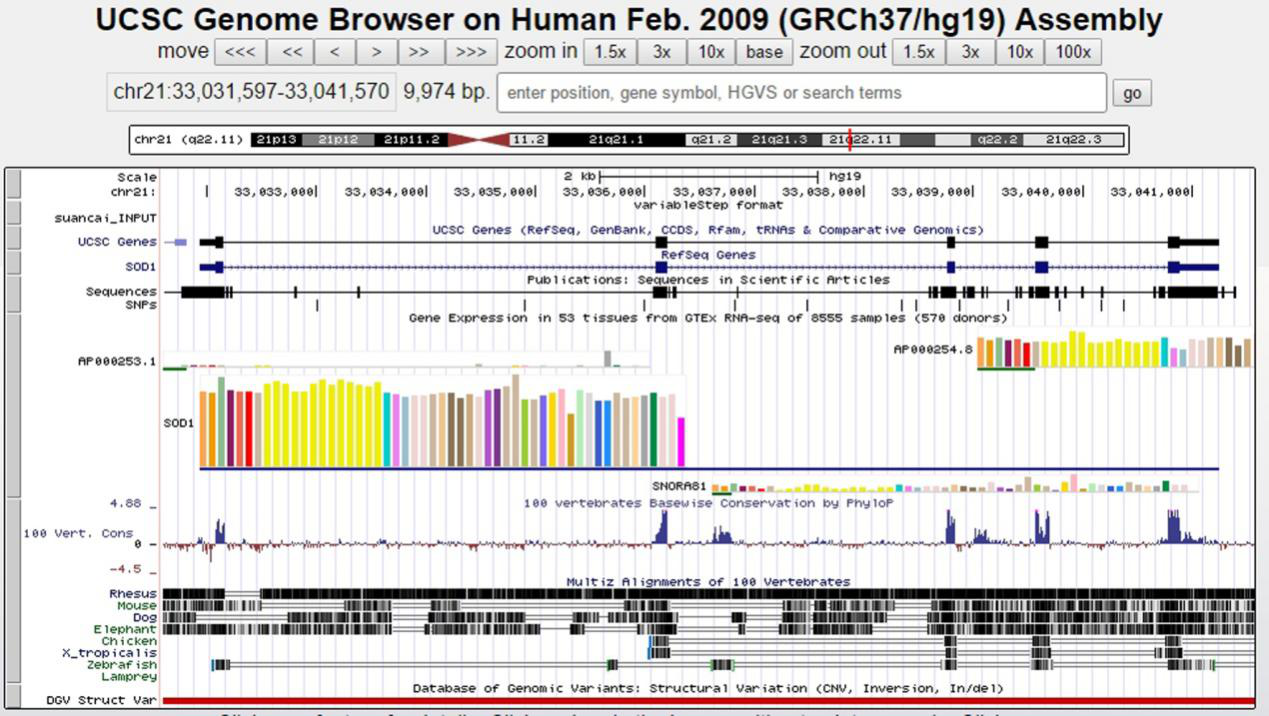
Figure 11. 比对结果可视化

