

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
Arraystar circRNA表观转录组芯片,可定量检测circRNA中m6A/m5C/m1A/ac4C/m7G/Ψ 的表观转录修饰水平。对于m6A修饰,我们提供了两种m6A- RNA富集方法可供选择:m6A抗体免疫沉淀(MeRIP)或GST-YTH pull down富集。
芯片优势
与MeRIP-seq相比,Arraystar表观转录组芯片有其独特的优势:
• 同时检测修饰位置以及修饰比例
• 反向剪接位点特异性探针与RnaseR预处理双重保障,提高circRNA修饰检测特异性
• 适用于circRNA研究,甚至用MeRIP-seq方法很难检测的circRNA也可用芯片检测
• 样本需求量少,总RNA量低至3ug
• 适用于多种样本,比如血清/血浆/全血样本
Arraystar circRNA表观转录组芯片产品列表
| 芯片 | 表观修饰 | 方法 | 规格 | 描述 |
|---|---|---|---|---|
| Human circRNA 表观转录组芯片 | m6A/m5C/m1A/ac4C/m7G/Ψ | 抗体法 | 8×15K | 13,617 circRNAs |
| Mouse circRNA 表观转录组芯片 | m6A/m5C/m1A/ac4C/m7G/Ψ | 抗体法 | 8×15K | 14,236 circRNAs |
| Rat circRNA 表观转录组芯片 | m6A/m5C/m1A/ac4C/m7G/Ψ | 抗体法 | 8×15k | 14,145 circRNAs |
| Human circRNA 表观转录组芯片 | m6A | GST-YTH pull down | 8×15k | 13,617 circRNAs |
| Mouse circRNA 表观转录组芯片 | m6A | GST-YTH pull down | 8×15k | 14,236 circRNAs |
| Rat circRNA 表观转录组芯片 | m6A | GST-YTH pull down | 8×15k | 14,145 circRNAs |
RNA转录后修饰,比如m6A, m1A, m5C, 和假尿嘧啶(Ψ)修饰,共同组成了表观转录组,是一种新层次的基因表达调控方式。m6A是mRNA和lncRNA上含量最丰富的修饰之一,在转录后各个水平影响mRNA/lncRNA 的代谢和功能。此外,m6A还参与了其它ncRNA的功能,包括circRNA非帽依赖的翻译起始,和pri-miRNA的加工过程。
RNA修饰的潜在功能不仅取决于其所修饰的是何种基因转录本,同时也取决于被修饰部分在该转录本中所占的百分比。然而,目前大部分转录组水平的的RNA修饰检测方法着重于寻找转录本上的修饰位点,不能够定量地检测被修饰转录本的百分比。这一类定量信息的缺乏已引起越来越多科研工作者的关注。
|
科学家最关注的问题 “mRNA修饰的潜在影响既取决于其分子效应,也取决于被修饰转录本的百分比。例如,一种可以加速mRNA降解的修饰,如果只有1%的转录本被修饰,显然不太可能产生任何生物功能,然而当一种修饰可以促使mRNA翻译成新的蛋白亚型时,即使修饰水平很低,也可能产生重要的生物功能。当前的m6A和Ψ检测方法局限性在于缺乏修饰程度的定量信息。m6A的调控作用可以通过pulldown m6A的方法,检测特定序列在不同状态下的相对富集程度进行推断,但不能从这些数据中获得被修饰mRNA的绝对量。新的可定量m6A和Ψ的高通量检测方法,将极大的促进该领域的发展。 [1] Wendy V. Gilbert, Tristan A. Bell, Cassandra Schaening. Science (2016)
另一个重要的问题是阐明RNA修饰的化学计量的动态变化。目前,表观转录组学研究的重点大多是哪些位点被修饰,而不是RNA中每个被修饰位点的占比。低通量分析mRNA和病毒RNA的m6A修饰位点显示,任何m6A修饰位点的占比都不会达到100%。修饰的化学计量变化可能是一种RNA生物学修饰的动态变化参数。修饰可以影响mRNA的结构,和(或)对RBPs的招募。任何特殊位点的修饰,会导致同一mRNA群体仅仅由于结构或是结合reader的不同,分为两个mRNA亚群。因此,改变修饰的化学计量数,可能是同一个RNA转录本行使不同功能的一种机制。目前急需可以检测修饰化学计量数的高通量方法,以阐明表观转录组学在这一方面的问题。 [4] Cole J.T. Lewis, Tao Pan, Auinash Kalsotra. Nat Rev Mol Cell Biol (2017) |
Arraystar circRNA表观观转录组芯片结合了双荧光通道芯片技术与RNA修饰免疫共沉淀技术,在转录本水平对RNA修饰进行定量检测。定量的表观转录组图谱可为RNA修饰调控研究提供重要信息。
参考文献
1. Gilbert WV, Bell TA, Schaening C: Messenger RNA modifications: Form, distribution, and function. Science 2016, 352(6292):1408-1412.[PMID: 27313037]
2. Yang Y et al: Extensive translation of circular RNAs driven by N(6)-methyladenosine. Cell Res 2017, 27(5):626-641.[PMID: 28281539]
3. Alarcon CR et al: HNRNPA2B1 Is a Mediator of m(6)A-Dependent Nuclear RNA Processing Events. Cell 2015, 162(6):1299-1308.[PMID: 26321680]
4. Lewis CJ, Pan T, Kalsotra A: RNA modifications and structures cooperate to guide RNA-protein interactions. Nat Rev Mol Cell Biol 2017, 18(3):202-210.[PMID: 28144031]
5. Qin Y et al: TRIM9 short isoform preferentially promotes DNA and RNA virus-induced production of type I interferon by recruiting GSK3beta to TBK1. Cell Res 2016, 26(5):613-628.[PMID: 26915459]
circRNA表观转录组研究的理想工具
•适用于环状RNA,芯片收集了高可信度的环状RNA(在≥2个实验组和≥4个样本中有表达)
•对MeRIP-seq很难检测的circRNA,也具有高灵敏度和准确度
circRNA本身表达丰度低,测序无法准确定量,详见 为什么circRNA芯片比RNA-seq更适合于circRNA的表达分析?, MeRIP-seq检测具有m6A修饰的circRNA junction位点更难,更无法准确定量
样本需求量少
目前MeRIP-seq技术需要≥120ug的总RNA起始量,所需样本量多,限制了MeRIP-seq的适用性。相比于MeRIP-seq,Arraystar表观转录组芯片只需1ug 总RNA的起始量,所需RNA的量大大减少(表 1),为珍贵样本和来源有限的样本研究RNA修饰提供了机会。
|
|
表观转录组芯片 |
MeRIP Seq |
|
RNA起始量 |
≥1ug total RNA |
≥120ug total RNA |
|
分离mRNA或去除rRNA |
不需要 |
需要 |
|
RNA是否完整 |
不需要 |
需要 |
GST- YTH是由YTH-DF2蛋白的m6A识别结构域YTH(对应385-579位氨基酸)与GST标签融合形成的重组融合蛋白,可用于富集带有m6A修饰的RNA。YTH是一个进化上保守的结构域,它可以选择性地“读取”并结合RRACH基序中的m6A [1]。从结构上看,YTH结构域中包含的特定两个或三个色氨酸残基,能够形成影响m6A结合的芳香笼以及结合口袋。此外,由于RNA上m6A修饰位置前后的核苷酸能够与YTH结构域中的氨基酸存在相互作用关系,从而使YTH结构域偏好结合RRACH基序 [2-4](图1)。也就是说,GST-YTH通过同时识别m6A修饰结构以及识别RRACH基序的序列依赖方式结合含有m6A的RNA [1]。这两种特殊的识别方法使得GST-YTH能够有效区分m6A与m6Am以及其他相似的RNA修饰。因此,GST-YTH pull down对m6A识别具有高度特异性,而不像采用m6A抗体的MeRIP那样,会与其他结构相似的RNA修饰 [5],特别是m6Am [6]产生交叉反应。
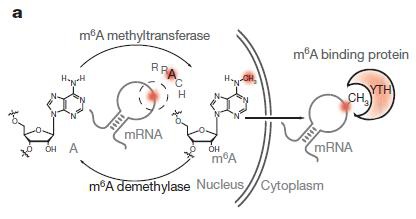
图 1. YTH以m6A结构和RRACH基序序列依赖的方式与m6A修饰的RNA结合,对m6A具有更高的特异性,而与其他类似的RNA修饰(如m6Am)没有交叉反应。
1. Wang X et al: N6-methyladenosine-dependent regulation of messenger RNA stability. Nature 2014, 505(7481):117-120.[PMID: 24284625]
2. Luo S, Tong L: Molecular basis for the recognition of methylated adenines in RNA by the eukaryotic YTH domain. Proc Natl Acad Sci U S A 2014, 111(38):13834-13839.[PMID: 25201973]
3. Theler D et al: Solution structure of the YTH domain in complex with N6-methyladenosine RNA: a reader of methylated RNA. Nucleic Acids Res 2014, 42(22):13911-13919.[PMID: 25389274]
4. Xu C et al: Structural basis for selective binding of m6A RNA by the YTHDC1 YTH domain. Nat Chem Biol 2014, 10(11):927-929.[PMID: 25242552]
5. Linder B et al: Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nat Methods 2015, 12(8):767-772.[PMID: 26121403]
6. https://sysy.com/product/202003
Human circRNA Epitranscriptomic microarray
|
探针总数 |
13,617 |
|
探针长度 |
60nt |
|
探针位点 |
circular junctions |
|
探针特异性 |
转录本特异性 |
|
标记方法 |
RNase R 预处理线性RNA,随机引物反转,体外转录标记circRNA |
|
circRNAs |
13,617 |
|
circRNA来源 |
Updated Databases: circbase, CircNet, circRNADb [1-3] Literatures: [4-10] |
|
芯片规格 |
8 × 15 K |
Mouse circRNA Epitranscriptomic microarray
|
探针总数 |
14,236 |
|
探针长度 |
60nt |
|
探针位点 |
Circular junctions |
|
探针特异性 |
转录本特异性 |
|
标记方法 |
RNase R 预处理线性RNA,随机引物反转,体外转录标记circRNA |
|
circRNAs |
14,236 |
|
circRNA来源 |
Updated Databases: circbase, CircNet, circRNADb [1-3] Literatures: scientific publications [4-5] |
|
芯片规格 |
8 × 15 K |
Rat circRNA Epitranscriptomic microarray
|
探针总数 |
14,145 |
|
探针长度 |
60nt |
|
探针位点 |
Circular junctions |
|
探针特异性 |
转录本特异性 |
|
标记方法 |
RNase R 预处理线性RNA,随机引物反转,体外转录标记circRNA |
|
circRNAs |
14,145 |
|
circRNA来源 |
Updated Databases: circbase, CircNet, circRNADb [1-3] Literatures: scientific publications [4-5] |
|
芯片规格 |
8 × 15 K |
Arraystar表观转录组芯片分析提供从您的总RNA样本开始的全面服务。该服务包括MeRIP / GST-YTH pull down、cRNA标记、芯片实验、数据分析和注释。实验过程中存在多步质量控制步骤,以确保项目成功并且数据质量可靠。您只需要把样品寄给我们,剩下的我们帮您完成!
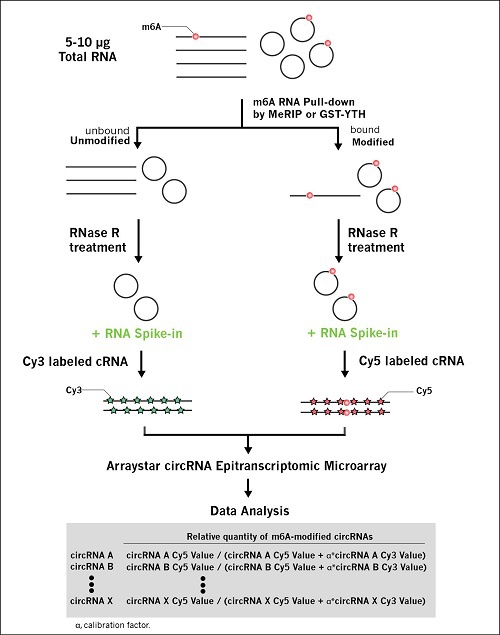
图2、m6A-circRNA表观转录组芯片实验流程
• 客户提供样本(具体见样本指南)
• RNA质量检测
• 通过m6A-MeRIP或者GST-YTH pull down富集有m6A修饰的RNA
• RNase R 处理去除线性RNA(比如rRNA, lncRNA, mRNA等)
• cRNA合成和标记(cy5标记IP-RNA,cy3标记上清RNA)
• 芯片杂交,洗脱和扫描
• 数据采集,数据注释,数据分析和总结
Arraystar在芯片表达检测、数据分析和结果注释方面有着专业而深入的知识与经验。为客户提供丰富而详细的表观转录组生物信息学数据分析结果。
差异m6A甲基化circRNA:


差异m6A甲基化circRNA聚类分析:


