

|
单细胞测序技术服务 靶向lncRNA单细胞全转录组测序 单细胞全转录组测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
RNA-蛋白相互作用 HyPro - MS CHIRP – MS RNA pull-down MS |
RNA-RNA/DNA相互作用 HyPro-seq/芯片 CHIRP-seq |
蛋白-RNA相互作用 AGO APP seq/芯片 RIP-RNA seq/芯片 |
蛋白-蛋白相互作用 CoIP-MS |
|
NGS测序技术服务 R-loop 测序分析服务 |
|
NGS测序技术服务 环状DNA测序 |
|
基因芯片技术服务 Small RNA修饰芯片 m6A单碱基分辨率芯片 mRNA&lncRNA表观转录组芯片 circRNA表观转录组芯片 |
NGS测序技术服务 表观转录组学测序服务 RNA m6A甲基化测序(MeRIP Seq) |
LC-MS mRNA碱基修饰检测 tRNA碱基修饰检测 |
PCR技术服务 MeRIP-PCR技术服务 m6A绝对定量RT-PCR技术服务 m6A单碱基位点PCR(MazF酶切法)技术服务 |
|
Ribo-seq Ribo seq |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC联合mRNA-seq |
|
蛋白表达定量 Label free非标定量技术 TMT标记定量技术 PRM靶向定量 |
Arraystar mRNA&lncRNA表观转录组芯片,可定量检测mRNA&lncRNA中m6A/m5C/m1A/ac4C/m7G/Ψ的表观转录修饰水平。对于m6A修饰,我们提供了两种m6A- RNA富集方法可供选择:m6A抗体免疫沉淀(MeRIP)或GST-YTH pull down富集。
芯片优势
与MeRIP-seq相比,Arraystar表观转录组芯片有其独特的优势:
• 可同时检测何种转录本携带修饰,以及不同条件下的修饰差异,更重要的是,还可以检测每一种转录本的修饰比例
• 全面覆盖了编码基因和LncRNA等非编码基因,甚至用MeRIP-seq方法很难检测的lncRNA也可用芯片检测
• 不需要去除rRNA,比MeRIP-seq更简单便捷
• 样本需求量少,总RNA量低至1ug
• 适用于多种样本,比如血清/血浆/全血样本
Arraystar mRNA&lncRNA表观转录组芯片产品列表
| 芯片 | 表观修饰 | 方法 | 规格 | 描述 |
|---|---|---|---|---|
| Human mRNA&lncRNA 表观转录组芯片 | m6A/m5C/m1A/ac4C/m7G/Ψ | 抗体法 | 8×60K | 44,122 mRNAs; 12,496 lncRNAs; 3,813 Mid-size ncRNAs |
| Mouse mRNA&lncRNA 表观转录组芯片 | m6A/m5C/m1A/ac4C/m7G/Ψ | 抗体法 | 8×60K | 48,161 mRNAs; 8,393 lncRNAs; 4,087 Mid-size ncRNAs |
| Rat mRNA&lncRNA 表观转录组芯片 | m6A/m5C/m1A/ac4C/m7G/Ψ | 抗体法 | 4×44k | 27,770 mRNAs; 10,582 lncRNAs; 2,505 Mid-size ncRNA |
| Human mRNA&lncRNA 表观转录组芯片 | m6A | GST-YTH pull down | 8×60K | 44,122 mRNAs; 12,496 lncRNAs; 3,813 Mid-size ncRNAs |
| Mouse mRNA&lncRNA 表观转录组芯片 | m6A | GST-YTH pull down | 8×60K | 48,161 mRNAs; 8,393 lncRNAs; 4,087 Mid-size ncRNAs |
| Rat mRNA&lncRNA 表观转录组芯片 | m6A | GST-YTH pull down | 4×44k | 27,770 mRNAs; 10,582 lncRNAs; 2,505 Mid-size ncRNA |
RNA转录后修饰,比如m6A, m1A, m5C, 和假尿嘧啶(Ψ)修饰,共同组成了表观转录组,是一种新层次的基因表达调控方式。m6A是mRNA和lncRNA上含量最丰富的修饰之一,在转录后各个水平影响mRNA/lncRNA 的代谢和功能。此外,m6A还参与了其它ncRNA的功能,包括circRNA非帽依赖的翻译起始,和pri-miRNA的加工过程。
RNA修饰的潜在功能不仅取决于其所修饰的是何种基因转录本,同时也取决于被修饰部分在该转录本中所占的百分比。然而,目前大部分转录组水平的的RNA修饰检测方法着重于寻找转录本上的修饰位点,不能够定量地检测被修饰转录本的百分比。这一类定量信息的缺乏已引起越来越多科研工作者的关注。
|
科学家最关注的问题 “mRNA修饰的潜在影响既取决于其分子效应,也取决于被修饰转录本的百分比。例如,一种可以加速mRNA降解的修饰,如果只有1%的转录本被修饰,显然不太可能产生任何生物功能,然而当一种修饰可以促使mRNA翻译成新的蛋白亚型时,即使修饰水平很低,也可能产生重要的生物功能。当前的m6A和Ψ检测方法局限性在于缺乏修饰程度的定量信息。m6A的调控作用可以通过pulldown m6A的方法,检测特定序列在不同状态下的相对富集程度进行推断,但不能从这些数据中获得被修饰mRNA的绝对量。新的可定量m6A和Ψ的高通量检测方法,将极大的促进该领域的发展。 [1] Wendy V. Gilbert, Tristan A. Bell, Cassandra Schaening. Science (2016)
另一个重要的问题是阐明RNA修饰的化学计量的动态变化。目前,表观转录组学研究的重点大多是哪些位点被修饰,而不是RNA中每个被修饰位点的占比。低通量分析mRNA和病毒RNA的m6A修饰位点显示,任何m6A修饰位点的占比都不会达到100%。修饰的化学计量变化可能是一种RNA生物学修饰的动态变化参数。修饰可以影响mRNA的结构,和(或)对RBPs的招募。任何特殊位点的修饰,会导致同一mRNA群体仅仅由于结构或是结合reader的不同,分为两个mRNA亚群。因此,改变修饰的化学计量数,可能是同一个RNA转录本行使不同功能的一种机制。目前急需可以检测修饰化学计量数的高通量方法,以阐明表观转录组学在这一方面的问题。 [4] Cole J.T. Lewis, Tao Pan, Auinash Kalsotra. Nat Rev Mol Cell Biol (2017) |
Arraystar mRNA&lncRNA表观观转录组芯片结合了双荧光通道芯片技术与RNA修饰免疫共沉淀技术,在转录本水平对RNA修饰进行定量检测。定量的表观转录组图谱可为RNA修饰调控研究提供重要信息。
参考文献
1. Gilbert WV, Bell TA, Schaening C: Messenger RNA modifications: Form, distribution, and function. Science 2016, 352(6292):1408-1412.[PMID: 27313037]
2. Yang Y et al: Extensive translation of circular RNAs driven by N(6)-methyladenosine. Cell Res 2017, 27(5):626-641.[PMID: 28281539]
3. Alarcon CR et al: HNRNPA2B1 Is a Mediator of m(6)A-Dependent Nuclear RNA Processing Events. Cell 2015, 162(6):1299-1308.[PMID: 26321680]
4. Lewis CJ, Pan T, Kalsotra A: RNA modifications and structures cooperate to guide RNA-protein interactions. Nat Rev Mol Cell Biol 2017, 18(3):202-210.[PMID: 28144031]
5. Qin Y et al: TRIM9 short isoform preferentially promotes DNA and RNA virus-induced production of type I interferon by recruiting GSK3beta to TBK1. Cell Res 2016, 26(5):613-628.[PMID: 26915459]
芯片可定量每一种转录本的修饰百分比
同一种RNA转录本的修饰亚群和非修饰亚群,由于其结构和结合蛋白的不同,会导致不同的命运(图1)。重要的是,转录本的修饰比例与它们的功能高度相关。然而目前的修饰检测方法,比如MeRIP-seq(m6A-seq),可以确定修饰的位置,却无法对一个特定RNA转录本修饰和非修饰部分的相对含量进行定量。Arraystar表观转录组芯片能够在同一个芯片中通过双荧光通道的方法检测每个转录本修饰亚群和非修饰亚群的百分比(图 2),同时对何种转录本发生修饰和不同条件下转录本的修饰差异进行鉴定。
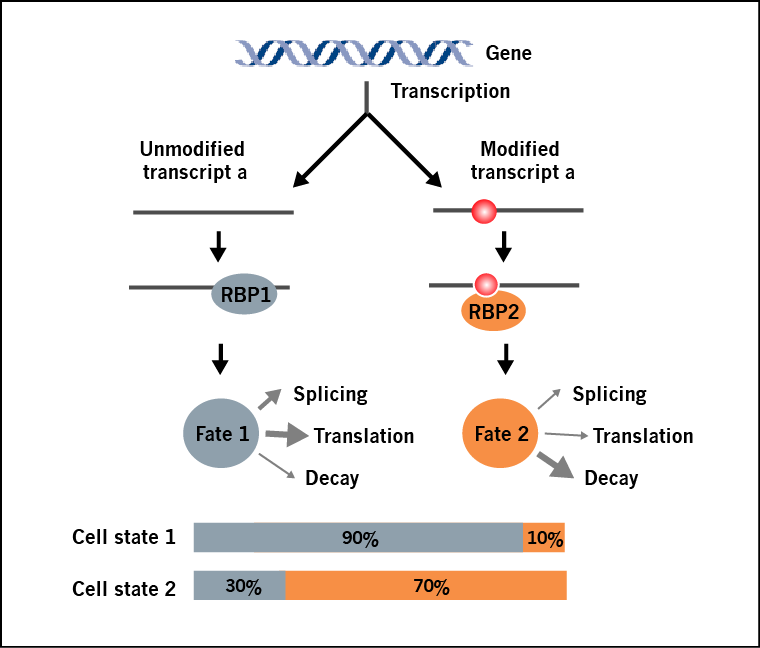
图 1.同一种RNA转录本,随着其修饰的化学计量数发生变化,其功能也随之改变。在某一种细胞条件下,携带修饰的“RNA 转录本a”所占百分比可能非常低(比如,细胞状态1),但在另外一种条件下丰度变的很高(比如,细胞状态2)。通过引起RNA结构改变,或招募修饰阅读蛋白, “转录本a”的命运发生变化,比如从蛋白翻译转变为RNA降解。
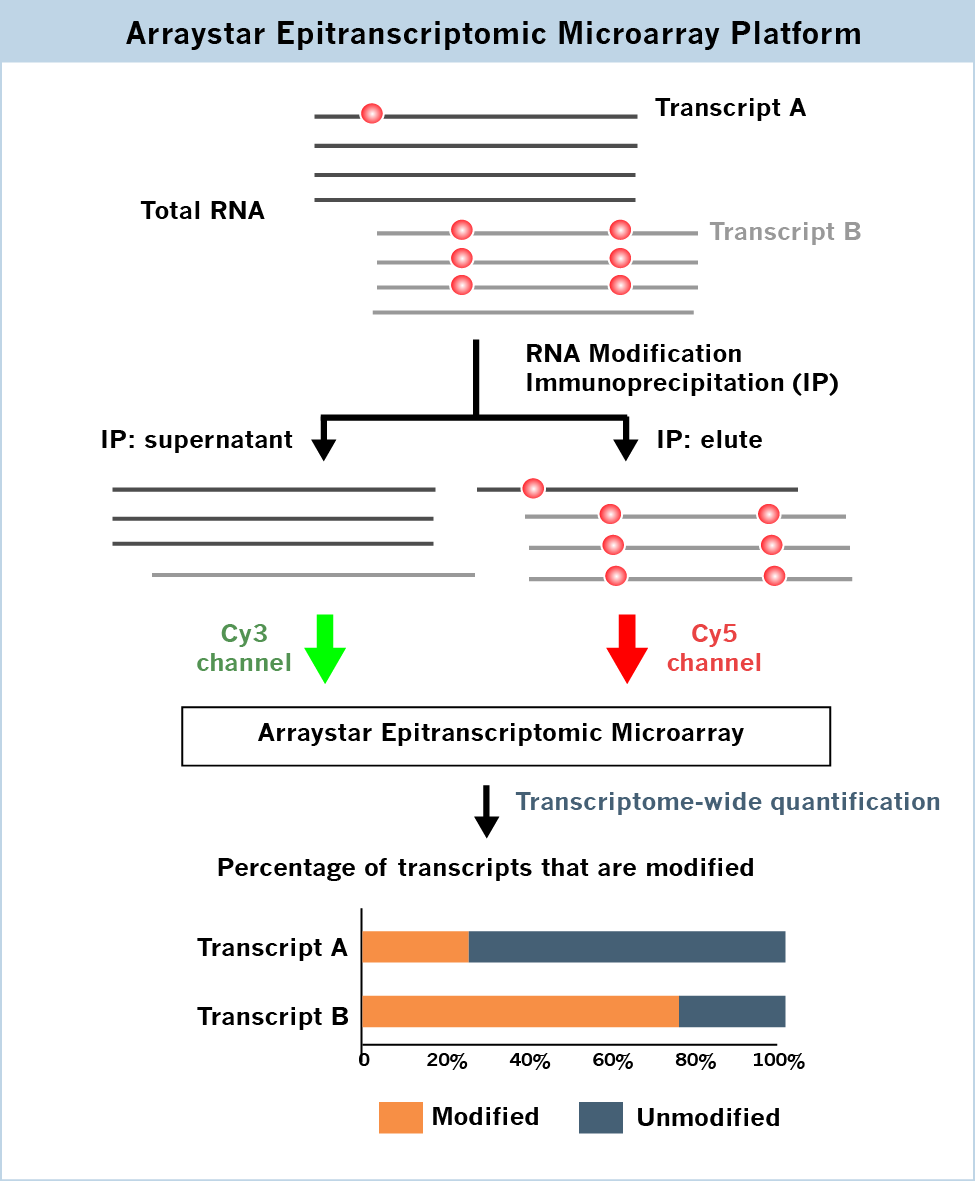
图 2.Arraystar表观转录组芯片可以在同一张芯片上使用Cy5检测免疫共沉淀的修饰RNA,用Cy3检测上清中的非修饰RNA,从而检测每一个转录本中修饰和非修饰的百分比。选择性剪切产生的转录本异构体可以被转录本特异性探针所检测。
覆盖编码基因和LncRNA等非编码基因的表观转录组
•适用于mRNA, lncRNA, pre-miRNA, pri-miRNA, snoRNA, 和 snRNA
•对MeRIP-seq很难检测的lncRNA,也具有高灵敏度和准确度
lncRNAs表达丰度低,测序无法准确定量,详见 RNA-seq对lncRNA检测的局限性 ,MeRIP-seq同样无法准确定量MeRIP富集到的lncRNAs
转录本特异性的RNA修饰检测
选择性剪切产生的转录本异构体具有组织特异性和不同的生物功能。例如,TRIM9短的异构体(NM_052978),而非长的异构体(NM_015163),能够促进DNA和RNA病毒引起的I型干扰素的表达。转录本异构体的修饰百分比发生改变,常与生物功能和疾病相关。
Arraystar表观转录组芯片,使用外显子和跨剪接位点特异性探针,确保了每个转录本异构体修饰水平检测的可信度与精确性,提供了更深层次的表观转录组学信息(图 3)。
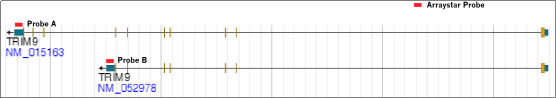
图 3. Arraystar表观转录组芯片使用转录本特异性探针A和探针B,分别检测TRIM9的长转录本(NM_015163)和短转录本(NM_052978)携带的RNA修饰。
样本需求量少
目前MeRIP-seq技术需要≥120ug的总RNA起始量,所需样本量多,限制了MeRIP-seq的适用性。相比于MeRIP-seq,Arraystar表观转录组芯片只需1ug 总RNA的起始量,所需RNA的量大大减少(表 1),为珍贵样本和来源有限的样本研究RNA修饰提供了机会。
|
|
表观转录组芯片 |
MeRIP Seq |
|
RNA起始量 |
≥1ug total RNA |
≥120ug total RNA |
|
分离mRNA或去除rRNA |
不需要 |
需要 |
|
RNA是否完整 |
不需要 |
需要 |
GST- YTH是由YTH-DF2蛋白的m6A识别结构域YTH(对应385-579位氨基酸)与GST标签融合形成的重组融合蛋白,可用于富集带有m6A修饰的RNA。YTH是一个进化上保守的结构域,它可以选择性地“读取”并结合RRACH基序中的m6A [1]。从结构上看,YTH结构域中包含的特定两个或三个色氨酸残基,能够形成影响m6A结合的芳香笼以及结合口袋。此外,由于RNA上m6A修饰位置前后的核苷酸能够与YTH结构域中的氨基酸存在相互作用关系,从而使YTH结构域偏好结合RRACH基序 [2-4](图1)。也就是说,GST-YTH通过同时识别m6A修饰结构以及识别RRACH基序的序列依赖方式结合含有m6A的RNA [1]。这两种特殊的识别方法使得GST-YTH能够有效区分m6A与m6Am以及其他相似的RNA修饰。因此,GST-YTH pull down对m6A识别具有高度特异性,而不像采用m6A抗体的MeRIP那样,会与其他结构相似的RNA修饰 [5],特别是m6Am [6]产生交叉反应。
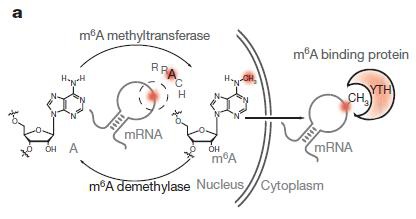
图 4. YTH以m6A结构和RRACH基序序列依赖的方式与m6A修饰的RNA结合,对m6A具有更高的特异性,而与其他类似的RNA修饰(如m6Am)没有交叉反应。
1. Wang X et al: N6-methyladenosine-dependent regulation of messenger RNA stability. Nature 2014, 505(7481):117-120.[PMID: 24284625]
2. Luo S, Tong L: Molecular basis for the recognition of methylated adenines in RNA by the eukaryotic YTH domain. Proc Natl Acad Sci U S A 2014, 111(38):13834-13839.[PMID: 25201973]
3. Theler D et al: Solution structure of the YTH domain in complex with N6-methyladenosine RNA: a reader of methylated RNA. Nucleic Acids Res 2014, 42(22):13911-13919.[PMID: 25389274]
4. Xu C et al: Structural basis for selective binding of m6A RNA by the YTHDC1 YTH domain. Nat Chem Biol 2014, 10(11):927-929.[PMID: 25242552]
5. Linder B et al: Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nat Methods 2015, 12(8):767-772.[PMID: 26121403]
6. https://sysy.com/product/202003
Human mRNA&lncRNA Epitranscriptomic microarray
|
探针总数 |
60,613 |
|
探针长度 |
60 nt |
|
探针位点 |
mRNA & lncRNA: 特异性外显子和选择性剪切位点 Mid-size ncRNAs: ncRNA的独特序列区域 |
|
探针特异性 |
转录本特异性 |
|
标记方法 |
全长标记cRNAs,无3’偏好性,丰度很低的降解RNA也可标记 |
|
蛋白编码mRNAs |
44,122 |
|
LncRNAs |
12,496 |
|
Mid-size ncRNAs |
1,366 (pre-miRNAs), 1,642 (pri-miRNAs), 19 (snRNAs), 786 (snoRNAs) |
|
mRNA 数据库 |
Refseq, UCSC, GENECODE, FANTOM5 CAT [2-6] |
|
lncRNA 数据库 |
Arraystar LncRNA 收集方式: 收录所有已更新至2018年的数据库和已发表文献中的lncRNAs, 每一个lncRNA基因优先挑选“Canonical” 或 “longest” 转录本 外部数据库 (更新至2018): Refseq, UCSC, GENCODE, FANTOM5 CAT, LncRNAdb, RNAdb, NRED [2-8] 参考文献: 收录至2018年所有文献中的lncRNA [9-45] |
|
Mid-size ncRNA 数据库 |
GENECODE, miRBase [1-2] |
|
芯片规格 |
8 × 60 K |
Mouse mRNA&lncRNA Epitranscriptomic microarray
|
探针总数 |
60,773 |
|
探针长度 |
60 nt |
|
探针位点 |
mRNA & lncRNA: 特异性外显子和选择性剪切位点 Mid-size ncRNAs: ncRNA的独特序列区域 |
|
探针特异性 |
转录本特异性 |
|
标记方法 |
全长标记cRNAs,无3’偏好性,丰度很低的降解RNA也可标记 |
|
蛋白编码mRNAs |
48,161 |
|
LncRNAs |
8,393 |
|
Mid-size ncRNAs |
701 (pre-miRNAs), 957 (pri-miRNAs), 1229 (snRNAs), 1,200 (snoRNAs) |
|
mRNA 数据库 |
Refseq, UCSC, GENECODE [2-3] |
|
LncRNA 数据库
|
Arraystar LncRNA 收集方式: 收录所有已更新至2018年的数据库和已发表文献中的lncRNAs, 每一个lncRNA基因优先挑选“Canonical” 或 “longest” 转录本 外部数据库 (更新至2018): Refseq, UCSC, GENCODE, GenBank, lncRNAdb, NRED [2-7] 参考文献s: lincRNA分类[8-11], T-UCRs [12], 进化保守 LncRNAs [13], 收录至2018年所有文献中lncRNA [14-22] |
|
Mid-size ncRNA 数据库 |
GENECODE, miRBase [1-2] |
|
芯片规格 |
8 × 60 K |
Rat mRNA&lncRNA Epitranscriptomic microarray
|
探针总数 |
40,991 |
|
探针长度 |
60nt |
|
探针位点 |
mRNA & lncRNA: 特异性外显子和选择性剪切位点 Mid-size ncRNAs: ncRNA的独特序列区域 |
|
探针特异性 |
转录本特异性 |
|
标记方法 |
全长标记cRNAs,无3’偏好性,丰度很低的降解RNA也可标记 |
|
蛋白编码mRNAs |
27,770 |
|
LncRNAs |
10,582 |
|
Mid-size ncRNAs |
432 (pre-miRNAs), 449 (pri-miRNAs), 155 (snRNAs), 1,469 (snoRNAs) |
|
mRNA 数据库 |
Refseq, Ensembl 92[1, 5] |
|
LncRNA 数据库 |
Arraystar LncRNA 收集方式: 收录所有已更新至2018年的数据库和已发表文献中的lncRNAs, 每一个lncRNA基因优先挑选“Canonical” 或 “longest” 转录本 正在更新的数据库: Ensembl 92 [1] , Refseq [5] 参考文献: T-UCRs [6-9] |
|
Mid-size ncRNA来源数据库 |
GENECODE, miRBase, GtRNADb [2-3] |
|
芯片规格 |
4 × 44K |
Arraystar表观转录组芯片分析提供从您的总RNA样本开始的全面服务。该服务包括MeRIP / GST-YTH pull down、cRNA标记、芯片实验、数据分析和注释。实验过程中存在多步质量控制步骤,以确保项目成功并且数据质量可靠。您只需要把样品寄给我们,剩下的我们帮您完成!
Arraystar mRNA&lncRNA Epitranscriptomic Microarray

图5、m6A-mRNA&LncRNA表观转录组芯片实验流程
• 客户提供样本(具体见样本指南)
• RNA质量检测
• 通过m6A-MeRIP或者GST-YTH pull down富集有m6A修饰的RNA
• cRNA合成和双色荧光标记(cy5标记IP-RNA,cy3标记上清RNA)
• 芯片杂交,洗脱和扫描
• 数据采集,数据注释,数据分析和总结
Arraystar在芯片表达检测,数据分析和结果注释方面有着专业而深入的知识与经验。为客户提供丰富而详细的表观转录组生物信息学数据分析结果。
差异m6A甲基化转录本(mRNA,lncRNA和mid-size ncRNA):

差异m6A甲基化mRNA聚类分析:

差异m6A甲基化mRNA GO富集分析:
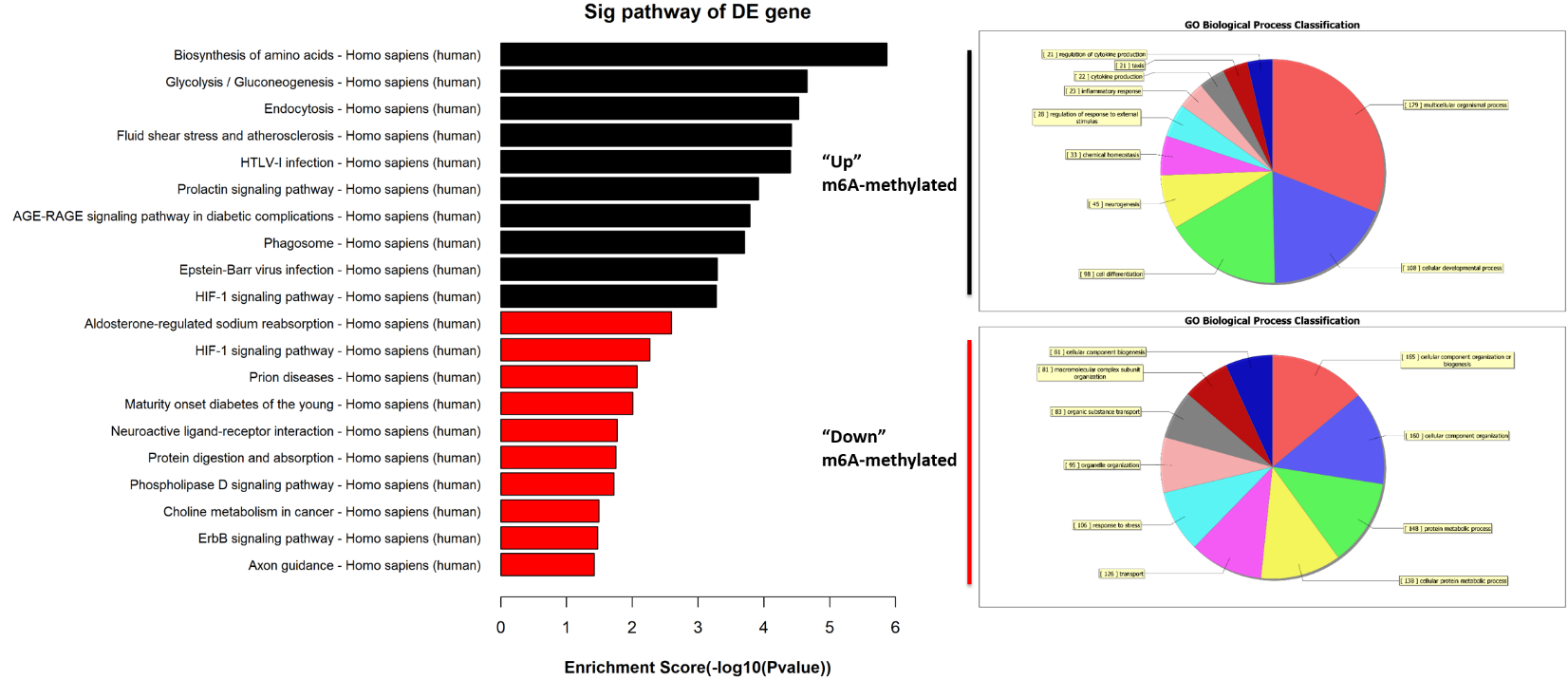
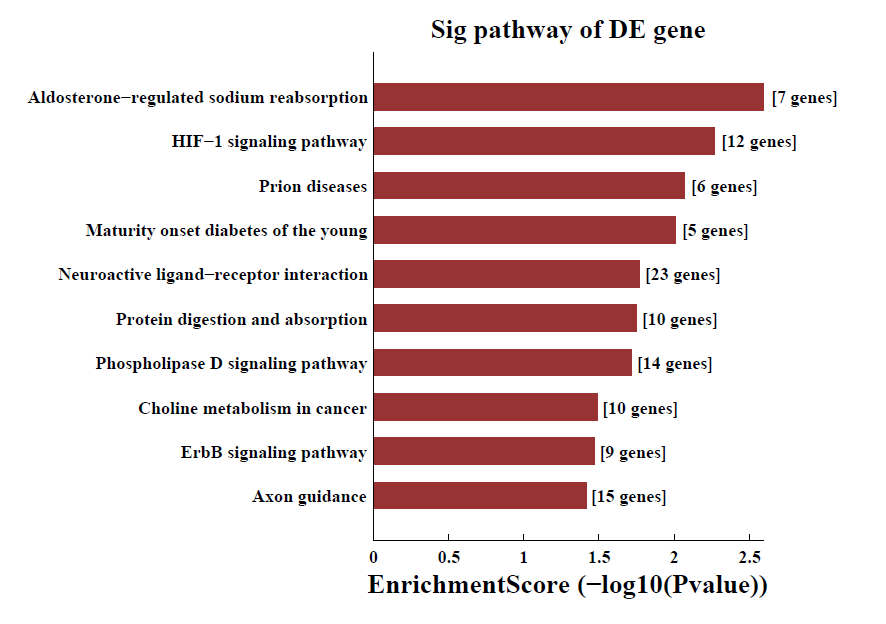
差异m6A甲基化mRNA pathway分析:
ALKBH5 regulates cardiomyocyte proliferation and heart regeneration by demethylating the mRNA of YTHDF1 . Theranostics . 2021
RBM15 facilitates laryngeal squamous cell carcinoma progression by regulating TMBIM6 stability through IGF2BP3 dependent . Journal of Experimental & Clinical Cancer Research . 2021
ALKBH5-mediated m6A mRNA Methylation Governs Human Embryonic Stem Cell Cardiac Commitment . Molecular Therapy-Nucleic Acids . 2021
ALKBH5 suppresses tumor progression via an m 6 A-dependent epigenetic silencing of pre-miR-181b-1/YAP signaling axis in osteosarcoma . Cell Death & Disease . 2021
RBM15-mediated N6-methyladenosine modification affects COVID-19 severity by regulating the expression of multitarget genes . Cell Death & Disease . 2021
The potential roles of m6 A modification in regulating the inflammatory response in microglia . Journal of Neuroinflammation . 2021
Microarray analysis of genes with differential expression of m6A methylation in lung cancer . Bioscience Reports . 2021
Bioinformatics analysis integrating metabolomics of m6A RNA microarray in intervertebral disc degeneration . Epigenomics . 2020
Fusarium infection alters the m6A-modified transcript landscape in the cornea . Experimental Eye Research . 2020
Epitranscriptome of the ventral tegmental area in a deep brain-stimulated chronic unpredictable mild stress mouse model . Translational Neuroscience . 2020

