|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 O-GlcNac修饰蛋白质组学 |
人,小鼠和大鼠基因组中存在481个完全相同超高保守区域(UCR), T-UCR是一类由这些超高保守区域转录出来的长链非编码RNA。T-UCR并不翻译成蛋白,是一类特殊的长链非编码RNA(LncRNA)。大部分T-UCR的功能仍然未知,但其高度保守性及其在多种组织中的大量表达表明了其在生物功能中的重要作用。
T-UCR的表达类型与表达量变化已被证明与人类癌症的生存率息息相关,包括慢性淋巴细胞白血病,结肠癌,原发性肝癌以及神经细胞瘤等。此外,研究显示某些T-UCR,特别是uc.73和uc.338能够作为癌基因启动癌细胞的大量生长。因此,T-UCR可作为癌症诊断和预后的标记物,并成为癌症治疗的潜在靶点。
Arraystar公司作为LncRNA研究先驱,推出了人T-UCR芯片。这款芯片不仅可以检测T-UCR的表达情况,还能检测UCR及其附近区域的RNA转录本,帮助您发现新的T-UCR。
Aksomics(原康成生物)为您提供一站式T-UCR芯片技术服务,您只需要提供保存完好的组织或细胞标本,Aksomics的芯片技术服务人员就可为您完成全部实验操作,并提供完整的实验报告。
Arraystar 人T-UCR芯片参数:
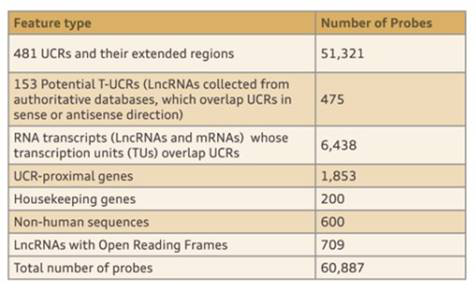
● 全面可靠的数据来源,收录转录组数据库预测的153条潜在T-UCRs
● 独特的60nt tiling探针设计,全面覆盖481个UCRs及上下游1kb区域,满足研究者检测已知T-UCR和发现新T-UCR的需要
● 准确检测UCR及其附近区域的RNA转录本,有助于发现T-UCR对附近蛋白编码基因的调控关系
● 根据疾病关联的基因区域对T-UCR进行分类和注释,让您轻松探索疾病标志物
1. 准确检测潜在T-UCR
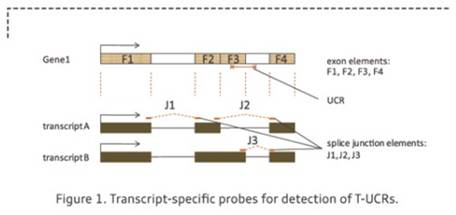
T-UCR可在人多种组织中表达[2],但其表达模式仍然未知。为帮助研究者更准确地研究T-UCR的功能,Arraystar Human T-UCR Microarray收录权威数据库如RefSeq,UCSC,Ensembl预测的153条潜在T-UCRs。可靠的数据来源确保对T-UCR更全面的覆盖。同时我们采用了“转录本特异”的探针设计,识别外显子和剪接位点,可以准确检测由一条原始转录本经可变剪切而产生的多个可变剪切异构体(Figure 1)。
2. 独特探针设计,发现新的T-UCR
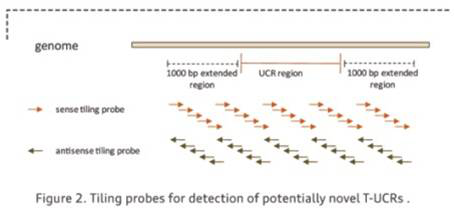
Arraystar Human T-UCR Microarray不仅能准确检测潜在T-UCR,还能助您发现新的T-UCR!数万探针(60nt tiling探针设计,间隔40nt),覆盖全部481个UCRs及上下游1kb区域(figure2),满足您发现新T-UCR的需要。
3. UCR相关转录本研究
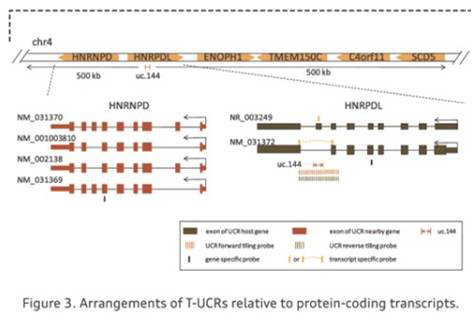
一些T-UCR存在于mRNA/LncRNA中,并且在它们的转录后调控中起着重要作用,比如可变剪切和mRNA加工。为了揭示T-UCR与这些RNA转录本之间的潜在功能联系,Arraystar T-UCR芯片还对UCR区域的1518条LncRNA和2261条mRNA的表达情况进行检测,并区分正义链和反义链(Figure 3)。
此外,一些T-UCR存在于基因间,作为远端调控或组织特异性增强子从而影响到邻近的蛋白编码基因的表达。为了研究这些mRNA,Arraystar T-UCR芯片采用“转录本特异”的探针设计,可以检测到1809条UCR附近500kb区域的蛋白编码转录本。这些特异性探针的设计,和潜在T-UCR探针一样,可以准确检测由一条原始转录本经可变剪切而产生的多个可变剪切异构体(figure 1)。
4. 结果助您前行
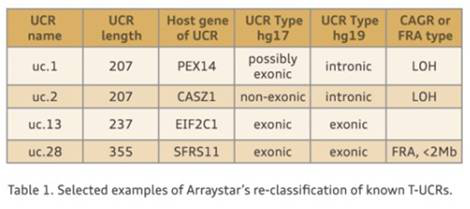
481个UCR区域最初是由Bejerano等[2]从人类基因组hg17版总结而来。Arraystar已经根据hg19版对其进行更新。我们还将这些T-UCR重新分为三组:外显子区,内含子区以及基因间隔区。更新后的分组有助于明确T-UCR的功能。最后我们还对位于两类疾病相关区域(与癌症关联的基因区域CAGR以及脆性位点FRA)的T-UCR进行了详细注释,让您可根据T-UCR结果寻找疾病诊断和预后的生物标志物,并探索疾病治疗的潜在靶点(Table 1)。