

|
单细胞测序技术服务 单细胞全转录组测序 |
|
NGS测序技术服务 DRIP-seq |
|
基因芯片技术服务 Small RNA表达芯片 Small RNA修饰芯片 m6A单碱基分辨率芯片 mRNA&lncRNA表观转录组芯片 circRNA表观转录组芯片 |
NGS测序技术服务 RNA m6A甲基化测序(MeRIP Seq) |
LC-MS mRNA碱基修饰检测 tRNA碱基修饰检测 |
PCR技术服务 MeRIP-PCR技术服务 m6A绝对定量RT-PCR技术服务 m6A单碱基位点PCR(MazF酶切法)技术服务 |
|
基因芯片技术服务 DNA甲基化芯片 DNA羟甲基化芯片 ChIP-chip |
NGS测序技术服务 DNA甲酰基胞嘧啶(5fc)修饰测序 DNA 5hmC 测序(化学法) DNA甲基化测序 DNA羟甲基化测序 染色质免疫共沉淀测序 |
PCR技术服务 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR |
|
蛋白相对定量 TMT标记定量技术 非标定量技术 |
蛋白修饰 TMT标记定量磷酸化 非标定量磷酸化 |
RNA/蛋白-蛋白相互作用 HyPro靶RNA临近标记技术 RNA-蛋白相互作用 蛋白-蛋白相互作用 |
相关服务
染色质免疫共沉淀测序 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR相关产品
相关资源
ChIP-chip(也被称为ChIP-on-chip)将染色质免疫共沉淀技术(ChIP)及与芯片方法(chip)结合,是一种研究体内DNA与蛋白相互作用的高通量技术。它的基本原理是在活细胞状态下固定蛋白质-DNA复合物,并将其随机切断为一定长度范围内的染色质小片段,然后通过免疫学方法沉淀此复合体,特异性地富集目的蛋白结合的DNA片段,通过对目的片断的纯化与检测,从而获得蛋白质与DNA相互作用的信息。ChIP-chip技术不仅可以用于研究蛋白因子与DNA的相互作用,还用可用于研究不同类型的组蛋白修饰与基因表达调控的关系等;ChIP-chip技术也已在一些至关重要的生命过程中为我们提供了重要的线索,包括细胞增殖、细胞分化、癌症发生、细胞周期、细胞凋亡以及神经生成等过程。 * 研究组蛋白的甲基化、乙酰化或磷酸化等修饰形式在所有基因启动子部位的结合状态 * 研究转录因子在所有基因启动子部位的结合状态(寻找转录因子调控的下游靶基因)
Aksomics(原康成生物)为您提供ChIP-chip全程技术服务,您只需要提供保存完好的组织或细胞标本,Aksomics技术人员就能为您完成实验操作。包括样品交联、超声打断基因组、染色质免疫共沉淀、荧光标记、芯片杂交、图像采集和数据分析等。
Arraystar启动子芯片是专门为研究启动子区域的甲基化、羟甲基化、组蛋白修饰以及转录因子结合而设计的产品,覆盖所有RefSeq数据库基因的启动子区(Arraystar RefSeq Promoter Array)或是所有非编码RNA的启动子区(Arraystar ncRNA Promoter Array),能够满足不同客户的需求。180K的芯片,启动子的覆盖范围近2kb,并覆盖了几乎所有启动子区附近的CpG岛,是一款高品质高性价比的羟甲基化芯片产品。
| 芯片名称 | 物种 | 规格 | 覆盖范围 |
|---|---|---|---|
| Arraystar Human Refseq Promoter Array | Human | 4x180K | 23,148 RefSeq promoters (-1,300 bp ~ +500 bp of TSS) |
| Arraystar Mouse Refseq Promoter Array | Mouse | 4×180K | 22,327 RefSeq promoters (-1,300 bp ~ +500 bp of TSS) |
| Arraystar Mouse RefSeq Promoter Array | Rat | 4×180K | 15,987RefSeq promoters (-1,300 bp ~ +500 bp of TSS) |
| 芯片名称 | 物种 | 规格 | 覆盖范围 |
|---|---|---|---|
| Arraystar Human ncRNA Promoter Arrar | Human | 4x180K | 27,248 lncRNA promoters (-1,300 bp ~ +500 bp of TSS) + 622 miRNA promoter (-50 kb to mature miRNA) |
| Arraystar Mouse ncRNA Promoter Array | Mouse | 4×180K | 18,552 lncRNA promoters (-1,300 bp ~ +500 bp of TSS) + 346 miRNA promoter (-50 kb to mature miRNA) |
Arraystar Cancer Block芯片
Arraystar Cancer Block芯片是专门为研究癌症相关的block区域而设计的产品,覆盖位于7088个block区域中的2554个蛋白编码基因、8481个长链非编码RNA、463个miRNA genes。通过这款芯片可以检测block区域中的基因和LncRNA的甲基化变化,组蛋白修饰以及转录因子结合情况。此外,结合Arraystar Cancer Block芯片,还可以了解甲基化变化与基因表达水平间的联系。
|
芯片名称 |
物种 | 规格 | 覆盖范围 |
|---|---|---|---|
| Arraystar Human Cancer Block Array | Human | 4x180K | 27MB。位于7088个block区域中的2554个蛋白编码基因、8481个lncRNA、463个miRNA genes |
研究表明:癌症中存在着一些低甲基化的区间(block),这些区间的长度在5kb到10M之间,长度中位值为28kb。1/3的基因转录起点都位于这些block中。此外,这些低甲基化的block与包括LADs*和LOCKs*在内的异染色质区域存在着很大重叠,表明癌症中的甲基化变化与染色质结构改变之间存在着很大的关联性。此外,低甲基化block中包含了绝大部分在肿瘤中表达变异较大的基因。而且,这些区域不仅整体甲基化水平下降,而且相对于正常样品,它们在不同肿瘤样品中甲基化水平变化更加剧烈。这表明:基因在癌症中的平均表达水平和平均甲基化程度固然很重要,它们在不同样品中的均一性也不容忽视,甚至是更加重要。
*:LADs:与核纤层蛋白结合的DNA区域。 LOCKs(large organized chromatin lysine modifications):富含异翻译后修饰(例如:组蛋白H3K9二甲基化修饰)的异染色质区域。
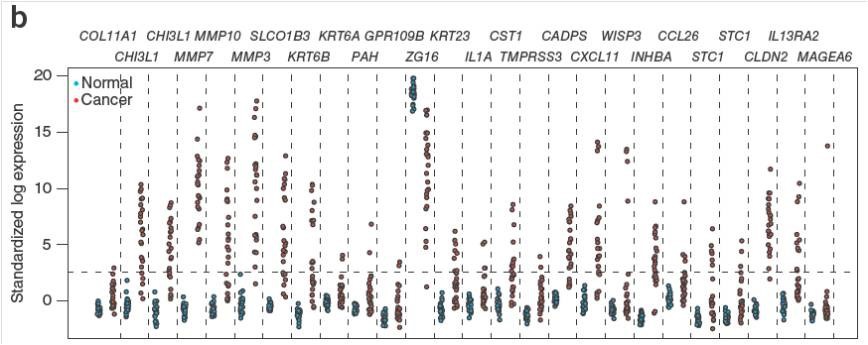
图1. 26个位于低甲基化block区域内的高变异基因的标准表达值(log转化后)。这些基因在肿瘤样品(红色点)中展现出剧烈的表达变异,而在正常样品(蓝色点)中表达变异很小。
Arraystar Cancer DMR芯片是专门为研究癌症相关的差异甲基化区域而设计的产品,覆盖12113个与癌症、组织及细胞分化相关的small DMRs, 以及11380个与这些small DMRs相邻的CpG岛及CpG岛岸。通过这款芯片不但可以检测癌症相关的甲基化变化,还可以了解引起这种甲基化变化的CpG岛边界漂移模式,从而更加全面直观的解析癌症甲基化组。
| 芯片名称 | 物种 | 规格 | 覆盖范围 |
|---|---|---|---|
| Arraystar Human Cancer DMR Array | Human | 4x180K | 51MB 。12113个与癌症、组织及细胞分化相关的small DMRs, 以及11380个与这些small DMRs相邻的CpG岛及CpG岛岸 |
癌症中,除了低甲基化的长区间(block),还存在着许多长度小于5kb的差异甲基化区域(DMRs),被称为small DMRs。大部分癌症或组织特异性的差异甲基化区域(small DMRs)都位于CpG岛边缘2kb以内;相对于CpG岛,这一CpG密度较低区域称之为CpG岛岸(CpG shore)。癌症中,CpG岛边界发生漂移,从而导致CpG岛岸的甲基化水平发生变化:当CpG岛边界向CpG岛内部移动时,CpG岛岸发生超甲基化;当CpG岛边界向外移动时,CpG岛岸发生低甲基化。CpG岛边界的变化导致了基因表达的改变(图1)。
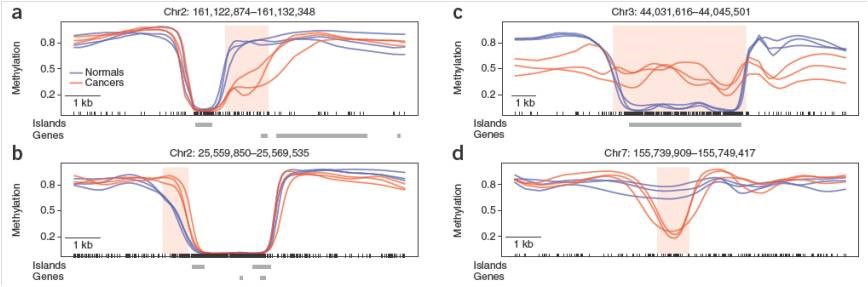
图1. DMR区域丧失甲基化稳定性的模式。图中横轴代表基因组特定区域,纵轴代表相应位点的甲基化程度,蓝色的线代表正常样品,红线代表癌症样品,DMRs区域用粉红色的背景标记。癌症相关的甲基化变化可以分为四类主要的模式:(a)甲基化边界外移;(b)甲基化边界内移;(c)甲基化边界消失;(d)通过去甲基化形成的新的DMR区域。
● 一站式服务:客户只需要提供保存完好的组织或细胞标本,Aksomics的芯片技术服务人员就可为您完成全部实验操作(下图)和数据分析流程,并提供完整的实验报告。
● 严格的质控体系:Aksomics在ChIP-chip每个关键步骤都加入了质控实验(下图)。这些QC数据能够评估每个步骤的实验质量。如果达不到标准,我们会重复实验步骤或者优化实验体系,使得每个样品都能够达到质控标准。
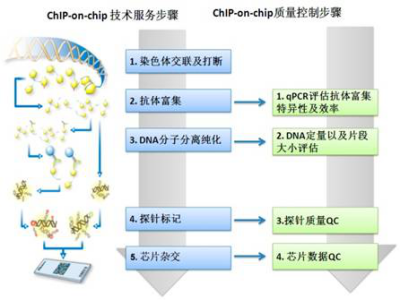
图释:ChIP-chip技术服务流程和质控。
● 丰富的生物信息学分析:除了严谨,可靠的实验体系,Aksomics还有强大的生物信息学团队,为客户提供paper级的图表和深入的数据分析服务
1.样品交联
Formaldehyde处理细胞,交联核酸与DNA结合蛋白
2.超声打断染色质
裂解细胞,并以超声波将染色质破碎成400-500bp片断
3.染色质免疫共沉淀
取步骤2所得片断与ChIP特异抗体结合进行免疫共沉淀,利用免疫磁珠法富集抗体复合物后解交联释放DNA片段,并进行纯化;取部分步骤2所得片断作对照(Input)不进行ChIP实验,但也解交联并纯化
4.线性扩增ChIP及Input DNA片段
使用Sigma WGA Kit对两份DNA样品(ChIP与Input)进行扩增。该步骤使检测的的灵敏度得到大幅度提升,用微量的检测样品就能得到精确的检测结果
5.荧光标记
分别对ChIP-DNA(Cy5)以及Input-DNA(Cy3)进行荧光标记
6.芯片杂交
混合标记后的样本后,ChIP-chip芯片杂交,清洗
7.图像采集和数据分析
使用芯片扫描仪扫描芯片的荧光强度,并将实验结果转换成数值型数据保存,使用专业商用分析软件对原始数据进行分析运算
8.提供实验报告 包括详细的实验方法和芯片实验数据和图表
Scanning Image: Cy3、Cy5荧光扫描图像
Raw Data:探针的荧光信号强度原始数据
Probe Report:包括每个探针的log2(ChIP/Input),此值代表每个探针在ChIP DNA和input DNA中的相对富集强度
Peaks Report:由专业商用软件计算出探针相对强度在统计学意义上显著的Peaks,代表可能的蛋白结合区。Peaks report包含每个Peaks的基因组位置信息,以及Peaks周围基因的相关信息
Summary Report: 提供多样本之间Peaks区域的比较以及汇总以提供参考
→ Interplay of miR-137 and EZH2 contributes to the genome-wide redistribution of H3K27me3 underlying the Pb-induced memory impairment . Cell death & disease . 2019
→ Sox9 transcriptionally regulates Wnt signaling in intestinal epithelial stem cells in hypomethylated crypts in the diabetic state. Stem Cell Research & Therapy. 2017
→ The Mechanism of Environmental Endocrine Disruptors (DEHP) Induces Epigenetic Transgenerational Inheritance of Cryptorchidism. Plos one. 2015
→ Extrauterine growth restriction on pulmonary vascular endothelial dysfunction in adultmale rats: the role of epigeneticmechanisms. Journal of Hypertension. 2014
→ Silencing of RASSF3 by DNA Hypermethylation Is Associated with Tumorigenesis in Somatotroph Adenomas. PLoS One. 2013
→ 125I seed irradiation induces up-regulation of the genes associated with apoptosis and cell cycle arrest and inhibits growth of gastric cancer xenografts. Journal of Experimental & Clinical Cancer Research. 2012
→ MiR-185 Targets the DNA Methyltransferases 1 and Regulates Global DNA Methylation in human glioma. Molecular Cancer. 2011
→ Genome-wide DNA methylation patterns in IVF-conceived mice and their progeny: A putative model for ART-conceived humans. Reprod Toxicol. 2011
→ Effects of DNMT1 silencing on malignant phenotype and methylated gene expression in cervical cancer cells. Journal of Experimental & Clinical Cancer Research. 2011

