

|
单细胞测序技术服务 靶向单细胞测序(lncRNA&mRNA) 单细胞测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
NGS测序技术服务 环状DNA测序(eccDNA测序) |
|
Ribo-seq Ribo seq(ribosome profiling) |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC-seq |
|
蛋白表达定量 DIA定量蛋白质组学 Label free非标定量 TMT标记定量 PRM靶向定量 |
蛋白修饰定量 N-糖基化蛋白组学 乳酸化修饰蛋白质组学 O-GlcNAc修饰蛋白质组学 |
平均每条mRNA上携带的m6A修饰在3个左右[1]。其中,大部分的mRNA只有一个m6A位点,而有些mRNA则可携带20个以上的m6A修饰 [2-5]。假如一个m6A位点附近(上游和下游)没有其他m6A位点,这个位点被定义为“单独m6A位点”。如果一段序列上含有多个m6A位点,相邻的m6A倾向于聚集在非常短的区域内[6]。假如这些m6A聚集的区域长度在20 nt以内,则被定义为“多聚m6A区”。当多个“单独m6A位点”或“多聚m6A区”彼此之间的距离小于100 nt,而跨越的总长度在500 nt以内,这些位点或区域被合并成为一个“m6A位点簇”。目前研究显示,单独或多聚m6A位点在分子层面、细胞层面和生物机体层面都具有显著功能。
单独m6A位点在细胞和机体内的功能
大部分携带m6A修饰的mRNA只包含一个m6A位点[5]。单独m6A位点的分子功能和mRNA翻译起始、延伸等过程有关。此外,这些位点还可调控非编码RNA的活性与降解。
单独m6A修饰在帽非依赖翻译起始中的功能
位于Hsp70 mRNA 5’UTR区域A103位置上的单独m6A修饰足以促进非经典的帽非依赖翻译过程(图1)[7]。在应激条件下,定位到细胞核的阅读蛋白YTHDF2保护该m6A位点不被FTO所去除。然后,翻译起始因子eIF3直接与m6A103位点结合,起始内部翻译,从而使得在应激细胞中帽依赖翻译被整体关停的情况下,HSP70的帽非依赖翻译过程得以启动。
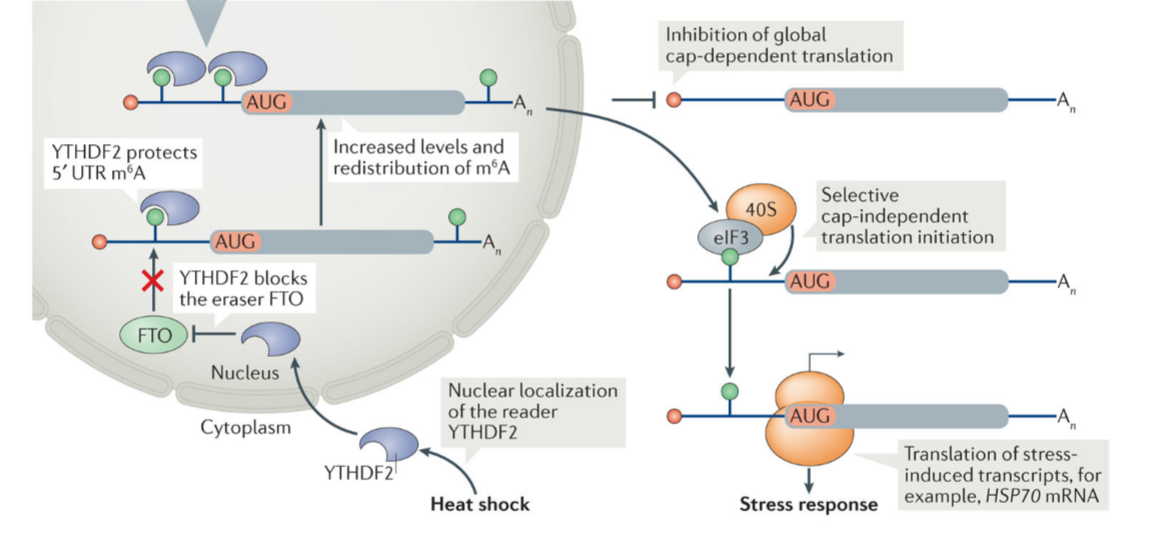
图1. 应激条件下单独m6A修饰促进HSP70 mRNA帽非依赖翻译过程的分子机制[7]。
单独m6A修饰影响mRNA翻译动态
mRNA编码区的单独m6A修饰能影响翻译延伸动态(图2)[8]。密码子上的m6A可降低其配对tRNA的解码速率,并且成为tRNA进入核糖体相应位置和翻译延伸的障碍或者调控者。m6A引起的翻译停滞可能导致蛋白翻译提前终止,并影响共翻译蛋白的折叠、陪伴蛋白的结合以及亚细胞定位信号识别等过程,从而导致不同的蛋白命运或活性[9]。可以想象的是,假如m6A修饰使得密码子编码的氨基酸发生改变,则可产生具备不同功能的变异蛋白,这些变异蛋白虽然占比不大,也有可能产生巨大的生物学效应[8]。

图2. 翻译延伸动态的改变影响共翻译新生蛋白的折叠或与其它陪伴蛋白的相互作用[8]。
单独m6A位点调控lncRNA降解
lnc-Dpf3上的单独m6A位点可被阅读蛋白YTH-DF2所识别,导致lncRNA被降解[10]。趋化因子激活CCR7受体后,lnc-Dpf3上的m6A被移除,阻止了m6A所介导的RNA降解。表达上调的lnc-Dpf3结合并抑制HIF1,从而抑制树突状细胞的迁移和免疫反应(图3)。
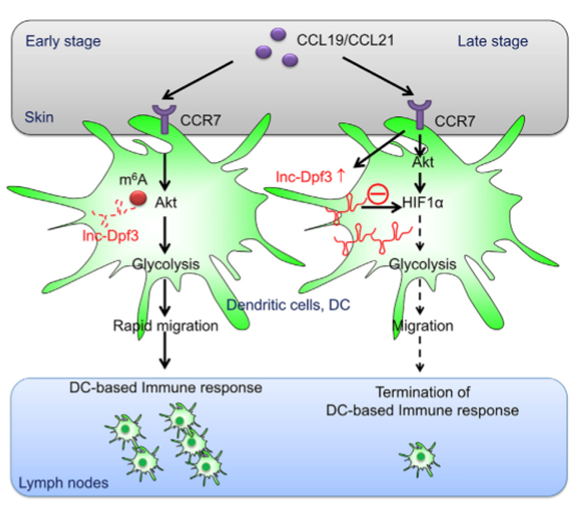
图3. lnc-Dpf3上的单独m6A修饰导致该RNA被降解。在趋化因子刺激下,m6A被移除,lnc-Dpf3稳定性增强。表达上升的lnc-Dpf3抑制HIF1,从而抑制树突状细胞介导的免疫反应[10]。
在另外一个lncRNA案例中,linc1281上的三个单独m6A位点 (A917、A1025和A1056) 为其吸附let-7家族miRNA所必需(图4)[11]。通过扣押这些多潜能miRNA,m6A修饰的linc1281确保了mESC细胞的正确分化。
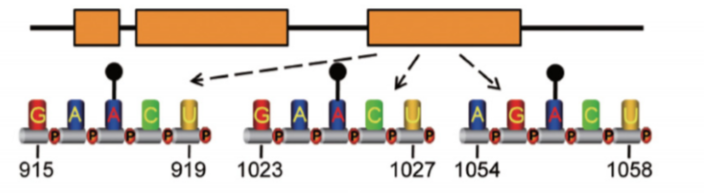
图4. linc1281末端外显子上的单独m6A修饰为其结合let-7家族miRNA所必需,从而作为内源竞争性RNA(ceRNA)调控mESC的分化过程。
28S rRNA上的单独m6A位点影响细胞增殖和肿瘤生长
位于28S rRNA A4220位点上的单独m6A修饰由新发现的m6A甲基转移酶ZCCHC4催化产生。m6A4220修饰为核糖体实现总体最佳翻译效率所必需,从而影响细胞增殖和肿瘤生长[12]。
多聚m6A位点和m6A位点簇在细胞和机体中的功能意义
m6A可参与调控细胞质中mRNA的半衰期,其中,聚集成簇的m6A位点可以显著促进mRNA降解方面的能力。携带m6A修饰的mRNA被m6A阅读蛋白识别,通过相分离过程被靶向至不同的亚细胞区域,从而进入不同的代谢途径。一些lncRNA的活性也依赖于多聚m6A位点。
多聚m6A位点或m6A位点簇通过相分离将mRNA分隔进不同的亚细胞区域
多聚m6A位点或m6A位点簇介导液态相分离过程,将mRNA群体分隔进入不同的亚细胞区域,而单独m6A位点无此功能(图5)[13]。在细胞内或体外条件下,阅读蛋白YTH-DF 1, 2, 3可自发形成液相。多聚甲基化的mRNA作为YTHDF结合的多价平台,被带入不同的相分离形成的亚细胞区域,比如P小体,压力应激颗粒或者神经RNA颗粒,从而产生不同的代谢结果。比如,储存在压力应激颗粒里的多聚甲基化mRNA的翻译过程被抑制,而由YTH-DF2蛋白靶向进入P小体的多聚甲基化mRNA则被降解[14]。单独m6A位点与YTHDF结合的亲和性不足,无法介导此类功能。
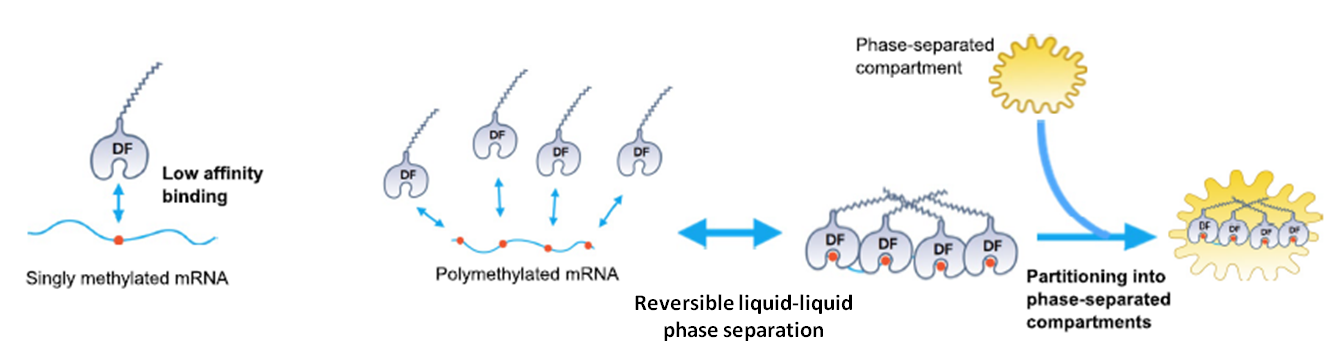
图5. 多聚m6A区域以高亲和力与阅读蛋白YTH-DF1, 2, 3结合,根据代谢目的被分隔进不同的亚细胞区域。单独m6A的低结合亲和性使其不能介导此类功能[13]。
多聚m6A调控mRNA稳定性
在MYC mRNA的CDS 3’端有一段长为250 nt的编码区域不稳定性决定因子(CRD),CRD区域含有多个m6A位点,可被阅读蛋白IGF2BP1, 2, 3识别,从而促进mRNA的稳定性和翻译过程,增加MYC的表达水平(图6)[15]。

图6. m6A阅读蛋白IGF2BP与MYC mRNA编码区域不稳定性决定因子(CRD)上的多聚m6A位点结合,促进mRNA的稳定性,增强MYC蛋白的表达水平。
与X染色体沉默相关的保守区域含有多聚m6A位点
lncRNA-XIST携带78个m6A修饰,远多于其它RNA(图7)。其中,与X染色体沉默活性相关的一段“A重复区域”在进化上十分保守,该保守区域也含有多聚m6A位点。m6A在YTH-DC1蛋白阅读并招募X染色体转录沉默调控因子过程中发挥必备功能 [16]。
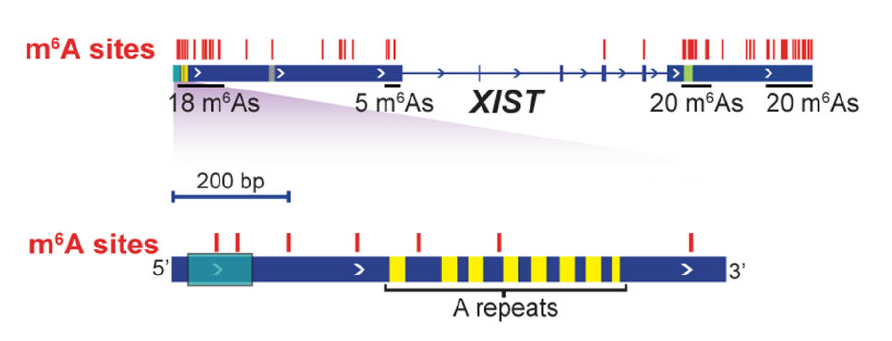
图7. lncRNA-XIST 上的78个m6A位点(红色竖线,图顶)与5’区域的多聚m6A位点的放大详情(图底)。进化保守的 “A重复区域”(黄色)为XIST介导X染色体失活起始过程所必需[16]。
参考文献
[1] Perry, R. P., et al. (1975) "The methylated constituents of L cell messenger RNA: evidence for an unusual cluster at the 5' terminus" Cell 4(4):387-94 [PMID: 1168101]
[2] Meyer, K. D., et al. (2012) "Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons" Cell 149(7):1635-46 [PMID: 22608085]
[3] Dominissini, D., et al. (2012) "Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq" Nature 485(7397):201-6 [PMID: 22575960]
[4] Linder, B., et al. (2015) "Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome" Nat Methods 12(8):767-72 [PMID: 26121403]
[5] Zaccara, S., et al. (2019) "Reading, writing and erasing mRNA methylation" Nat Rev Mol Cell Biol [PMID: 31520073]
[6] Zhang, Z., et al. (2019) "Single-base mapping of m(6)A by an antibody-independent method" Sci Adv 5(7):eaax0250 [PMID: 31281898]
[7] Zhou, J., et al. (2015) "Dynamic m(6)A mRNA methylation directs translational control of heat shock response" Nature 526(7574):591-4 [PMID: 26458103]
[8] Choi, J., et al. (2016) "N(6)-methyladenosine in mRNA disrupts tRNA selection and translation-elongation dynamics" Nat Struct Mol Biol 23(2):110-5 [PMID: 26751643]
[9] Kim, S. J., et al. (2015) "Protein folding. Translational tuning optimizes nascent protein folding in cells" Science 348(6233):444-8 [PMID: 25908822]
[10] Liu, J., et al. (2019) "CCR7 Chemokine Receptor-Inducible lnc-Dpf3 Restrains Dendritic Cell Migration by Inhibiting HIF-1alpha-Mediated Glycolysis" Immunity 50(3):600-615 e15 [PMID: 30824325]
[11] Yang, D., et al. (2018) "N6-Methyladenosine modification of lincRNA 1281 is critically required for mESC differentiation potential" Nucleic Acids Res 46(8):3906-3920 [PMID: 29529255]
[12] Ma, H., et al. (2019) "N(6-)Methyladenosine methyltransferase ZCCHC4 mediates ribosomal RNA methylation" Nat Chem Biol 15(1):88-94 [PMID: 30531910]
[13] Ries, R. J., et al. (2019) "m(6)A enhances the phase separation potential of mRNA" Nature 571(7765):424-428 [PMID: 31292544]
[14] Wang, X., et al. (2014) "N6-methyladenosine-dependent regulation of messenger RNA stability" Nature 505(7481):117-20 [PMID: 24284625]
[15] Huang, H., et al. (2018) "Recognition of RNA N(6)-methyladenosine by IGF2BP proteins enhances mRNA stability and translation" Nat Cell Biol 20(3):285-295 [PMID: 29476152]
[16] Patil, D. P., et al. (2016) "m(6)A RNA methylation promotes XIST-mediated transcriptional repression" Nature 537(7620):369-373 [PMID: 27602518]

