

概述
转运RNA(Transfer Ribonucleic Acid,tRNA)是生物体内含量最为丰富的短链非编码RNA分子。它携带并转运氨基酸,参与蛋白翻译,是连接mRNA与蛋白质的重要桥梁。尽管tRNA广泛存在于生物体内,但不同机体基因组对于特定密码子的偏好性不同,从而导致tRNA谱的差异。密码子偏好性会影响翻译效率和精确性[1-3]。因此,tRNA谱的改变会对多种细胞生理过程产生显著影响。Arraystar公司的nrStar™ Human tRNA Repertoire PCR Array产品包含66对细胞核tRNA引物和22对线粒体tRNA引物,使研究人员可以对tRNA谱进行方便快捷的分析。这款产品囊括了GtRNAdb和tRNAdb所提供的所有反密码子,是研究人类tRNA谱的强有力工具。
tRNA谱与细胞命运
研究表明,tRNA谱的变化会影响细胞发育过程中的命运决定。细胞增殖[4]、细胞分化[4, 5]和细胞凋亡[6]等一系列的生命活都伴随着tRNA水平的变化。细胞维持相关基因与细胞多态性相关基因在密码子使用上存在着明显差异。细胞增殖和细胞分化所诱导的tRNA谱变化分别与细胞维持相关基因和细胞多态性基因相对应(图1)。另外,细胞维持相关基因mRNA在增殖细胞和肿瘤细胞中特异性地诱导表达,提示转录与翻译之间存在直接的关联性[4]。过表达起始tRNA(tRNAiMet)能够显著地影响对整个tRNA表达谱,并且进一步导致细胞代谢能力的提升和增殖能力的加强[5]。tRNA还能够影响细胞色素C介导的凋亡小体形成,参与调节细胞凋亡过程[6]。通过体外显微注射tRNA,研究人员发现tRNA能够抑制细胞色素C介导的凋亡途径[7]。总而言之,tRNA谱能够以多种形式调控细胞的生理状态。
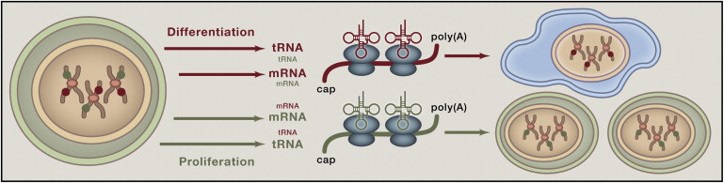
图1. tRNA谱与转录组的变化相适应。
tRNA谱与疾病
tRNA谱不仅能影响多种细胞生理状态,而且在各类疾病发生发展过程中起着重要作用。比如tRNA的表达紊乱可以诱导肿瘤的发生[5]。因此,tRNA表达谱研究对于揭示人类疾病的病理机制有着重要意义。
癌症
Gingold等人发现tRNA表达谱在肿瘤细胞与分化细胞之间存在着显著差异[4]。在分化细胞或者静息状态的细胞中高表达的tRNA,其表达在增殖细胞中受到抑制。反之,高表达于增殖细胞的tRNA,在分化或静息状态的细胞中具有较低的表达量。研究人员还发现,癌细胞能够对tRNA谱进行调整,从而有利于自身的发生发展。通过比较乳腺癌与正常乳腺组织中的tRNA表达水平,Pavon-Eternod发现乳腺癌细胞核与线粒体中的tRNA发生了显著改变,提示我们tRNA能够作为乳腺癌的分子标记物[12]。最近,Goodarzi等人也证实乳腺癌细胞在获得转移能力的过程中,某些特定的tRNA表达水平上升。他们认为tRNAGlu-UUC和tRNAArg-CCG表达水平变化可能是乳腺癌转移的诱发因素之一。进一步的研究则表明tRNAGlu-UUC可以促进EXOSC2和GRIPAP1的蛋白表达,从而促发肿瘤的转移[8]。总之,tRNA的表达谱紊乱对肿瘤的发生发展有着深远影响[5, 8-15]。
亨廷顿舞蹈症
亨廷顿舞蹈症是一种发病率较高的遗传性神经退行性疾病,由亨廷顿蛋白末端多聚谷氨酰胺增多而引起。对亨廷顿舞蹈症病人的脑部组织进行分析后发现,在多聚谷氨酰胺中存在多聚丙氨酸和多聚丝氨酸的踪迹。这些多聚氨基酸,可能是由于编码谷氨酰胺的CAG阅读框发生移码,产生编码丙氨酸的GCA阅读框或者编码丝氨酸的AGC阅读框。Girstmair等人发现,tRNAGln-CUG表达不足是诱发移码翻译错误的关键所在。另外,谷氨酰胺与丙氨酸的比例会影响移码翻译蛋白的形态。这些结果表明tRNA的表达紊乱及其导致的移码翻译可能与亨廷顿舞蹈症的发生以及疾病个体异质性有关[16]。
病毒感染
众所周知,病毒蛋白质合成完全依赖于宿主的翻译体系,包括tRNA库的使用。因此一般认为,病毒选择性地适应宿主细胞的tRNA库。由于宿主密码子的使用情况决定了宿主的tRNA库,病毒蛋白合成只有在使用与宿主基因高度相似的密码子时,才具有高翻译效率。然而,在很多情况下,病毒基因的密码子使用并不与宿主基因相匹配。Pavon-Eternod发现甲型流感病毒(influenza A)和牛痘病毒(vaccinia virus)能够调整被感染宿主的tRNA库来适应它们自身基因的翻译[17]。还有研究表明,艾滋病病毒(HIV-1)早期基因与宿主基因的密码子使用高度相似,而艾滋病病毒的晚期基因则非如此。病毒感染后期,宿主tRNA库更适用于艾滋病病毒的蛋白翻译[18]。因此,病毒能够调整宿主tRNA库,以此来适应自身蛋白质合成的需求。
参考文献
[1] Drummond DA, Wilke CO. Mistranslation-induced protein misfolding as a dominant constraint on coding-sequence evolution. Cell 2008;134:341-52.
[2] Plotkin JB, Kudla G. Synonymous but not the same: the causes and consequences of codon bias. Nature reviews Genetics 2011;12:32-42.
[3] Shah P, Gilchrist MA. Explaining complex codon usage patterns with selection for translational efficiency, mutation bias, and genetic drift. Proceedings of the National Academy of Sciences of the United States of America 2011;108:10231-6.
[4] Gingold H, Tehler D, Christoffersen NR, Nielsen MM, Asmar F, Kooistra SM, et al. A dual program for translation regulation in cellular proliferation and differentiation. Cell 2014;158:1281-92.
[5] Pavon-Eternod M, Gomes S, Rosner MR, Pan T. Overexpression of initiator methionine tRNA leads to global reprogramming of tRNA expression and increased proliferation in human epithelial cells. Rna 2013;19:461-6.
[6] Mei Y, Stonestrom A, Hou YM, Yang X. Apoptotic regulation and tRNA. Protein & cell 2010;1:795-801.
[7] Mei Y, Yong J, Liu H, Shi Y, Meinkoth J, Dreyfuss G, et al. tRNA binds to cytochrome c and inhibits caspase activation. Molecular cell 2010;37:668-78.
[8] Goodarzi H, Nguyen HC, Zhang S, Dill BD, Molina H, Tavazoie SF. Modulated Expression of Specific tRNAs Drives Gene Expression and Cancer Progression. Cell 2016;165:1416-27.
[9] Berns A. A tRNA with oncogenic capacity. Cell 2008;133:29-30.
[10] Waldman YY, Tuller T, Sharan R, Ruppin E. TP53 cancerous mutations exhibit selection for translation efficiency. Cancer research 2009;69:8807-13.
[11] Kushner JP, Boll D, Quagliana J, Dickman S. Elevated methionine-tRNA synthetase activity in human colon cancer. Proceedings of the Society for Experimental Biology and Medicine Society for Experimental Biology and Medicine 1976;153:273-6.
[12] Marshall L, Kenneth NS, White RJ. Elevated tRNA(iMet) synthesis can drive cell proliferation and oncogenic transformation. Cell 2008;133:78-89.
[13] Pavon-Eternod M, Gomes S, Geslain R, Dai Q, Rosner MR, Pan T. tRNA over-expression in breast cancer and functional consequences. Nucleic acids research 2009;37:7268-80.
[14] Zhou Y, Goodenbour JM, Godley LA, Wickrema A, Pan T. High levels of tRNA abundance and alteration of tRNA charging by bortezomib in multiple myeloma. Biochemical and biophysical research communications 2009;385:160-4.
[15] Begley U, Sosa MS, Avivar-Valderas A, Patil A, Endres L, Estrada Y, et al. A human tRNA methyltransferase 9-like protein prevents tumour growth by regulating LIN9 and HIF1-alpha. EMBO molecular medicine 2013;5:366-83.
[16] Girstmair H, Saffert P, Rode S, Czech A, Holland G, Bannert N, et al. Depletion of cognate charged transfer RNA causes translational frameshifting within the expanded CAG stretch in huntingtin. Cell reports 2013;3:148-59.
[17] Pavon-Eternod M, David A, Dittmar K, Berglund P, Pan T, Bennink JR, et al. Vaccinia and influenza A viruses select rather than adjust tRNAs to optimize translation. Nucleic acids research 2013;41:1914-21.
[18] van Weringh A, Ragonnet-Cronin M, Pranckeviciene E, Pavon-Eternod M, Kleiman L, Xia X. HIV-1 modulates the tRNA pool to improve translation efficiency. Molecular biology and evolution 2011;28:1827-34.