

|
基因芯片技术服务 DNA甲基化芯片 DNA羟甲基化芯片 ChIP-chip |
NGS测序技术服务 DNA甲基化测序 DNA羟甲基化测序 染色质免疫共沉淀测序 |
PCR技术服务 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR |
| TMT标记定量技术 非标定量技术 |
蛋白修饰 TMT标记定量磷酸化 非标定量磷酸化 |
RNA/蛋白-蛋白相互作用 RNA-蛋白相互作用 蛋白-蛋白相互作用 |
相关服务
nrStar™ ncRNA PCR芯片 LncRNA实时定量PCR RNA-蛋白相互作用 RNA测序 DNA甲基化测序 DNA羟甲基化测序 DNA甲基化芯片 DNA羟甲基化芯片 全基因表达谱芯片相关产品
nrStar™ Human Functional LncRNA PCR 芯片 Arraystar SYBR® Green Real-time qPCR Master Mix rtStar™ First-Strand cDNA Synthesis Products相关资源
Cancer Discovery:Arraystar lncRNA芯片应用于胃癌研究 Cell子刊:应用Arraystar LncRNA芯片研究宿主抗病毒反应的文章 Cell Metabolism:应用Arraystar LncRNA芯片,研究小鼠代谢疾病长链非编码RNA (LncRNA)是一类长度超过200nt的RNA,它们本身并不编码蛋白,而是以RNA的形式在多种层面上(表观遗传调控、转录调控以及转录后调控等)调控基因的表达水平。近年来的研究表明:LncRNA广泛参与各种生物学过程,LncRNA的异常表达与包括癌症在内的多种疾病密切相关。通过LncRNA芯片,研究人员能够快速高通量的获得与特定生物学过程或者疾病相关的LncRNA的表达变化,从而为后续的LncRNA功能研究或生物标志物筛选提供极大的便利。
Arraystar LncRNA芯片可同时检测LncRNA和mRNA,这一突破性芯片平台的构建,使Arraystar成为LncRNA研究领域的先驱和领导者。迄今为止,康成客户采用Arraystar LncRNA芯片已发表的SCI论文已超180篇,其中多篇发表在Cancer Cell,Mol Cell,Hepatology,Blood,EMBO等国际顶尖杂志上。
康成生物是Arraystar中国区唯一代理商,独家为您提供Arraystar公司长链非编码RNA芯片全程一站式技术服务。您只需要提供保存完好的组织或细胞标本,康成的芯片技术服务人员就可为您完成全部实验操作,并提供完整的实验报告。同时,根据您的研究需要,康成还提供各种深入数据挖掘服务。
Arraystar公司LncRNA芯片产品列表
| 服务 | 芯片 | 规格 | 描述 |
|---|---|---|---|
| Human LncRNA Microarray Service | Human LncRNA Array V4.0 | 8x60K | LncRNAs (40173) + coding genes (20730) |
| Mouse LncRNA Microarray Service | Mouse LncRNA Array V3.0 | 8×60K | LncRNAs (35923) + coding genes (24881) |
| Rat LncRNA Microarray Service | Rat LncRNA Array V2.0 | 4×44K | LncRNAs (13611) + coding genes (24626) |
• 检测lncRNA表达量灵敏度高、技术最成熟:lncRNA表达水平比mRNA低,芯片比RNA-seq更适合对低丰度RNA分子的检测。
• 收录lncRNA更新最及时,覆盖最全面、最可靠:可同时检测lncRNA和编码蛋白的mRNA。
• 系统而实用的lncRNA注释:注释LncRNA基因组信息、分类及潜在的调控机制,为深入研究lncRNA复杂生物学功能提供参考信息。
• 转录本特异型的探针设计:Arraystar lncRNA芯片采用转录本特异性的探针设计,对不同转录本检测更准确、特异性更高。
• 提供丰富的数据信息:用于调控型lncRNA与编码蛋白的mRNA之间共表达及关联研究。
1.样品总RNA抽提
若实验对象为组织样品,取适量(50-100mg)新鲜组织样品或正确保存的组织样品,加1ml的RNA抽提试剂Trizol(Invitrogen),匀浆后抽提RNA。
若实验对象为细胞样品,每份样品取1×106~1×107细胞,完全吸去培养液后加1ml的RNA抽提试剂Trizol(Invitrogen),裂解后抽提RNA。
2. RNA质量检测
使用Nanodrop测定RNA在分光光度计260nm、280nm和230nm的吸收值,以计算浓度并评估纯度。
使用甲醛变性琼脂糖凝胶电泳检测RNA纯度及完整性
3. cDNA样品合成和标记
4.标记效率质量检测
使用Nanodrop检测荧光标记效率,以保证后续芯片实验结果的可靠性。
5.芯片杂交
在标准条件下将标记好的探针和高密度芯片进行杂交。
6.图像采集和数据分析
使用GenePix 4000B芯片扫描仪扫描芯片的荧光强度,并将实验结果转换成数字型数据保存,使用配套软件对原始数据进行分析运算。
7.提供实验报告
芯片扫描图
实验方法中英文报告
RNA质检报告
芯片数据结果报告,包括差异表达LncRNA列表,差异表达基因列表
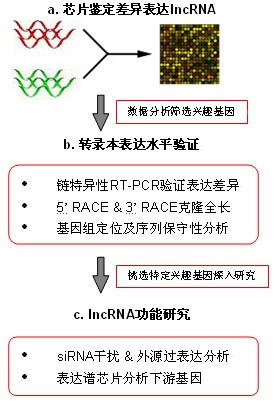
基本数据分析
1. 差异LncRNA的筛选
对于每个实验组有三次及三次以上生物学重复的实验设计,我们应用T检验筛选任意两个实验组之间差异表达的LncRNA,方差分析(ANOVA)则用于从三个以上实验组筛选差异表达的LncRNA,并以邻近(<100kb)mRNAs进行注释。康成生物除了提供常规的通过p值筛选的差异LncRNA,还提供通过 FDR筛选的差异LncRNA。与p值筛选相比,FDR筛选对多重筛选过程中的假阳性率进行控制,是一种更加严谨的筛选方法,更适合于生物学重复较多的临床样本。
适用范围:一般用于两组或多组实验状态下的比较,建议每组实验至少有3次生物学重复。

2. 差异表达的LncRNA的聚类图
为了全面而直观地展示样品之间的关系及基因表达差异情况,将表达基因做层次聚类分析。用挑选的基因的表达情况来计算样品直接的相关性。一般来说,同一类样品能通过聚类出现在同一个簇(Cluster)中,聚在同一个簇的基因可能具有类似的生物学功能。
适用范围:两组或多组样品的表达谱数据

3. 差异LncRNA的邻近基因分析
很多LncRNA是通过调控邻近发挥生物学功能,因此通过邻近基因的分析可以为后续LncRNA的功能研究提供线索。康成生物将差异LncRNAs数据与其临近(< 100 kb)mRNAs的差异表达数据整合,以期提供LncRNAs的功能推断。
适用范围:两组或多组数据比较获得的差异LncRNA
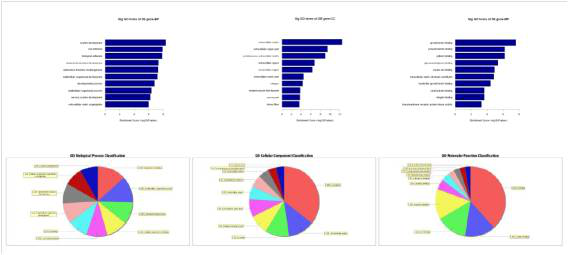
1) GO分析
Gene Ontology(简称GO)是基因功能国际标准分类体系。GO可分为分子功能(Molecular Function),生物过程(Biological Process)和细胞组成(Cellular Component)三个部分。康成生物对位于差异LncRNA附近(< 100 kb),并且差异表达的蛋白编码基因进行了GO分析。这一方面便于客户从整体上了解差异LncRNA所涉及的GO条目,另一方面也为客户挑选LncRNA提供了线索:客户可以挑选与感兴趣的GO条目相关的差异LncRNA进行后续研究。
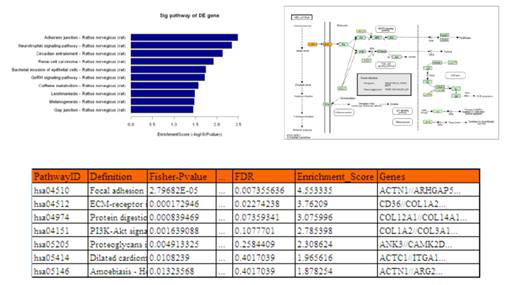
2) Pathway分析
康成生物对位于差异LncRNA附近(< 100 kb),并且差异表达的蛋白编码基因进行了Pathway分析。这一方面便于客户从整体上了解差异LncRNA所参与的信号通路,另一方面也为客户挑选LncRNA提供了线索:客户可以挑选与感兴趣的信号通路有关的差异LncRNA进行后续研究。
4. LncRNAs的子类分析
适用范围:两组或多组数据比较获得的差异LncRNA
(1)差异antisence LncRNAs与相应mRNAs联合分析
在LncRNA中,研究得比较深入的是反义(Antisense) LncRNA,有超过30%的已注释的人类转录本有相应的反义LncRNA。这些反义LncRNA通过多种机制在转录水平或转录后水平调控相应的正义(sense)mRNA,从而发挥生物学功能。如下图中反义LncRNA诱导染色质和DNA的表观遗传改变,从而影响相应的正义mRNA的表达。康成生物将差异antisense LncRNAs与相应的sense mRNAs信息进行整合,以推断LncRNAs的功能.
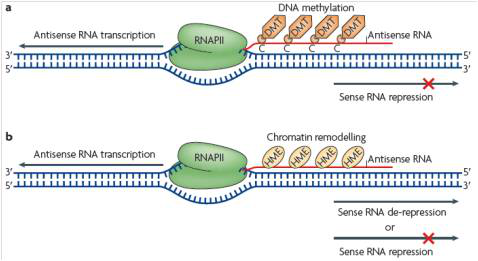
*图释:反义LncRNA诱导染色质和DNA的表观遗传改变,从而影响相应的正义mRNA的表达。

(2)差异Enhancer LncRNAs与相应mRNAs联合分析
LncRNA可以发挥类似增强子的功能,增强邻近蛋白质编码基因的表达,从而在发育和分化等过程中发挥关键作用。康成生物对Ørom UA等人在人类细胞系中发现的3千多条具有增强子功能的LncRNA 进行了详细的注释,并将差异的Enhancer LncRNAs与相应的mRNAs(<300kb)进行了整合,以帮助客户推断这些LncRNAs的功能。
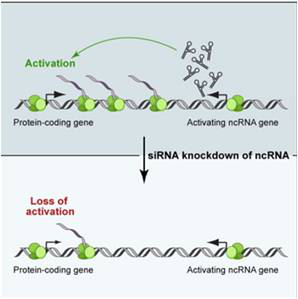
*图释:LncRNA作为增强子,增强邻近蛋白质编码基因表达水平;下图:经siRNA敲除ncRNA后,邻近蛋白质编码基因表达水平下降

(3)差异LincRNAs与相应mRNAs联合分析
lincRNAs是目前研究的热点之一。康成生物根据Rinn等构建的lincRNAs数据库,将芯片中符合标准的差异LncRNA挑选出来与相应mRNA(< 300 kb)进行联合分析,以推断lincRNA功能。

高级数据分析
1. CNC(coding-noncoding gene co-expression)分析
CNC分析是一种通过LncRNA和mRNA共表达数据,将LncRNA与mRNA联系起来的分析方法。通过CNC分析可以发现与某个LncRNA具有相同表达模式的mRNA,通过这些mRNA的功能,可以将LncRNA与特定信号通路或疾病状况联系起来,从而便于预测LncRNA的功能,并揭示其作用机制。
适用范围:芯片数量≥6张
康成生物CNC分析结果展示,下图数据来源于康成客户已发表的文献(Hepatology,影响因子:12.003):
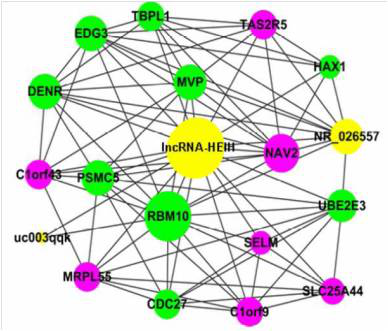
*图释:LncRNA-HEIH在HCC中的共表达网络。黄色的节点代表LncRNA,绿颜色的节点代表与肿瘤生长和药物耐受相关的蛋白编码基因,紫色节点代表功能未知的蛋白编码基因。
2. 生物标志物分析
康成生物还为客户提供包括课题设计到最终数据分析在内的一站式biomarker分析,以最常见的筛选预后biomarker为例,我们首先通过Cox回归模型筛选预后相关的LncRNA,然后通过Logistic regression,Nearest Template Prediction (NTP)等多种统计学方法构建预测模型,然后从中挑选出效果最好的预测模型用于后续分析。无论是LncRNA的筛选,还是预测模型的建立,都及其严谨和科学,能最大限度的为客户发表文章提供帮助。
1. LncRNA生物标记物案例分析
LncRNA胰腺癌诊断生物标记物筛选(Expression profile of long non-coding RNAs in pancreatic cancer and their clinical significance as biomarkers. Oncotarget, 2015)
该研究首先通过Arraystar LncRNA芯片对胰腺癌和慢性胰腺炎组织(8 vs. 4)中的LncRNA表达谱进行了检测,找到了33个在胃癌中差异表达的LncRNA。之后作者从上调最明显的LncRNA中挑选了三个(XLOC_006390, HOTTIP-005和RP11-567G11.1),在144个临床组织样本中进行了qPCR验证,锁定了HDRF和RDRF(HOTTIP-005和RP11-567G11.1来源的RNA片段)。结合qPCR的数据和临床信息,进一步在胰腺癌(n=122)和正常对照(n=127)的血浆样本中验证了HDRF和RDRF的诊断潜能。实验证明,HDRF和RDRF与胰腺癌发生密切相关,是潜在的诊断和预后marker。
2. LncRNA生物标记物和功能机制联合研究案例
LncRNA-NKILA作为乳腺癌预后生物标记物和其在NF-κB信号通路及乳腺癌转移中的功能机制研究(A Cytoplasmic NF-κB Interacting Long Noncoding RNA Blocks IκB Phosphorylation and Suppresses Breast Cancer Metastasis [J]. Cancer Cell, 2015) IF: 23.523
该研究首先通过Arraystar LncRNA芯片检测了多种炎症因子((TNF)-a, IL-1b和LPS)处理的乳腺癌细胞(MDA-MB-231)中的lncRNA表达谱,并找出了23个上调10倍的lncRNAs和28个下调10倍的lncRNAs。其中,NKILA在三组炎症因子处理中均上调12倍以上,因此,作者选择NKILA作为研究对象。临床研究中,作者利用qRT-PCR在上千例乳腺癌组织中检测NKILA的表达情况,并结合临床数据进行分析,结果表明:NKILA高表达与乳腺癌预后不良显著相关,是潜在的乳腺癌预后marker。功能机制研究中,作者在细胞系和小鼠模型中进行NKILA的敲除和过表达,验证其生物学功能,并进一步通过RIP、RNA pull down等实验探索其分子机制,结果表明:NKILA能够与NF-κB通路相互作用,通过结合NF-κB/IκB抑制了IKK诱导的IκB磷酸化,使得NF-κB信号通路失活,从而抑制乳腺癌肿瘤转移。
→ A novel lncRNA GClnc1 promotes gastric carcinogenesis and may act as a modular scaffold of WDR5 and KAT2A complexes to specify the histone modification pattern. Cancer discovery. 2016
→ Non-coding RNAs participate in the regulatory network of CLDN4 via ceRNA mediated miRNA evasion. Nature Communications. 2017
→ Long Non-coding RNA DILC Represses Self-renewal of Liver Cancer Stem Cells via Inhibiting Autocrine IL-6/STAT3 Axis. Journal of Hepatology 2016
→ Long non-coding RNA LINC00092 acts in cancer-associated fibroblasts to drive glycolysis and progression of ovarian cancer Cancer Research 2017
→ A novel oncogene, promotes proliferation and metastasis by activating the VEGF pathway in gastric cancer. Oncogene. 2017
→ The TMSB4 Pseudogene LncRNA Functions as a Competing Endogenous RNA to Promote Cartilage Degradation in Human Osteoarthritis. Molecular Therapy. 2016
→ A novel lncRNA uc. 134 represses hepatocellular carcinoma progression by inhibiting CUL4A-mediated ubiquitination of LATS1. Journal of Hematology & Oncology. 2017
→ Up-regulation of lncRNA CASC9 promotes esophageal squamous cell carcinoma growth by negatively regulating PDCD4 expression through EZH2. Molecular Cancer. 2017
→ Long non-coding RNA LINC00161 sensitises osteosarcoma cells to cisplatin-induced apoptosis by regulating the miR-645-IFIT2 axis. Cancer Letters. 2016
→ Circulating “lncRNA OTTHUMT00000387022” from Monocytes as a Novel Biomarker for Coronary Artery Disease. Cardiovascular Research. 2016
→ DC-SIGNR by influencing the lncRNA HNRNPKP2 upregulates the expression of CXCR4 in gastric cancer liver metastasis. Molecular Cancer. 2017
→ Microarray analysis of long noncoding RNA and mRNA expression profiles in human macrophages infected with Mycobacterium tuberculosis. Scientific Reports. 2016
→ LncRNAs expression profiling in normal ovary, benign ovarian cyst and malignant epithelial ovarian cancer. Scientific Reports. 2016

