

|
基因芯片技术服务 DNA甲基化芯片 DNA羟甲基化芯片 ChIP-chip |
NGS测序技术服务 DNA甲基化测序 DNA羟甲基化测序 染色质免疫共沉淀测序 |
PCR技术服务 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR |
|
蛋白相对定量 TMT标记定量技术 非标定量技术 |
蛋白修饰 TMT标记定量磷酸化 非标定量磷酸化 |
RNA/蛋白-蛋白相互作用 RNA-蛋白相互作用 蛋白-蛋白相互作用 |
全基因组基因表达谱的检测对疾病研究有非常重要的意义。一方面,基因表达谱的检测可以应用于疾病生物标记物的筛选。另一方面,基因表达谱的检测可以为基因功能研究提供线索,有助于揭示疾病发生发展的分子机制。
康成生物为您提供全基因组表达谱芯片技术服务,您只需要提供保存完好的组织或细胞标本,康成的芯片技术服务人员就可为您完成全部实验操作,并提供完整的实验报告。同时,根据您的研究需要,康成还提供基因网络关系分析、分子标志物筛选分析等数据挖掘服务。
| 芯片名称 | 种属 | 规格 | 基因数 | Database Source |
|---|---|---|---|---|
| Whole human genome | 人 | 4 x 44K | ~41,000 | Goldenpath, Ensembl, Unigene, Human Genome (Build 33), Refseq, GebBank |
| whole mouse genome | 小鼠 | 4 x 44K | ~41,000 | USC mRNA known genes, Natl. Institute on Aging, Genbank, Unigene, Refseq, Ensembl, RIKEN |
| whole rat genome | 大鼠 | 4 x 44K | ~41,000 | Ensembl, UCSC Goldenpath, Unigene, Refseq, Genbank |
| SurePrint G3 Human Gene Expression 8x60K v2 Microarray | 人 | 8 x 60 K | ~50,000 | RefSeq, Ensemble, Unigene, GenBank, Broad Institute Human lincRNA and TUCP catalog |
| SurePrint G3 Mouse GE 8x60K Microarray | 小鼠 | 8 x 60 K | ~40,000 | RefSeq, Ensembl, Unigene, GenBank, RIKEN |
| SurePrint G3 Rat GE 8x60K Microarray | 大鼠 | 8 x 60 K | ~30,000 | RefSeq, Ensembl,Unigene, GenBank |
Agilent还提供多类物种的表达谱芯片:
动物:兔、猪、斑马鱼、果蝇等 植物:水稻、玉米、拟南芥、烟草等
微生物:大肠杆菌、酵母等
● 原位喷墨合成技术:原位合成的探针使得芯片平台具有极高的稳定性,喷墨合成技术可以大规模、灵活地合成探针,为客户提供最新的芯片设计。
● 60mer的长探针:与短探针相比,具有更高的信噪比、灵敏度和特异性。
● 权威公认:2006年美国FDA发表的主流芯片平台评估报告MAQC中,Agilent array的C-V值、检测基因数目、与Taqman探针的重复性均处于国际同行业领先水平。
1. 样品RNA抽提:
a. 实验对象为组织样品,取适量(50-100mg)新鲜组织样品或正确保存的组织样品,使用BioPulverizerTM冰冻粉碎组织,加1ml的RNA抽提试剂TRIzol(Invitrogen),使用Mini- Bead-Beater-16匀浆后抽提RNA
b. 实验对象为细胞样品,每份样品取1×106~1×107细胞,加1ml的RNA抽提试剂TRIzol(样品为贴壁细胞,每10cm2培养皿TRIzol使用量为1ml),裂解后抽提RNA
2. RNA质量检测:
a. 使用Nanodrop测定RNA在分光光度计260nm、280nm和230nm的吸收值,以计算浓度并评估纯度
b. 用甲醛电泳试剂进行变性琼脂糖凝胶电泳,检测RNA纯度及完整性
c. 提供RNA QC报告
注意:用于芯片检测的RNA样品,必须是高质量的,完整的,没有RNase污染(降解的样品不能用于标记和芯片检测),没有基因组污染。
3. cDNA/aRNA样品合成和标记
4. 标记效率质量检测:
使用Nanodrop检测荧光标记效率,标记效率合格以保证后续芯片实验结果的可靠性
5. 芯片杂交:
在标准条件下将标记好的探针和高密度基因组芯片进行杂交
6. 图像采集和数据分析:
使用GenePix 4000B芯片扫描仪扫描芯片的荧光强度,并将实验结果转换成数字型数据保存,使用配套软件对原始数据进行分析运算
7. 提供实验报告 包括详细的实验方法以及芯片实验数据和图表,以双色标记实验为例:
Scanning Image: Cy3、Cy5荧光扫描图像
Scatter Plot: 散点图,X轴为Control(Cy3)数据值,Y轴为Exp(Cy5)数据值,表示芯片上两通道数据总体分布集中趋势;
MA-plot: MA图,X轴为A值[log2(Exp)+log2(Control)]/2,代表点的整体信号强度,Y轴为M值log2(Exp)-log2(Control)表示点的两通道信号差,可由MA图观察芯片数据是否存在强度依赖的系统偏移以及信号差异点的比例
Raw Data: 探针的扫描荧光信号强度原始数据
Normalized Ratio: 原始数据经过统计学方法标准化后的比值的对数,log2(Exp/ Control)
p –value: T-test统计分析显著差异表达的基因,p-value越小,该基因在两样本间差异表达越显著
Significant Up & Down Regulated Gene List: 给出Fold Change>=2,p-value<0.05的基因列表
1.差异基因筛选
对于每个实验组有三次以上生物学重复的实验设计,我们应用T检验筛选任意两个实验组之间差异表达的基因。方差分析(ANOVA)则用于从三个以上实验组筛选差异表达的基因。康成生物除了提供常规的通过p值筛选的差异基因,还提供通过FDR筛选的差异基因。与p值筛选相比,FDR筛选对多重筛选过程中的假阳性率进行控制,是一种更加严谨的筛选方法。
适用范围:一般用于两组或多组实验状态下的比较,建议每组实验至少有3次生物学重复。
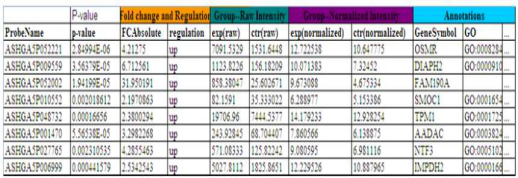
一般筛选标准:Fold Change>=2,p-Value<=0.05
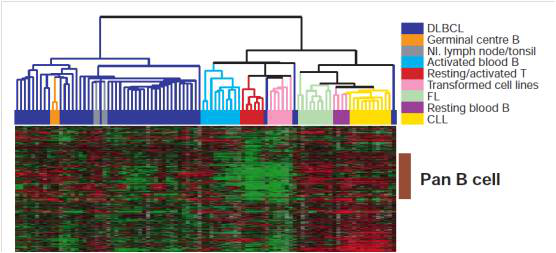
2.聚类分析
为了全面而直观地展示样品之间的关系及基因表达差异情况,将表达基因做层次聚类分析。用挑选的基因的表达情况来计算样品直接的相关性。一般来说,同一类样品能通过聚类出现在同一个簇(Cluster)中,聚在同一个簇的基因可能具有类似的生物学功能。
适用范围:两组或多组样品的表达谱数据
3.GO分析
Gene Ontology(简称GO)是基因功能国际标准分类体系。GO可分为分子功能(Molecular Function),生物过程(Biological Process)和细胞组成(Cellular Component)三个部分。GO-Analysis对差异基因等按GO分类,并对分类结果进 行基于离散分布的显著性分析、误判率分析、富集度分析,得出与实验目的有显著联系的、低误判率的、靶向性的基因功能分类,该分类即导致样本性状差异的最重要的功能差别。通过该分析有可能找到导致性状变化的重要功能,并且找到该功能所对应的基因。
适用范围:两组或多组数据比较获得的差异基因

4.Pathway分析
信号通路(Pathway)是多个基因间相互作用,共同调节细胞功能和代谢活动的过程。而信号通路分析是通过对差异基因按照Pathway的主要公共数据库KEGG来进行分类,得到与实验目的有显著联系的Pathway类别。
适用范围:两组或多组数据比较获得的差异基因

高级数据分析展示
1. 趋势分析(STEM-Analysis)
从芯片数据中挑选出一群随时间或浓度显著性变化的基因,并同时给出相应的统计检验值来帮助我们筛选具有显著性的基因群体。
适用范围:实验设计为随时间或浓度梯度变化的实验分组数据,并且时间点或浓度梯度不超过8个。

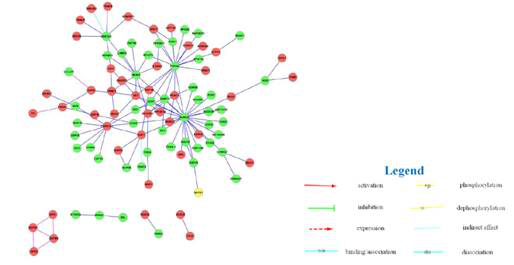
2. 基因间相互作用网络分析
通常在表达谱芯片分析后会得到大量的差异表达基因。我们利用主流的几个蛋白相互作用数据库(BIOGRID, INTACT, MINT, DIP, BIND, HPRD)的数据,来获取这些差异表达基因之间,或差异表达基因与其它相关联基因之间蛋白-蛋白相互作用网络,进而得到在网络中处于核心地位的关键基因。
适用范围:两组或多组数据比较获得的差异基因,用于分析的基因数目一般情况下应小于100个。
联合分析
(1)mRNA表达谱& miRNA表达谱联合分析
根据表达谱芯片结果和miRNA芯片结果联合分析,进一步定位差异miRNA发挥功能的途径以及相应的差异表达靶基因可能引起的生物学通路变化。首先利用差异表达的miRNA找寻靶基因,将其与表达谱中的差异表达基因取交集。这样能找到既是差异miRNA的靶基因又在表达谱芯片中差异表达的基因,可以进一步缩小miRNA可能影响的基因的范围,能够更加准确地研究这些基因所发挥的生物学功能。
适用范围:同时具有mRNA表达谱数据和miRNA表达谱数据的样品,一般情况下用于分析的差异miRNA数目应小于10
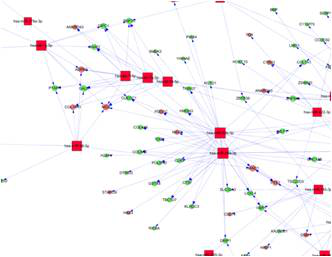
*图释:其中方块节点为miRNA,圆形节点为gene。箭头表示预测的靶向关系。红色的点是芯片中上调的节点,绿色的点是芯片中下调的节点。节点的大小与边的多少成正相关。边越多节点越大。
(2)mRNA表达谱&甲基化数据联合分析
帮助客户找寻典型的甲基化调控基因:典型情况是找寻mRNA表达上调且对应Promoter区域甲基化程度减少的基因,在联合分析基础上可以展示甲基化与mRNA层面的相关性,找到与甲基化负相关的mRNA。mRNA表达下调且对应Promoter区域甲基化程度增加的基因罗列成表格形式。
适用范围:同时具有mRNA表达谱和甲基化谱的样品
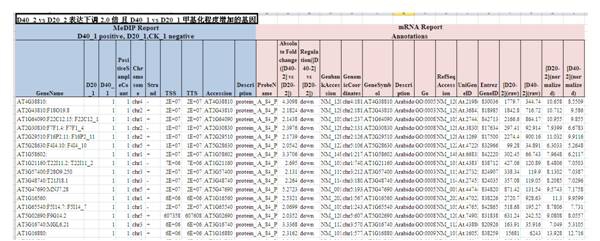
1.生物标记物案例
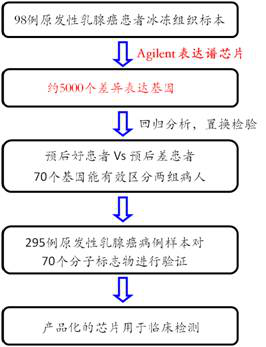
乳腺癌预后生物标记物筛选(Van't Veer L J. et al. Nature, 2002. Van De Vijver M J. et al. New England Journal of Medicine, 2002)
乳腺癌是女性最常见的恶性肿瘤之一,原发性乳腺癌患者往往会有不同的预后,而传统的方法(淋巴结状态、组织学分级)不能作出准确预测。为了解决这个问题,作者使用特定基因表达特征预测原发性乳腺癌的预后。使用Agilent表达谱芯片对98例原发性乳腺癌患者进行检测,5000个基因表达水平发生显著变化。通过生物信息学手段:回归分析和置换检验,发现其中70个差异表达基因能够有效区分预后好和预后差的原发性乳腺癌患者。进一步,在295例原发性乳腺癌病例样本中进行验证,这70个基因的表达特征能够很好地预测原发性乳腺癌患者的预后。值得一提的是,2007年,基于该70个基因的表达谱芯片获得FDA认证,用于原发性乳腺癌患者的临床预后。
技术路线
结果展示

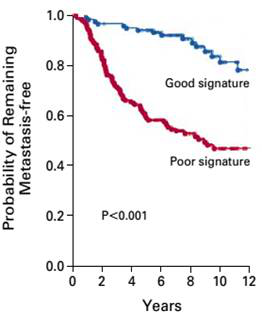
*图释:左图A:每一行代表一个原发性乳腺癌标本,每一列代表一个基因。每一个点代表单个基因在单个样本中的表达水平。虚线代表区分预后的阈值线。B:红点代表5年内发生乳腺癌转移的病例(预后差),蓝点代表5年内未出现乳腺癌转移病例的随访时间。右图:Kaplan–Meier分析,基于70个基因的表达特征区分的预后好和预后差乳腺癌患者未发生肿瘤转移的可能性。预后好的病例未发生肿瘤转移的概率明显高于预后差的病例。
2.功能机制研究案例
表达谱芯片用于糖尿病引发的前列腺病变的分子机制研究(Ye C. et al. Laboratory Investigation, 2011)
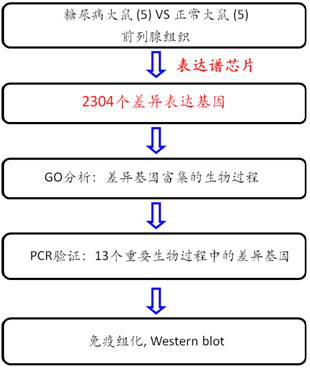
糖尿病是由于胰岛素分泌缺陷或其生物作用受损导致的以高血糖为特征的代谢性疾病。糖尿病会影响前列腺上皮细胞增殖、分化和死亡,引发前列腺炎,严重地可能会导致前列腺癌。但是,糖尿病引起前列腺病变的分子机制仍不清楚。
为了研究这个问题,作者构建了STZ(链脲霉素)诱导的大鼠糖尿病模型。使用表达谱芯片分析糖尿病大鼠和正常大鼠的前列腺组织,组间比较发现2304个基因的表达水平发生显著变化。差异基因根据功能可归类为:DNA损伤修复、细胞循环检测、细胞增殖和细胞凋亡等。选择参与上述过程的13个基因进行PCR验证,与DNA损伤修复相关的基因MRE11和XRCC3在糖尿病大鼠前列腺组织中显著下调,这与芯片结果一致。通过免疫组化实验,
技术路线
结果展示

*图释:左图:差异表达基因显著富集的生物学过程及其中的差异表达基因数目。
→ Loss of Gsα impairs liver regeneration through a defect in the crosstalk between cAMP and growth factor signaling. J Hepatol. 2016
→ FOXP3 promotes tumor growth and metastasis by activating Wnt/β-catenin signaling pathway and EMT in non-small cell lung cancer. Molecular Cancer. 2017
→ Long noncoding RNA CRNDE stabilized by hnRNPUL2 accelerates cell proliferation and migration in colorectal carcinoma via activating Ras/MAPK signaling pathways. Cell Death & Disease. 2017
→ In vitro expansion impaired the stemness of early passage mesenchymal stem cells for treatment of cartilage defects. Cell Death & Disease. 2017
→ PMP22 Regulates Self-Renewal and Chemoresistance of Gastric Cancer Cells. Molecular Cancer Therapeutics. 2017
→ Increased Variability of Genomic Transcription in Schizophrenia. Scientific Reports. 2016
→ Altered expression of mRNA profiles in blood of early-onset schizophrenia. Scientific Reports. 2016
→ ABCF2, an Nrf2 target gene, contributes to cisplatin resistance in ovarian cancer cells. Molecular Carcinogenesis. 2017
→ Bifunctional enzyme SpoT is involved in the clarithromycin tolerance of Helicobacter pylori by up-regulating the transporters HP0939, HP1017, HP0497, and HP0471. Antimicrobial Agents and Chemotherapy. 2017
→ VNN1 Promotes Atherosclerosis Progression in apoE–/– Mice Fed a High-Fat/High-Cholesterol Diet. Journal of lipid research. 2016
→ MeCP2, a target of miR-638, facilitates gastric cancer cell proliferation through activation of the MEK1/2-ERK1/2 signaling pathway by upregulating GIT1. Oncogenesis. 2017
→ Comparative analysis of gene expression profiles in normal hip human cartilage and cartilage from patients with necrosis of the femoral head. Arthritis research & therapy. 2016
→ Comparative analysis of gene expression profiles in normal hip human cartilage and cartilage from patients with necrosis of the femoral head. Arthritis Research & Therapy. 2016
→ Upregulation of miR-483-3p contributes to endothelial progenitor cells dysfunction in deep vein thrombosis patients via SRF. Journal of Translational Medicine. 2016
→ Construction and analysis of circular RNA molecular regulatory networks In liver cancer. Cell Cycle. 2017
→ Transcriptome landscape of porcine intramuscular adipocytes during differentiation. Journal of Agricultural and Food Chemistry. 2017
→ Global expression profile of tumor stem-like cells isolated from MMQ rat prolactinoma cell. Cancer Cell Internationa. 2017
→ Transcriptome reveals the overexpression of a kallikrein gene cluster (KLK1/3/7/8/12) in the Tibetans with high altitude-associated polycythemia. International Journal of Molecular Medicine. 2016
→ Prognostic Significance of MAGE-A11 in Esophageal Squamous Cell Carcinoma and Identification of Related Genes Based on DNA Microarray. Archives of Medical Research. 2016
→ MicroRNA expression profiles in chronic epilepsy rats and neuroprotection from seizures by targeting miR-344a. Neuropsychiatric Disease and Treatment. 2017
→ Identification of gene expression profiles associated with doxorubicin resistance in paired doxorubicin-resistant and doxorubicin-sensitive osteosarcoma cell lines. Int J Clin Exp Pathol. 2017
→ Role of MAPK/ERK signal pathway in recurrent miscarriage patients by case-control analysis. Int J Clin Exp Pathol. 2016

