

|
单细胞测序技术服务 靶向lncRNA单细胞全转录组测序 单细胞全转录组测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
RNA-蛋白相互作用 HyPro - MS CHIRP – MS RNA pull-down MS |
RNA-RNA/DNA相互作用 HyPro-seq/芯片 CHIRP-seq |
蛋白-RNA相互作用 AGO APP seq/芯片 RIP-RNA seq/芯片 |
蛋白-蛋白相互作用 CoIP-MS/AP-MS |
|
NGS测序技术服务 DRIPc-seq |
|
NGS测序技术服务 环状DNA测序 |
|
基因芯片技术服务 Small RNA修饰芯片 m6A单碱基分辨率芯片 mRNA&lncRNA表观转录组芯片 circRNA表观转录组芯片 |
NGS测序技术服务 表观转录组学测序服务 RNA m6A甲基化测序(MeRIP Seq) |
LC-MS mRNA碱基修饰检测 tRNA碱基修饰检测 |
PCR技术服务 MeRIP-PCR技术服务 m6A绝对定量RT-PCR技术服务 m6A单碱基位点PCR(MazF酶切法)技术服务 |
|
NGS测序技术服务 DNA(羟)甲基化测序(抗体法) DNA甲酰基胞嘧啶(5fc)修饰测序 DNA 5hmC 测序(化学法) 染色质免疫共沉淀测序 |
PCR技术服务 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR |
|
Ribo-seq Ribo seq |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC联合mRNA-seq |
南京医科大学公共卫生学院王建明教授课题组从事circRNA表观遗传调控网络、食管等癌症分子流行病学的研究。近期其实验室用Arraystar CircRNA芯片研究乳腺癌组织中circRNA表达谱,发现有1155个差异表达circRNA,715个circRNA上调,440个circRNA下调。通过qPCR验证发现 hsa_ circ_103110,hsa_circ_104689 和hsa_circ_104821在乳腺癌中上调, hsa_circ_006054,hsa_circ_100219和hsa_circ_406697在乳腺癌中下调。联合应用3个下调的circRNA分子作为Biomarker时,用于区分乳腺癌的AUC=0.82 (95% CI: 0.73-0.90)。机制研究发现,差异表达的circRNA分子可以通过吸附miRNA,参与乳腺癌发生发展。该研究成果刊登在国际期刊Oncotarget(IF:5.168)上。(芯片实验由康成生物提供技术服务)
研究背景
乳腺癌发病非常普遍并且是女性癌症致死首要疾病。据统计,2012年全世界有170万乳腺癌患者,并有521,900人死于乳腺癌。流行病学研究发现,肥胖、高龄产妇、使用雌激素和孕酮,缺乏运动和酗酒等影响增大了乳腺癌发病风险。这些因素对于治疗后的预后也有影响。在乳腺癌发病中,遗传和表观遗传因素都起作用。近几年的研究发现,lncRNA、microRNA、circRNA等在癌症发生发展中起重要作用。其中circRNA可以通过海绵作用吸附microRNA,在斑马鱼脑发育中起调控作用。在结肠、基底细胞和膀胱癌中都发现circRNA的异常表达,乳腺癌中也有相关报道,但是缺乏实验和临床证据。Nair采用Circ-seq的方法发现circRNA可以作为乳腺癌增殖的生物标志物,可以区分乳腺癌的不同亚型。Galasso采用生物信息预测工具发现circRNA在乳腺癌中的预测价值,但是检测到类群比较少。王建明老师课题组的研究采用分子流行病学,在中国人群中建立circRNA表达谱,鉴定人乳腺癌发病中异常表达的circRNA。
研究思路
为了研究circRNA在乳腺癌中的表达及作用。本次研究应用美国Arraystar Human CircRNA芯片筛选4对侵袭性导管乳腺癌中差异表达的circRNA。高通量筛选之后,研究者募集了51名乳腺癌病人进行验证。患者平均年龄46.5岁(18-79)。在4组配对样本中,设定筛选标准为差异倍数>2倍,P value<0.05,发现病变组和正常组相比,有715个circRNA显著上调,440个circRNA显著下调。考虑到多重比对可能引起假阳性,数据筛选时设定了FDR校正,16个circRNA显著上调,5个circRNA显著下调。后期采用qRT-PCR验证芯片结果。验证采用的数据筛选指标为:(1)长度200-3000bp;(2)倍数改变≥2;(3)P值<0.01;(4)原始强度>200;(5)外显子类型的circRNA;(6)保守的。其中有6个分子经过验证,并且进一步用于评估这6个分子的诊断价值。
技术路线

结果展示
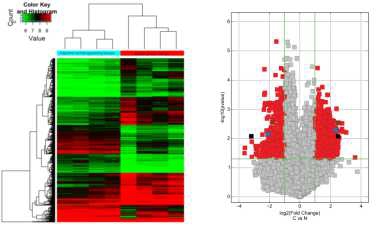
图一:CircRNA表达聚类图。颜色按照绿色(低强度) ,黑色(中强度),红色(高强度)标注。火山图展示乳腺癌和临近的正常外形的组织circRNA表达差异。垂直的绿色线条代表上调和下调2倍,水平绿色线条代表P值<0.05。
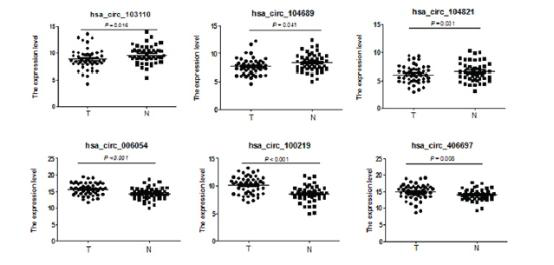
图二:用qRT-PCR验证乳腺癌和临近的正常外形的组织。散点代表乳腺癌(C)和临近正常外形组织(N),参考线上方的标记代表下调的circRNA。
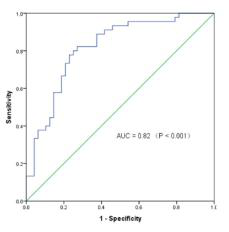
图三:区分乳腺癌的circRNA生存曲线。AUC:曲线面积;circRNA组合:has_circ_006054, has_circ_100219和has_circ_406697。
研究意义
本研究用Arraystar Human CircRNA芯片对乳腺癌中circRNA进行高通量挖掘和筛选。该研究揭示了乳腺癌和临近正常外形组织中的circRNA表达谱,发现has_circRNA_104689、 has_circ_104821和 has_circ_100219显著上调,has_circRNA_005054、has_circ_406697和has_circ_100219显著下调。这些CircRNA可通过吸附microRNA参与癌症相关信号通路,参与乳腺癌发生。
论文链接
Identification of circular RNAs as a promising new class of diagnostic biomarkers for human breast cancer.

