

|
单细胞测序技术服务 靶向lncRNA单细胞全转录组测序 单细胞全转录组测序 |
|
生物分子凝聚体研究 HyPro靶RNA临近标记技术 |
|
RNA-蛋白相互作用 HyPro - MS CHIRP – MS RNA pull-down MS |
RNA-RNA/DNA相互作用 HyPro-seq/芯片 CHIRP-seq |
蛋白-RNA相互作用 AGO APP seq/芯片 RIP-RNA seq/芯片 |
蛋白-蛋白相互作用 CoIP-MS/AP-MS |
|
NGS测序技术服务 DRIPc-seq |
|
NGS测序技术服务 环状DNA测序 |
|
基因芯片技术服务 Small RNA修饰芯片 m6A单碱基分辨率芯片 mRNA&lncRNA表观转录组芯片 circRNA表观转录组芯片 |
NGS测序技术服务 表观转录组学测序服务 RNA m6A甲基化测序(MeRIP Seq) |
LC-MS mRNA碱基修饰检测 tRNA碱基修饰检测 |
PCR技术服务 MeRIP-PCR技术服务 m6A绝对定量RT-PCR技术服务 m6A单碱基位点PCR(MazF酶切法)技术服务 |
|
NGS测序技术服务 DNA(羟)甲基化测序(抗体法) DNA甲酰基胞嘧啶(5fc)修饰测序 DNA 5hmC 测序(化学法) 染色质免疫共沉淀测序 |
PCR技术服务 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR |
|
Ribo-seq Ribo seq |
核糖体-新生肽链复合物(RNC) RNC联合 circRNA芯片 RNC联合 lncRNA芯片 RNC联合mRNA-seq |
技术优势:
● 专注非编码RNA领域,完善的lncRNA启动子分析流程
● 可与lncRNA表达谱芯片联合应用,实现平台间无缝对接
● 可视化数据展示,提供paper级结果图表
● 优化的IP实验方法,可信赖的检测平台及数据结果
介绍:
人类基因组中仅有约2%的DNA序列最终编码生成蛋白质,其余绝大部分区域转录形成长链非编码RNA。随着lncRNA的生物学功能的发现,这些原先被认为是“junk DNA”的区域的研究地位上升为“暗物质”。lncRNA的转录受到严格的调控,其表达谱细胞特异性和组织特异性甚至高于蛋白编码基因。寻找和发现疾病过程中LncRNA表达变化的原因,了解lncRNA上游调控机制,是lncRNA研究领域重要组成部分,而表观遗传学正是从基因组层面研究RNA转录调控的重要领域。
启动子区域的DNA的甲基化对RNA的转录起抑制作用。研究发现,在肝癌以及精神分裂患者样品中转录形成LncRNA MEG3的基因组区域发生DNA甲基化修饰程度的异常改变;食管癌具有特征性lncRNA启动子DNA甲基图谱,其区分癌与癌旁组织的效果要优于mRNA启动子;乳腺癌中高达57.18%的lncRNA发生启动子DNA甲基化水平的改变,lncRNA启动子的甲基化程度和lncRNA的表达水平显著相关。
康成生物在MeDIP-seq数据中加入lncRNA启动子DNA甲基化分析,客户可以同时得到lncRNA,mRNA的DNA甲基化相关数据,可与Arraystar lncRNA芯片联合应用,实现两个平台的无缝对接。lncRNA启动子分析同样适用于hMeDIP-seq平台,可以快速便捷地确定DNA羟甲基化修饰在lncRNA启动子区域的分布。

图1. 甲基化/羟甲基化测序lncRNA启动子分析流程
重要数据结果展示:
1. 两组/个样品的lncRNA启动子差异(羟)甲基化分析
康成生物使用软件分析两组/个样品间的差异(羟)甲基化区域。与传统的先合并reads,再计算差异富集区,或者先分别计算差异富集区,再合并的方法相比,这种方法更加稳健,能够更好地利用重复数据内的变异,获得更好的统计结果。根据基因组坐标,利用UCSC RefSeq数据库对位于lncRNA启动子区(TSS - 2000 bp到TSS + 2000 bp)的差异(羟)甲基化区域进行注释。
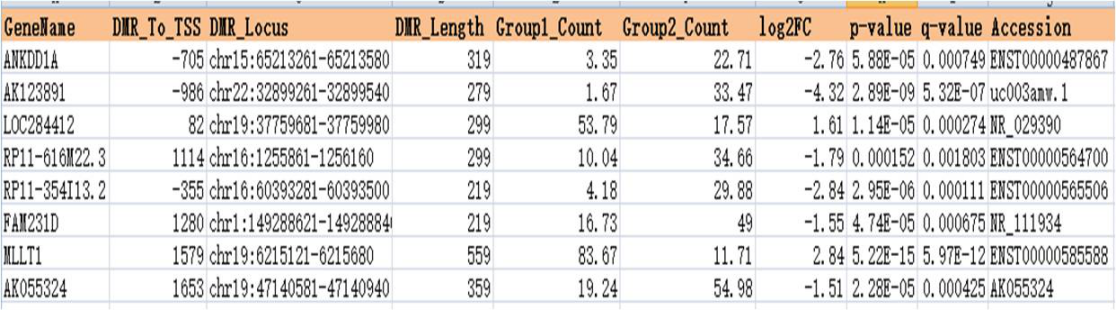
表1:lncRNA启动子差异甲基化区域列表
2. 与lncRNA表达谱芯片的联合分析
甲基化测序/羟甲基化测序数据和lncRNA表达谱数据结合分析,可绘制差异(羟)甲基化情况聚类热图。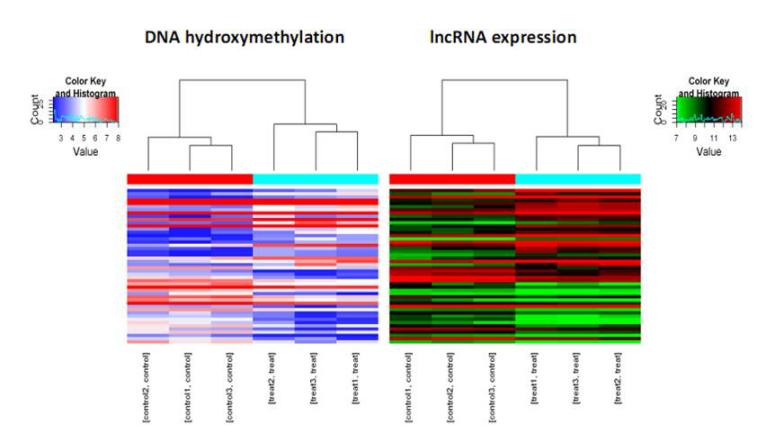
图2. DNA羟甲基化测序与LncRNA 表达谱联合分析聚类图
3. lncRNA启动子甲基化/羟甲基化可视化分析
甲基化测序/羟甲基化测序结果中的wig文件能够上传到UCSC基因组浏览器中,以进行lncRNA启动子区域信号的可视化。
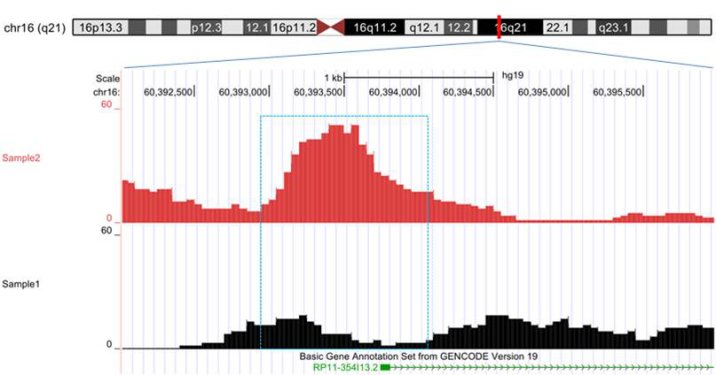
图3. 可视化分析
参考文献
1. Anamaria Necsulea. et al.(2014) Nature 505(7485):635-40 [PMID:24463510]
2. John P. Thomson. et al.(2013) Nucleic Acids Res 41(11):5639-54[PMID:23598998]
3. Wenjing Wu. et al.(2013) Gastroenterology 144(5):956-966.e4 [PMID:23333711]
4. Yongsheng Li. et al.(2015) Scientific Reports 5;5:8790 [PMID:25739977]

